Generierung und Charakterisierung patientenspezifischer hiPSCs und hiPSC-CMs
hiPSC-Linien wurden von zwei TOF-DG-Patienten, zwei TOF-ND-Patienten und zwei gesunden Kontrollpersonen mit verifizierten Pluripotenzmarkern und Keimschichtmarkern etabliert (ergänzende Abbildungen). 1 machen 2). Die Sequenzierung des gesamten Genoms bestätigte das Vorhandensein bzw. Fehlen einer 22q11.2-Deletion in den hiPSC-Linien von TOF-DG- und TOF-ND-Patienten (ergänzende Abbildung). 3). In keiner der hiPSC-Linien wurden Missense- oder Nonsense-Mutationen zuvor gemeldeter KHK-bezogener Gene gefunden. Die Herzdifferenzierung wurde durch das APLNR erreicht+ Sortierprotokoll, das von unserem Labor entwickelt wurde32. Alle hiPSC-Linien zeigten eine zufriedenstellende Differenzierungseffizienz mit über 70 % TNNT2+-Zellen. Am 12. Tag nach der Differenzierung wurden hiPSC-CMs in hCAS umgewandelt und vor der transkriptomischen und funktionellen Beurteilung weitere 10 Tage reifen gelassen.
Patientenspezifische TOF-DG-hiPSC-CMs weisen eine fehlerhafte ventrikuläre Spezifikation auf
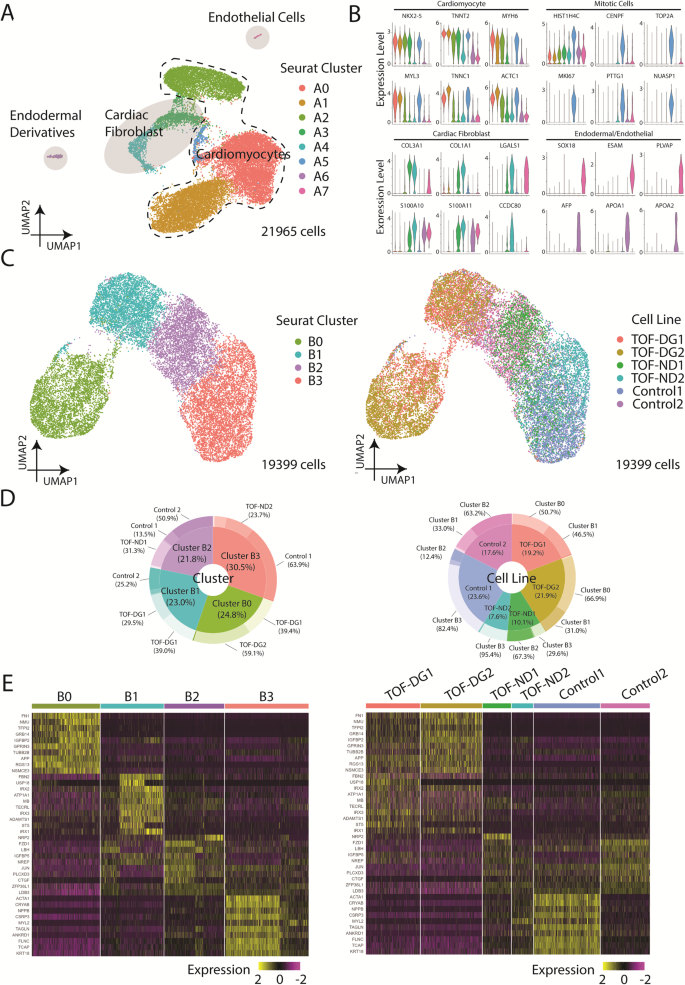
Wir haben zunächst das transkriptomische Profil differenzierter hiPSC-CMs von Patienten und Kontrollen durch scRNA-seq mithilfe der 10X Genomics-Plattform bestimmt. Die Clusterung von 21,965 Zellen mittels Uniform Manifold Approximation and Projection (UMAP) identifizierte 7 verschiedene Seurat-Cluster (Abb. 1A). Die Seurat-Cluster A0, A1, A2, A5 exprimierten relativ höhere Mengen an Herzgenen, einschließlich NKX2-5, Myosin-Leicht-/Schwerkette und Troponin (Abb. 1B), der diese Cluster als Kardiomyozyten identifizierte. Cluster A5 exprimierte auch höhere Mengen an mitotischen Genen, was auf die proliferative Natur der Kardiomyozyten schließen lässt. Die Cluster A3 und A4 exprimierten relativ geringere Mengen an Herzgenen (NKX2-5, TNNT2), aber angereichert in der Expression von Genen, die mit der Umgestaltung der extrazellulären Matrix (ECM) zusammenhängen (Abb. 1B), dessen Profil mit Herzfibroblasten kompatibel ist. Die Cluster A6 und A7 wurden durch die endodermale Abstammungslinie definiert (AFP, APOA1, APOA2) und Endothel (FLT1, ESAM, PLVAP) Markierungen bzw. (Abb. 1B). Daher waren unter den sequenzierten Zellen die meisten Kardiomyozyten (86 %), einige waren Herzfibroblasten (10 %), und nur sehr wenige waren endodermale (<1 %) und endotheliale Derivate (<1 %).
A UMAP-Präsentation aller in hCAS identifizierten Zelltypen (TOF-DG, TOF-ND und Kontrolle). Färbung: Seurat-Cluster; gestrichelte Linie: Kardiomyozyten; hellgrau: Nicht-Myozyten. B Violindiagramme der Genexpression von Kardiomyozyten, mitotischen Zellen und Nicht-Myozyten: Herzfibroblasten, endodermale Derivate und Endothelzellen. Färbung: Seurat-Zellcluster wie in (A). C UMAP-Präsentationen aller in hCAS identifizierten hiPSC-CMs (TOF-DG, TOF-ND und Kontrolle). Die Daten wurden nach Seurat-Clustern (linkes Feld) bzw. Zelllinie (rechtes Feld) gefärbt. D Donut-Diagramme der Zusammensetzungen in jedem Seurat-Cluster (linkes Feld) bzw. Zelllinie (rechtes Feld). Die Daten wurden nur aus hiPSC-CMs berechnet. E Heatmap-Präsentationen der zehn am häufigsten hochregulierten Gene, die in jedem hiPSC-CMs-Seurat-Cluster exprimiert werden. Die Daten wurden nach Seurat-Cluster (linkes Feld) bzw. Zelllinie (rechtes Feld) gruppiert.
Mit Schwerpunkt auf der Untersuchung des Herztranskriptoms isolierten wir hiPSC-CMs (Cluster A0, A1, A2, A5) und führten Clustering mithilfe von UMAP durch (Abb. 1C). Neue Seurat-Cluster (B0, B1, B2, B3) wurden identifiziert (Abb. 1C). Von Patienten und Kontrollen stammende hiPSC-CMs waren ungleichmäßig auf die Cluster verteilt (Abb. 1D). Cluster B0 bestand fast ausschließlich (98 %) aus hiPSC-CMs, die von TOF-DG-Patienten stammten, Cluster B1 bestand hauptsächlich aus Zellen einer Kontrollperson und die restlichen Zellen von TOF-DG-Patienten, während die Cluster B2 und B3 aus hiPSC-CMs bestanden abgeleitet von TOF-ND-Patienten und Kontrollen.
Es wurde festgestellt, dass Cluster B0 eine hochregulierte Expression von Nicht-Myozyten-Genen aufweist, wobei 4 von 10 der am stärksten hochregulierten Gene selten (<1 %) in anderen Clustern gefunden wurden (Abb. 1E). Diese Nicht-Myozyten-Gene waren nicht endodermal oder endothelial verwandt. Das Fehlen einer Anreicherung von Genen im Zusammenhang mit der Umgestaltung der extrazellulären Matrix unterschied auch Cluster-B0-Zellen von Herzfibroblasten. Cluster B1 wurde im angereichert IRX Genfamilie (IRX1, IRX2, IRX3), während Cluster B2 durch markiert war LBH Ausdruck (Abb. 1E). Die Orthologen von IRX Gene und LBH Es wurde berichtet, dass sie in frühen in vivo differenzierten murinen Kardiomyozyten exprimiert werden33,34. Cluster B2 wies im Vergleich zu Cluster B1 eine hochregulierte Expression ventrikulärer Kardiomyozytenmarker auf (MYH7, NPPB). Cluster B3 zeigte die höchste Expression des ventrikulären Kardiomyozytenmarkers (MYL2) (Feige. 1E). Das Vorhandensein der Cluster B1, B2 und B3 deutete auf eine fortschreitende Spezifizierung von hiPSC-CMs hin zu einem ventrikulären Transkriptomprofil hin.
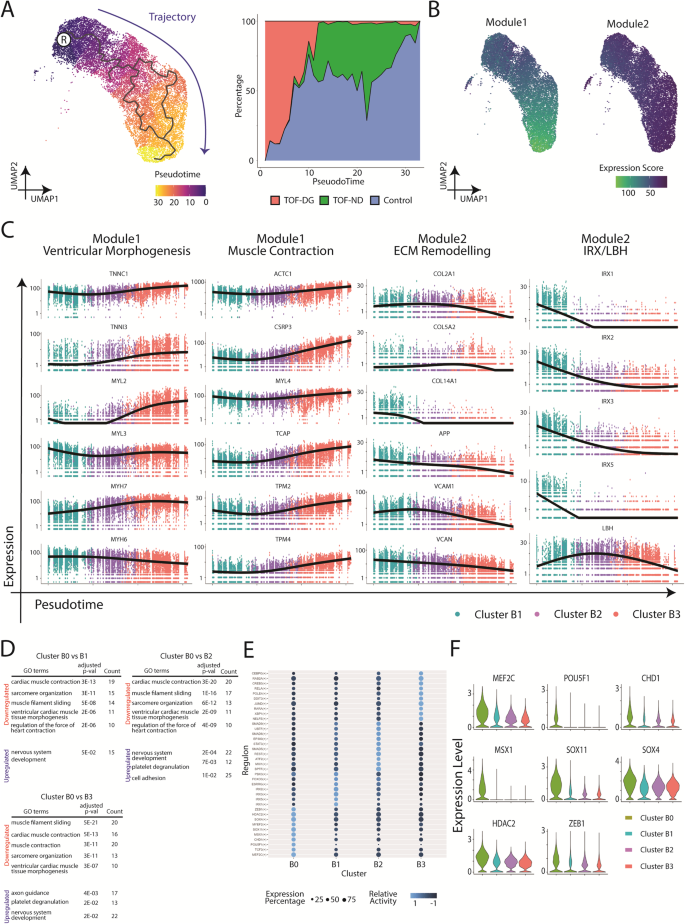
Um die transkriptomischen Veränderungen während der ventrikulären Spezifikation weiter zu untersuchen, führten wir eine Pseudozeitanalyse für die Cluster B1, B2 und B3 durch. Basierend auf der Bereicherung im IRX Gene-Familie, Cluster B1 wurde manuell als Wurzelknoten für die Pseudozeitanalyse ausgewählt (Abb. 2A). Entlang der Flugbahn waren TOF-DG-hiPSC-CMs hauptsächlich am Anfang verteilt, wohingegen TOF-ND-hiPSC-CMs hauptsächlich in der Mitte bis zum Ende verteilt waren. Kontroll-hiPSC-CMs konnten entlang der gesamten Flugbahn gefunden werden (Abb. 2A).
A Pseudozeitanalyse von hCAS-hiPSC-CMs. UMAP-Koordination und Genexpression der Cluster B1, B2 und B3 (Abb. 1C) wurden für die Monocle3-Berechnung extrapoliert. Der Wurzelknoten wurde mit ® gekennzeichnet. Die Flugbahn war blau eingefärbt. B Genmodule dienen als Funktion der Pseudozeitanalyse und Trajektorieninferenz von hCAS-hiPSC-CMs. C Genexpressionsdynamik entlang der Trajektorie. Ventrikuläre Morphogenese, Muskelkontraktion und ECM-Remodellierung waren die in den Genmodulen angereicherten GO-Begriffe. X-Achse: Pseudozeit ; Y-Achse: Ausdrucksebene. Extrazelluläre ECM-Matrix. D GO-Anreicherungsanalyse von Cluster B0 aus hCAS-hiPSC-CMs. Es wurde gezeigt, dass GO-Terme in den herunterregulierten/hochregulierten Genen von Cluster B0 (im Vergleich zu Cluster B1/B2/B3) angereichert sind. Die P-Werte wurden für Bonferroni-Mehrfachvergleiche angepasst. E Regulon-Analyse von hCAS-hiPSC-CMs. Die Top-Regulons (höchstens 10) mit höherer relativer Aktivität in jedem Seurat-Cluster wurden im Punkt-Heatmap-Diagramm angezeigt. F Genexpression der Top-Regulons (identifiziert als DEGs) aus Cluster B0. Genexpressionen wurden im Geigendiagramm dargestellt.
Gene mit ähnlichen Expressionsmustern wurden in Module gruppiert (Abb. 2B) und einer Gene Ontology (GO)-Anreicherungsanalyse unterzogen. Signifikante GO-Begriffe wurden sowohl in Modul 1 als auch in Modul 2 gefunden (Abb. 2C). Modul 1 zeigte hohe Expressionswerte entlang der Trajektorie und war mit Genen angereichert, die mit der ventrikulären Morphogenese zusammenhängen (TNNI3, MYL2, MYH7) und Muskelkontraktion (ACTC1, CSRP3, TCAP). Modul 2, das abnehmende Expressionswerte entlang der Flugbahn zeigt, wurde mit Genen angereichert, die mit der Umgestaltung der extrazellulären Matrix zusammenhängen (COL2A1, COL5A2, COL14A1). Die IRX Gene Familie und LBH wurden ebenfalls Modul 2 zugeordnet und zeigten abnehmende Ausdruckswerte entlang der Flugbahn.
Zusammenfassend lässt sich sagen, dass die Pseudozeitanalyse von hiPSC-CMs, die von der hCAS-Plattform abgeleitet wurden, eine progressive ventrikuläre Spezifikation mit erhöhter kardialer Genexpression identifizierte. Kontroll- und TOF-ND-hiPSC-CMs waren in solchen progressiven Spezifikationen vergleichbar. Die meisten TOF-DG-hiPSC-CMs behielten jedoch das primitivere Expressionsprofil bei.
Eine Untergruppe patientenspezifischer TOF-DG-hiPSC-CMs zeigte eine Herunterregulierung der kardialen Genexpression, aber eine Hochregulierung der neuronalen Genexpression
Um die Nicht-Myozyten-Genexpression in Cluster B0 weiter zu analysieren, wurden differentiell exprimierte Gene (DEGs) identifiziert, indem Cluster B0 mit jedem der anderen Cluster verglichen wurde. Mehr als 200 DEGs, hochreguliert oder herunterreguliert, wurden aus Cluster B0 (Supplementary Data) identifiziert 1). Die GO-Anreicherungsanalyse identifizierte konsistent signifikante GO-Begriffe im Zusammenhang mit der Herzmuskelkontraktion bei herunterregulierten Genen und solche im Zusammenhang mit der neuronalen Entwicklung bei hochregulierten Genen im Cluster B0 (Abb. 2D). Daher unterschied sich Cluster B0 vom Rest der Cluster durch eine herunterregulierte kardiale Genexpression sowie eine hochregulierte neuronale Genexpression. Dennoch konnte keines der oben genannten DEGs, einschließlich herunterregulierter Herzgene und hochregulierter neuronaler Gene, in beiden TOF-DG-hiPSC-Linien der haploinsuffizienten Region zugeordnet werden.
Wir haben außerdem versucht, die entsprechenden Transkriptionsfaktoren (TFs) und genregulatorischen Netzwerke aus jedem der Cluster durch Python-Implementierung des Single-Cell rEgulatory Network Inference and Clustering (pySCENIC) zu identifizieren. Unter den Top 10 TFs mit höherer Aktivität im Cluster B0 waren 4 an der neuronalen Entwicklung beteiligt (TCF3, SOX11, SOX4 machen ZEB1) und 2 in der Herz- und Nervenentwicklung (MEF2C machen MYEF2) (Feige. 2E). Die Expressionsniveaus von fünf neuronalen TFs (SOX11, SOX4, ZEB1, MEF2C machen MYEF2) waren in Cluster B0 im Vergleich zu denen in anderen Clustern hochreguliert (Abb. 2F). Der Befund einer hochregulierten Expression dieser neuronalen TFs stimmte mit einer erhöhten Expression neuronaler Gene im Cluster B0 überein. Ähnlich wie die DEGs wurden die oben genannten neuronalen TFs in beiden TOF-DG-Zelllinien nicht der haploinsuffizienten Region zugeordnet.
Daher zeigte ein erheblicher Teil der hiPSC-CMs aus TOF-DG (Cluster B0) eine Herunterregulierung der Herzgene und eine Hochregulierung der ektopischen neuralen Gene. Das Versäumnis, die DEGs und TFs im Cluster B0 der Region 22q11.2 zuzuordnen, deutete darauf hin, dass diese transkriptomische Signatur wahrscheinlich eine indirekte Folge der Haploinsuffizienz ist. Im Gegensatz zu den hiPSC-CMs aus der Kontroll- und der TOF-ND-Gruppe, die ein Kontinuum in der ventrikulären Spezifikation zeigten, bildete Cluster B0 einen anderen Cluster als die übrigen hiPSC-CMs aus TOF-DG (Cluster B1). Der Ursprung des Clusters B0 wurde wie unten beschrieben weiter untersucht.
Gegabelte Herzdifferenzierung von TOF-DG-Herzvorläufern
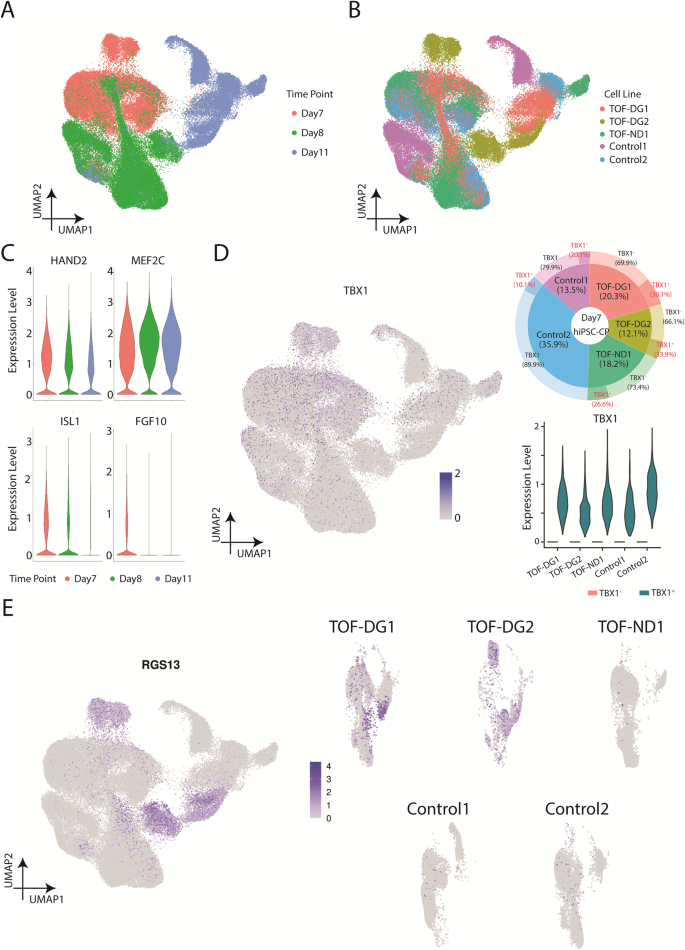
Um die Entstehung des TOF-DG-Clusters B0 zu verstehen, untersuchten wir das transkriptomische Profil von In-vitro-Herzvorläufern. Basierend auf unserer vorherigen Studie32Die Genexpression des zweiten Herzfeldes (SHF) erreichte am 7. und 8. Tag (D) nach der Differenzierung ihren Höhepunkt. Wir führten daher eine scRNA-Seq an hiPSC-Herzvorläufern (hiPSC-CPs) an D7 und D8 und an unreifen hiPSC-CMs an D11 durch 2 Kontrollpersonen (Kontrolle 1 und 2) und 2 TOF-DG-Patienten (TOF-DG1 und 2) und 1 TOF-ND-Patienten (TOF-ND1) (Abb. 3A,B). Insgesamt wurden 87834 Zellen zurückgehalten, nachdem weniger als 1 % der endodermalen Derivate und Endothelzellen ausgeschlossen wurden. Pseudotime- und GO-Anreicherungsanalysen identifizierten eine Hochregulierung der kardialen Genexpression und eine Herunterregulierung der Transkription und Translation sowie der Zellzyklusaktivität entlang der Flugbahn (ergänzende Abbildung). 4A–D). Ähnlich wie in unserer vorherigen Studie32, SHF-Genexpression, einschließlich ISL1, MEF2C, HAND2 und FGF10, wurden in den sequenzierten Zeitpunkten gefunden (Abb. 3C). TBX1, eines der Gene innerhalb der Mikrodeletionsregion 22q11.2, ist ebenfalls ein Marker für SHF. Unter den sequenzierten Zeitpunkten TBX1 Der Ausdruck wurde am 7. Tag gefunden, mit einem variablen Anteil von TBX1+ hiPSC-CPs (10–30 %), gefunden aus verschiedenen Linien (Abb. 3D). Die TBX1 Das Expressionsniveau war vergleichbar zwischen dem D7-TBX1+ hiPSC-CPs aus verschiedenen Linien (Abb. 3D).
A UMAP-Präsentationen aller hiPSC-CPs und hiPSC-CMs (TOF-DG, TOF-ND und Kontrolle). Die Daten wurden nach sequenziertem Zeitpunkt eingefärbt. B UMAP-Präsentationen aller hiPSC-CPs und hiPSC-CMs (TOF-DG, TOF-ND und Kontrolle). Die Daten wurden nach Zelllinie gefärbt. C Violindiagramme der SHF-Genexpression, einschließlich ISL1, MEF2C, HAND2 machen FGF10. D TBX1 Expression in hiPSC-CPs. TBX1 Die Expression der In-vitro-Herzdifferenzierung am 7./8./11. Tag wurde im UMAP-Diagramm gezeigt. Höchste TBX1 Die Expression wurde in D7-hiPSC-CPs gefunden, und der Anteil der TBX1+ wurde im Donut-Plot angezeigt und mit einem roten Etikett markiert. TBX1 Ausdruck in TBX1+ D7-hiPSC-CPs wurden im Violinplot dargestellt. E UMAP-Diagramme von RGS13 Expression am 7./8./11. Tag in vitro Herzdifferenzierung. RGS13 Expression wurden mit allen Zelllinien zusammen dargestellt (linkes Feld) und in jede einzelne Zelllinie aufgeteilt (rechtes Feld).
Ein zeitlich abgestimmter Vergleich mit Kontrollen identifizierte nur 20, 10 und 22 DEGs aus D7-, D8- und D11-TOF-DG-hiPSC-CP/CMs, ohne dass signifikante GO-Terme gefunden wurden. Keines dieser DEGs wurde den 22q11.2-Mikrodeletionsregionen in den beiden TOF-DG-hiPSC-Linien zugeordnet. Anschließend wurden ektopische Genexpressionen aus dem TOF-DG-Cluster B0 in den hiPSC-CPs und unreifen hiPSC-CMs untersucht. Im Gegensatz zum TOF-DG-Cluster B0 (differenzierte hiPSC-CMs) wurden in TOF-DG-hiPSC-CPs (D7 und D8) und D11-unreifen hiPSC-CMs keine neuronalen Gene gefunden. Andererseits eine Teilmenge von Zellen, die mit gekennzeichnet sind RGS13 Expression, eines der zehn am häufigsten hochregulierten Gene im TOF-DG-Cluster B1 (Abb. 1E) wurde in allen sequenzierten Zeitpunkten (D7, D8 und D11) konsistent in TOF-DG-abgeleiteten Zellen gefunden (Abb. 3E), jedoch nicht in TOF-ND1 und den Kontrollgruppen.
Kürzlich wurde über eine ektopische Genexpression bei den Vorläufern von berichtet Tbx1 bedingte Null (Tbx1-cKO) Maus35. Um unsere hiPSC-CP- und unreifen hiPSC-CM-Daten mit den zu vergleichen Tbx1-cKO-Maus haben wir einen artübergreifenden Vergleich mit dem R-Paket SingleCellNet durchgeführt36. Vierzehn Cluster wurden aus dem identifiziert Tbx1-cKO-Mausdatensatz (ergänzende Abb. 5A) mit in der Veröffentlichung angegebener Genexpression35 (Ergänzende Abb. 5B), darunter vier verschiedene kardiale Vorläufer (Multilineage Progenitor, MLP; anteriorer SHF, aSHF; posteriorer SHF, pSHF und Proepikard; PEO) und Kardiomyozyten. Unsere D7-hiPSC-CPs ähnelten größtenteils aSHF, wohingegen D11-hiPSC-unreife Kardiomyozyten größtenteils Kardiomyozyten ähnelten (ergänzende Abbildung). 5C). In der Tbx1-cKO-Mausdatensatz, Pax8, das wichtigste ektopische Gen, über das Nomaru et al.35wurde ausschließlich in MLP und Lungenvorläufern gefunden, jedoch nicht in aSHF und Kardiomyozyten (ergänzende Abb. 5D). Wir haben uns nicht identifiziert PAX8 Ausdruck in unserem D7-D11-Datensatz. Auch, RGS13, das in unserem D7-D11-Datensatz gefundene ektopische Gen, wurde im nicht identifiziert Tbx1-cKO Maus. Mit anderen Worten: In beiden Fällen wurde eine ektopische Genexpression gefunden Tbx1-cKO-Maus und unseren DG-hiPSC-CPs/CMs unterscheiden sich die Identität und die Population, die das ektopische Gen exprimiert.
Veränderte elektrophysiologische Parameter und erhöhte Arrhythmogenität bei TOF-DG-hiPSC-CMs
Zusätzlich zur transkriptomischen Profilierung haben wir die Elektrophysiologie und Arrhythmogenität der differenzierten hiPSC-CMs mithilfe unserer hCAS-Plattform untersucht, um eine umfassendere Bewertung der hiPSC-CMs zu ermöglichen. Elektrophysiologische Parameter, einschließlich Aktionspotential (AP), Kalziumhandhabung und effektive Refraktärzeit (ERP), wurden unter elektrischer Stimulation bei 1 Hz gemessen.
Im Vergleich zu beiden Kontrollen zeigte TOF-DG2-hCAS eine signifikante Verkürzung der AP-Dauer (50 und 90 % bis zur Repolarisation, APD50 und APD90) (Abb. 4A). In ähnlicher Weise zeigte TOF-DG2-hCAS im Vergleich zu beiden Kontrollen eine signifikante Verkürzung der AP-Aufwärtshubzeit und der Zeit bis zum Abfall (50 und 90 % der Spitzenwerte) (Abb. 4A). Daher unterschieden sich die AP-Eigenschaften zwischen TOF-DG2 und den Kontrollen erheblich.
A Punktdiagramme der Charakteristik des Aktionspotentials (AP). Dauer des APD-Aktionspotentials. B Punktdiagramme der Calcium-Transienten-Charakteristik (CaT). C Dotplot der effektiven Refraktärzeit. Die Daten wurden im Mittelwert ± SD für Abb. dargestellt. 1A – C. Statistischer Test: Gewöhnliche einfaktorielle ANOVA, gefolgt von Tukeys Mehrfachvergleichstest (APD50, AP90 % Zerfall, CaT 50 % Zerfall, CaT-Aufwärtshub und effektive Refraktärzeit); Kruskal-Wallis-Test, gefolgt von Dunns Mehrfachvergleichstest (APD90, AP-Aufwärtshub, AP50 %-Zerfall und CaT 90 %-Zerfall). Signifikante Befunde zwischen TOF(DG/ND) und Kontrolle wurden mit Sternchen markiert. * P <0.05; ** **. P <0.01; ***. P < 0.001; **** P <0.0001. D Prozentsatz des arrhythmischen Wiedereintrittsereignisses von TOF (DG/ND) und Kontroll-hCAS während der Steady-State-Stimulation (SteadyState) und der programmierten elektrischen Stimulation (PES). Statistischer Test: Exakter Test nach Fisher. *** P <0.001. E Repräsentative Aktionspotentialverfolgung (AP) und Isochronenkarte des Wiedereintrittsereignisses während PES von TOF-DG2. Das obere Feld zeigte die repräsentative AP-Verfolgung während der PES (S1S2), gefolgt von der Häufigkeit arrhythmischer Wiedereintrittsereignisse. Das untere Feld zeigte zwei Isochronenkarten, die einem normalen AP (links) und einem arrhythmischen AP (rechts) entsprechen. F Herunterregulierung des Herzgens im Cluster B0-TOF-DG2. Für den Vergleich zwischen Cluster B0-TOF-DG2 und Cluster B0-TOF-DG1 wurde eine GO-Anreicherungsanalyse durchgeführt. GO-Begriffe, die mit den herunterregulierten Genen angereichert sind, sind in der Tabelle (links) und die entsprechenden Gene im Violindiagramm (rechts) aufgeführt.
Der Umgang mit Kalzium wurde anhand der Aufwärtshubzeit des Kalziumtransienten (CaT) und der Zeit bis zum Abfall (50 und 90 % vom Peak) beurteilt. Bei TOF-DG2-hCAS wurde eine signifikante Verkürzung der CaT-Aufwärtshubzeit festgestellt, wohingegen sowohl bei TOF-DG50-hCAS als auch bei TOF-ND2-hCAS im Vergleich zu beiden Kontrollen eine signifikante Verkürzung der Zeit bis zum Abfall (1 % vom Peak) festgestellt wurde ( Feige. 4B).
Wir haben außerdem die ERP der hiPSC-CMs untersucht, deren Verkürzung die Entstehung von Herzrhythmusstörungen begünstigt. Bei TOF-DG2-hCAS wurde eine signifikante Verkürzung des EPR festgestellt, während bei TOF-ND2-hCAS eine Verlängerung des ERP festgestellt wurde (Abb. 4C). Wichtig ist, dass TOF-DG2-hCAS eine signifikant höhere Inzidenz von Reentry-Arrhythmien während der programmierten elektrischen Stimulation (PES) zeigte (Abb. 4D). Eine repräsentative AP-Verfolgung mit der Isochronenkarte der Wiedereintrittsarrhythmie von TOF-DG2-hCAS ist in Abb. dargestellt. 4E.
Während TOF-DG2-hCAS eine signifikante Verkürzung der AP-Dauer, der CaT-Aufwärtshubzeit und der Abklingzeit sowie der ERP zeigte, war TOF-DG1-hCAS hinsichtlich dieser bewerteten elektrophysiologischen Parameter den Kontrollen ähnlich (Abb. 4A–D). Wir haben daher die transkriptomischen Unterschiede zwischen TOF-DG1 und TOF-DG2 im Cluster B0 weiter untersucht. Im Vergleich zum Cluster B0-TOF-DG1 zeigte Cluster B0-TOF-DG2 eine Herunterregulierung von Herzgenen im Zusammenhang mit der Muskelkontraktion (Abb. 4F). Während diese Herzgene sowohl im Cluster B0-TOF-DG1 als auch im Cluster B0-TOF-DG2 im Vergleich zu anderen Clustern (B1, B2 und B3) herunterreguliert waren (Abb. 4F), war ihr Ausmaß der Herunterregulierung im Cluster B0-TOF-DG2 stärker ausgeprägt (Abb. 4F). Außerdem, PKP2Es wird berichtet, dass seine Mutation mit arrhythmogener RV-Dysplasie assoziiert ist37Es wurde festgestellt, dass es zu den herunterregulierten Herzgenen im Cluster B0-TOF-DG2 gehört (Abb. 4F). Das unterschiedliche Ausmaß der Herunterregulierung von Herzgenen könnte daher für die funktionelle Diskrepanz verantwortlich sein, die zwischen Zelllinien verschiedener Patienten in der hCAS-Plattform beobachtet wird.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- Quelle: https://www.nature.com/articles/s42003-023-05344-6