Sehr geehrter Herausgeber,
Die einzigartigen biologischen Eigenschaften von Fledermäusen, darunter Langlebigkeit, antivirale Eigenschaften, Winterschlaf und Echoortung, haben in der wissenschaftlichen Gemeinschaft große Aufmerksamkeit erregt. Um die Nutzung von Fledermäusen in biologischen Experimenten zu verbessern, haben Forscher Anstrengungen unternommen, um induzierte pluripotente Stammzellen (iPSCs) aus Fledermausfibroblasten zu erzeugen1,2. Es bleibt jedoch ungewiss, ob transgenfreie iPSCs in vivo differenzieren können, was auf ein hohes Maß an Stammzellqualität hinweist und ihr Potenzial für verschiedene Anwendungen erheblich erweitert.
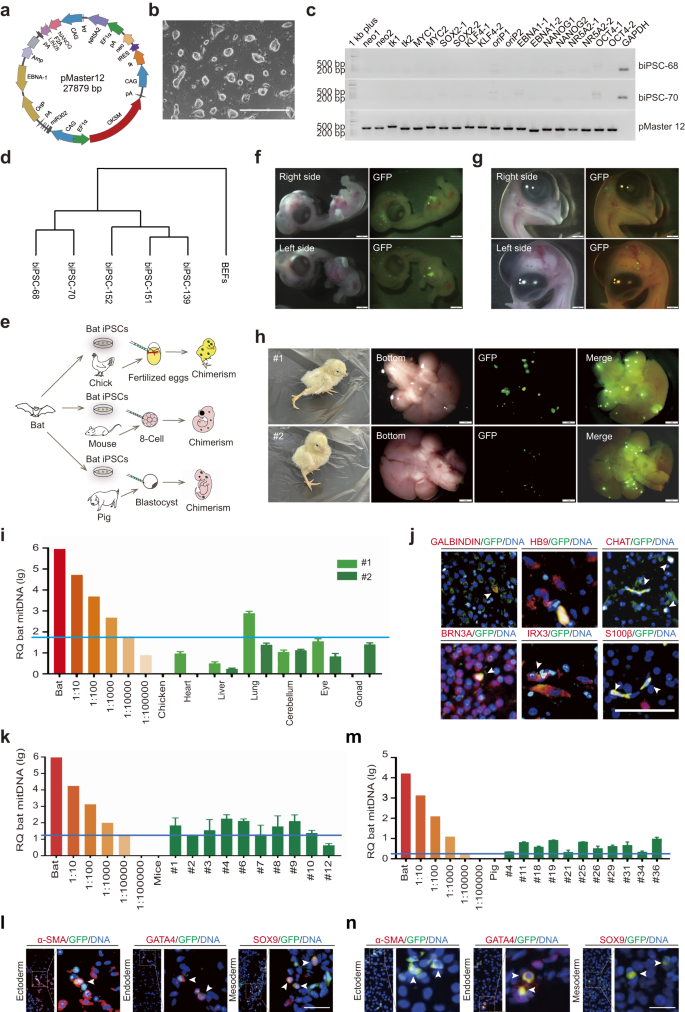
Um transgenfreie iPSCs zu erzeugen, werden embryonale Fibroblasten (BEFs) von Fledermäusen verwendet Myotis Lucifugus wurden mit einem Satz von 8 Reprogrammierungsfaktoren transfiziert (OCT4, SOX2, cMYC, KLF4, NANOG, LIN28, NR5A2Und eine miR302/367 Cluster) im 3i/LIF-Medium, das niedermolekulare Inhibitoren (PD0325901, CHIR99021 und A8301) enthält (Abb. 1a, b). Nach positiver (Neomycin) und negativer (Thymidinkinase) Selektion von iPSCs wurden monoklonale Zellen für den Nachweis exogener Faktoren ausgewählt. Wir verwendeten 20 Sätze Primerpaare, um die Sequenz des Plasmids zu amplifizieren3. Die Ergebnisse zeigten, dass zwei transgenfreie iPSC-Klone erhalten wurden (Abb. 1c).
a Schematische Darstellung des induzierbaren Vektors pMaster12, der 8 Reprogrammierungsfaktoren mit positiven/negativen Selektionsgenen kodiert. b Bild von biPSCs; Maßstabsbalken, 1000 µm. c Die PCR-Analyse bestätigte das Fehlen von pMaster12-Vektorsequenzen in biPSC-68 und biPSC-70. GAPDH wurde als internes Referenzgen verwendet und gereinigte pMaster12-DNA diente als Positivkontrolle. d Dendrogramm-Clustering von Genexpressionsprofilen: BEFs, Transgene und transgenfreie biPSCs. e Anschauliches Modell: Transgenfreie biPSCs erzeugen in vivo Interspezies-Chimären (Maus, Schwein, Küken). f Repräsentative Bilder von chimären D6.5-Fledermaus-/Hühnerembryonen mit GFP-Fluoreszenz im Kopf/Brust. Maßstabsbalken, 1 mm. g Repräsentative Bilder von chimären D9.5-Fledermaus-/Hühnerembryonen, die GFP-Fluoreszenz im Kopf zeigen. Maßstabsbalken, 1 mm. h Repräsentative Bilder von Interspezies-Lebendgeburten-Chimären aus Fledermäusen und Küken 3 Tage nach der Geburt, die GFP-Fluoreszenz im Kopf zeigen. Maßstabsbalken, 1 mm. i Quantitative genomische PCR-Analyse von Fledermaus-mtDNA in chimären neugeborenen Küken (Nr. 1 und Nr. 2). Für die Analyse wurden Fledermaus-Hühner-Zellverdünnungen verwendet. Die blaue Linie stellt den Fledermaus-mtDNA-Nachweislevel dar (1 Fledermauszelle pro 10,000 Kükenzellen). j Repräsentative IF-Färbungsbilder des Kopfes der Fledermaus-/Küken-Chimäre Nr. 2, die eine positive Färbung für neuronale Marker zeigen (GALBINDIN, HB9, CHAT, BRN3A, IRX3, S100β). Weiße Pfeile zeigen Zellen an, die sowohl für GFP als auch für neuronale Marker positiv sind. Maßstabsbalken, 50 μm. k Quantitative genomische PCR-Analyse von Fledermaus-mtDNA in Geweben von E8.5-chimären Mausembryonen unter Verwendung von Fledermaus-Maus-Zellverdünnungen. Die blaue Linie stellt das Fledermaus-mtDNA-Erkennungsniveau dar (1 Fledermauszelle pro 10,000 Mauszellen). l Repräsentative IF-Färbungsbilder, die die Integration von GFP-exprimierenden biPSCs in Keimschichten von Mäusen zeigen. Weiße Pfeile zeigen Zellen an, die sowohl für GFP als auch für Abstammungsmarker (α-SMA, GATA4, SOX9) positiv sind. Maßstabsbalken, 50 µm. m Quantitative genomische PCR-Analyse von Fledermaus-mtDNA in chimären E25-Schweineembryonen unter Verwendung von Fledermaus-Schwein-Zellverdünnungen. Die blaue Linie stellt den Fledermaus-mtDNA-Nachweislevel dar (1 Fledermauszelle pro 10,000 Schweinezellen). n Repräsentative IF-Färbungsbilder, die die Integration von GFP-exprimierenden biPSCs in Schweinekeimschichten zeigen. Weiße Pfeile zeigen Zellen an, die doppelt positiv für GFP und Abstammungsmarker sind (α-SMA, GATA4, SOX9). Maßstabsbalken, 50 µm.
Unsere Fledermaus-iPSCs (biPSCs) exprimierten Pluripotenzmarker sowohl auf RNA- als auch auf Proteinebene (ergänzende Abb. S1a, geb). Um die Pluripotenz zu bestätigen, haben wir die biPSCs mit h transfiziertOCT4-GFP und die Zellen zeigten eine starke GFP-Expression (ergänzende Abb. S1c), was auf die Aufrechterhaltung der Pluripotenz in biPSCs hinweist. Die biPSCs zeigten eine positive Färbung mit alkalischer Phosphatase (AP) und behielten nach 42 Passagen einen normalen Karyotyp (37 + XY) bei (ergänzende Abb. S1d, z). Sie konnten unter Feeder-freien Bedingungen auf Matrigel kultiviert werden (ergänzende Abb. S2a–c). In vitro könnten biPSCs Embryoidkörper bilden und sich dann in drei embryonale Keimschichten differenzieren (ergänzende Abb. S3a, geb). Die RNA-Sequenzierungsanalyse ergab eine deutliche Häufung der Genexpression zwischen den biPSCs und BEFs (Abb. 1d), mit hoher Expression von Pluripotenzgenen in biPSCs (ergänzende Abb. S3c). Diese Ergebnisse legen nahe, dass die biPSCs transgenfrei und pluripotent sind.
Um den Chimärismus und die Entwicklungsfähigkeit von biPSCs zu beurteilen, haben wir die biPSCs mit GFP/tdTomato markiert (ergänzende Abbildung). S4a–d) zur Zellverfolgung. Aufgrund der einfachen Manipulation wurden Hühnerembryonen als Plattform für heterologe Zellchimärismusstudien ausgewählt. Wir haben 5×10 gespritzt5 GFP-markierte biPSCs in Blutgefäße befruchteter Hühnerembryonen 52 Stunden nach dem Schlüpfen (Abb. 1e). Am Tag 6.5 (D6.5) wurde eine signifikante grüne Fluoreszenz im Kopf und in der Brust der chimären Hühnerembryonen beobachtet (Abb. 1f), bestätigt durch GFP-Färbung auf Paraffinschnitten (ergänzende Abb. S5a). Die gleichzeitige Färbung chimärer Hühnerembryoabschnitte mit GFP und Keimschichtmarkern bestätigte die Integration differenzierter biPSCs in alle drei Keimschichten (ergänzende Abb. S5b). Bei D9.5 war die grüne Fluoreszenz auf den Kopf chimärer Küken beschränkt (Abb. 1g).
Nach der Inkubation wurden zwei lebende chimäre Küken erhalten, und in ihren Gehirnen wurde eine signifikante GFP-Fluoreszenz beobachtet (Abb. 1h). Um die chimären Anteile in anderen Organen zu bestimmen, führten wir eine mitochondriale DNA-Analyse (mtDNA) durch. Unsere Ergebnisse zeigten einen unterschiedlichen Grad an Chimärismus in verschiedenen Geweben, der zwischen 1/100,000 und 1/1000 lag. Wir entdeckten die Integration grüner Zellen in die Gonaden der chimären Küken, wobei ein Küken keinen Chimärismus aufwies und das andere weniger als 1/10,000 Chimärismus aufwies (Abb. 1i). Darüber hinaus zeigte die Immunfluoreszenz (IF)-Färbung von Paraffinschnitten des Gehirns (Nr. 2) die Differenzierung von biPSCs in verschiedene neuronale Zelltypen (Abb. 1j). Diese Ergebnisse legen nahe, dass sich biPSCs im chimären Hühnermodell in funktionelle neuronale Zellen differenzieren können.
Um das Chimärenbildungspotenzial von biPSCs in zwei verschiedenen Säugetiermodellen, Mäusen und Schweinen, zu untersuchen, führten wir zunächst 10 GFP-markierte biPSCs in Mausembryonen im 8–16-Zellen-Stadium ein. Unter ihnen zeigten 98.09 % der injizierten Embryonen eine signifikante GFP-Fluoreszenz mit durchschnittlich 17.78 ± 0.95 biPSCs pro Blastozyste (ergänzende Abbildung). S6a–c). BiPSCs sind in die innere Zellmasse (ICM) chimärer Blastozysten integriert, mit durchschnittlich 2.93 ± 0.37 Zellen pro ICM (ergänzende Abbildung). S6d, z). Wir haben auch die Integration von biPSCs in das Trophektoderm der Maus untersucht und ihren Beitrag sowohl zum ICM als auch zum Trophektoderm aufgezeigt (ergänzende Abbildung). S7a). Bemerkenswerterweise zeigten einzelne biPSCs eine starke Proliferation innerhalb von Mäuseembryonen, wobei 31.25 % der chimären Embryonen eine Einzelzellproliferation zeigten (ergänzende Abbildung). S7b und Tabelle S1). Darüber hinaus führte die gleichzeitige Injektion von tdTomato-markierten biPSCs und GFP-markierten embryonalen Stammzellen (mESCs) der Maus zu einer räumlichen Überlappung der roten und grünen Fluoreszenzsignale (ergänzende Abbildung). S7c), was auf ein gemeinsames Entwicklungspotenzial zwischen den biPSCs und mESCs innerhalb der embryonalen Mikroumgebung hindeutet.
Als nächstes untersuchten wir das chimäre Potenzial von biPSCs in Mausembryonen nach der Implantation (Abb. 1e). Wir implantierten 553 chimäre Blastozysten, die von GFP-markierten biPSCs abgeleitet waren, in 33 Ersatzmäuse. Es kam zu erfolgreichen Schwangerschaften, und am Embryonaltag 8.5 (E8.5) wurden chimäre Embryonen gesammelt. Der Chimärismus wurde mittels genomischer PCR mit fledermausspezifischen Primern bestätigt (ergänzende Abb. S8a) und die mtDNA-Analyse ergab hohe Anteile an Chimärismus bei 8 von 10 Föten, wobei 3 bei über 1/1000 lagen (Abb. 1k). Der IF-Assay bei E10.5 zeigte eine signifikante GFP-Expression in chimären Proben (ergänzende Abb. S8b), wobei die Koexpression von GFP und Abstammungsmarkern auf die Differenzierung von biPSCs in alle drei Keimblätter in den chimären Mausembryonen hinweist (Abb. 1l). Unsere Ergebnisse zeigen das bemerkenswerte chimäre Potenzial von biPSCs in frühen Mausembryonen, obwohl dieses Potenzial mit fortschreitender Mausentwicklung abnehmen kann und wir daher keine interspezifischen Chimären bei Neugeborenen erreichen.
Anschließend untersuchten wir das chimäre Potenzial von biPSCs in Schweinembryonen. GFP-markierte biPSCs wurden in parthenogene E5-Schweineembryonen injiziert. Von den injizierten Embryonen zeigten 92.79 % GFP-Fluoreszenz mit durchschnittlich 14.14 ± 1.14 biPSCs pro Blastozyste (ergänzende Abbildung). S9a–c). Die IF-Färbung zeigte den Beitrag von biPSCs zum ICM mit durchschnittlich 3.78 ± 0.31 biPSCs pro ICM (ergänzende Abbildung). S9d, z). Diese Ergebnisse unterstreichen die Lebensfähigkeit und Integrationsfähigkeit von biPSCs in die ICM früher Schweineembryonen.
Wir haben auch das chimäre Potenzial von biPSCs in Schweinembryonen nach der Implantation untersucht. Wir transplantierten 610 in vitro befruchtete chimäre Blastozysten in 4 Ersatzsauen. Alle vier Leihmütterinnen wurden schwanger, was zur Entnahme von insgesamt 4 Embryonen zwischen E39 und E25 führte. Die MtDNA-Genotypisierung bestätigte, dass es sich bei 27 Embryonen um Chimären handelte, wobei der biPSC-Chimärismus 11/1 überstieg (Abb. 1m). Die IF-Färbung mit GFP-Antikörper auf Abschnitten dieser chimären Embryonen bestätigte außerdem das Vorhandensein von Fledermauszellen (ergänzende Abb. S10). Die IF-Färbung von Abstammungsmarkern zeigte die Differenzierung von biPSCs in alle drei embryonalen Keimschichten innerhalb der chimären Schweinembryonen (Abb. 1n). Diese Ergebnisse legen nahe, dass biPSCs in frühen Schweinembryonen eine höhere chimäre Effizienz aufweisen und die Fähigkeit besitzen, sich in vivo in alle drei embryonalen Keimschichten zu differenzieren.
In dieser Studie beobachteten wir die erfolgreiche Integration von biPSCs in Maus- und Schweinembryos vor der Implantation. Der bei Embryonen nach der Implantation beobachtete Chimärismus war jedoch begrenzt, was durch Faktoren wie evolutionäre Distanz, Schwangerschaftsdauer und Zellkonkurrenz beeinflusst werden kann. Frühere Studien haben gezeigt, dass Ratten-ESCs und Maus-iPSCs keinen Beitrag zu Schweinembryonen leisten konnten und dass der Beitrag menschlicher PSCs zu Schweinembryonen nach der Implantation sehr begrenzt war4. Diese Einschränkung kann auf die evolutionäre Distanz zwischen den Arten zurückgeführt werden5. Die evolutionäre Divergenz zwischen Fledermäusen und Mäusen fand schätzungsweise vor etwa 94 bis 81 Millionen Jahren statt (http://www.timetree.org/) kann die Effizienz des Xenochimismus erheblich beeinträchtigen.
In dieser Studie wurden das höchste Chimärismusverhältnis und die erfolgreiche Erzeugung lebender chimärer Küken beobachtet, wenn biPSCs in Hühnerembryonen injiziert wurden. Die chimären Küken zeigten eine Differenzierung von biPSCs in funktionelle Zellen im Nervensystem und lieferten wertvolle Einblicke in die Entwicklung und Differenzierung von biPSCs in heterologen Wirten. Hühnerembryonen sind ein bewährtes experimentelles Modell für Xenotransplantate und bieten ein praktisches System zur Untersuchung der Embryonalentwicklung und -differenzierung6. Der Hühnerembryo bietet ein wirksames Bewertungssystem für das In-vivo-Entwicklungspotenzial von Xenozellen, da frühere Studien die Pluripotenz menschlicher PSCs in Hühnerembryonen gezeigt haben7. Die gesteigerte Effizienz der Fledermaus-/Hühner-Chimäre könnte auf die anfänglich höheren injizierten Zellzahlen und das noch nicht vollständig entwickelte Immunsystem in Hühnerembryonen zurückzuführen sein8, was zu einer verringerten exogenen Zellabstoßung führte. Der eingeschränkte Gonaden-Chimärismus war auf die Injektion in einem weiter fortgeschrittenen Entwicklungsstadium (HH14–HH15) im Vergleich zu dem im Blastozystenstadium zurückzuführen. Das Testen verschiedener Injektionsstadien, -stellen und -methoden in zukünftigen Studien könnte das Ergebnis des Chimärismus verbessern.
Insgesamt haben wir transgenfreie biPSCs durch Elektroporation episomaler Plasmide erzeugt. Diese biPSCs zeigten bei drei Modelltieren chimäres Potenzial. Transgenfreie iPSCs sind von höherer Qualität, da sie keine Verzerrung der Zellliniendifferenzierung aufweisen und das Risiko der Tumorbildung und des Todes bei chimären Nachkommen vermeiden9,10,11. Sie können sich auch in ein breites Spektrum an Zelltypen oder Organoiden differenzieren12 unter entsprechenden Bedingungen. Unsere biPSCs haben das Potenzial, sich in vitro in relevante Organoide zu differenzieren, was mechanistische Untersuchungen der Langlebigkeit von Fledermäusen, der antiviralen Abwehr und der Echoortung ermöglicht.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Automobil / Elektrofahrzeuge, Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- ChartPrime. Verbessern Sie Ihr Handelsspiel mit ChartPrime. Hier zugreifen.
- BlockOffsets. Modernisierung des Eigentums an Umweltkompensationen. Hier zugreifen.
- Quelle: https://www.nature.com/articles/s41421-023-00587-3