Hefen, denen die C-5-Sterol-Desaturase Erg3 fehlt, weisen eine auffallend erhöhte Toleranz gegenüber Aescin auf
Wir stellten die Hypothese auf, dass, wenn die Aescin-Toxizität durch direkte Wechselwirkung mit Ergosterol vermittelt wird, die Mutanten der Ergosterol-Biosynthese sich in der Aescin-Toleranz unterscheiden könnten. Es wird angenommen, dass Sterine eine Schlüsselrolle bei der Aufrechterhaltung der Homöostase der Plasmamembrandynamik spielen28, jedoch fehlen Hefestämme Enzyme, die die letzten fünf Schritte der Ergosterol-Biosynthese katalysieren (Erg2, Erg3, Erg4, Erg5 oder Erg6; Abb. 1b und S1) sind lebensfähig. Aufgrund der Substratpromiskuität der letztgenannten Enzyme in der Ergosterol-Biosynthese akkumulieren diese Deletionsmutanten eine Mischung von Sterolstrukturen, die sich von Ergosterin in der Anzahl und Position der Doppelbindungen im Sterol-B-Ring und der Sterol-Seitenkette unterscheiden29. Wir verwendeten Mikrotiterplattenkulturen des Wildtypstamms BY4741 (WT) und Mutanten aus der Yeast Deletion Collection30, um das Wachstum der Ergosterol-Biosynthesemutanten in komplexem reichhaltigem Medium (YPD) in Gegenwart und Abwesenheit von Escin zu beurteilen (Abb. 1C). Die Sterolextraktion und die Analyse mittels Gaschromatographie-Massenspektrometrie bestätigten, dass sich Ergosterol in den Mutantenstämmen nicht anreicherte (Abb. S2 machen S3). Die minimale Hemmkonzentration (MHK) von Escin für den WT-Stamm im YPD-Medium betrug 150 µg/ml. Ähnliche MICs wurden für beobachtet erg2Δ, erg4Δ, erg5Δ und erg6Δ Dehnungen, jedoch Wachstum der erg3Der Δ-Stamm war bis zur höchsten getesteten Konzentration (1000 µg/ml; Abb. S4).
Transkriptomanalyse
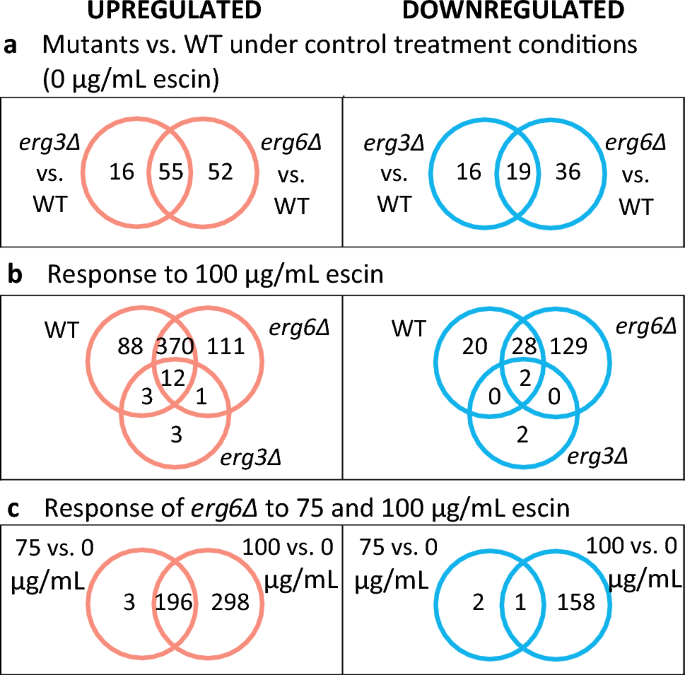
Um den Einfluss von Escin auf WT-Zellen und den Mechanismus der Escin-Toleranz weiter zu untersuchen erg3Δ, wir haben die Transkriptome von WT analysiert, erg3Δ und erg6Δ-Zellen, 0 Stunde lang mit 100 oder 1 µg/ml Escin in YPD behandelt, in Kulturen im Schüttelkolben. Der erg6Der Stamm Δ wurde in dieses Experiment einbezogen, da dieser Stamm viele ähnliche Phänotypen aufweist erg3Δ29, weist aber keine erhöhte Aescin-Toleranz auf (MHK 150 µg/ml; Abb. 1C). Im Rahmen dieses Experiments haben wir auch die Transkriptome von analysiert erg3Δ und erg6Δ-Zellen, die 75 Stunde lang mit 1 µg/ml Escin behandelt wurden.
K bedeutet, dass die Clusterbildung der 2000 Gene mit den variabelsten Expressionsniveaus in Abb. dargestellt ist. S5, mit vollständigen Details zur Gen-Set-Anreicherungsanalyse, die in den Zusatzinformationen (SI) enthalten sind. Cluster-B-Gene (n = 82) werden im Allgemeinen auf einem höheren Niveau exprimiert erg3Δ und erg6Δ im Vergleich zu WT unter allen Bedingungen und wie erwartet ist dieser Cluster an Genen angereichert, die sich auf die Sterolbiosynthese, den Steroltransport, den Siderophortransport und die Regulierung der Transkription durch Glucose beziehen. Cluster-A-Gene (n = 821) sind in WT und herunterreguliert erg6Δ als Reaktion auf die Aescin-Behandlung. Dieser Cluster ist mit Genen angereichert, die mit der Ribosomenbiogenese und der RNA-Verarbeitung in Zusammenhang stehen. Die Gene der Cluster C (n = 897) und D (n = 200) sind in WT und im Allgemeinen hochreguliert erg6Δ als Reaktion auf die Aescin-Behandlung, wobei die Gene von Cluster D stärker hochreguliert sind als von Cluster C. Cluster D ist an Genen angereichert, die mit dem Trehalose-, Mannose-, Fruktose- und Glutamatstoffwechsel sowie der Glykolyse und der Zellwandorganisation in Zusammenhang stehen. Cluster D ist außerdem reich an Genen, die mit Reaktionen auf osmotischen, oxidativen, Temperatur- und Hungerstress in Zusammenhang stehen. Cluster C ist reich an Genen, die sich auf die späte Nukleophagie, den Lipidkatabolismus, die Schwefelassimilation, den Tricarbonsäurezyklus und die Gluconeogenese beziehen.
Differentiell exprimierte Gene (DEGs) mit ≥ 2-facher differentieller Expression zwischen den interessierenden Bedingungen (Abb. 2) werden im Folgenden ausführlicher besprochen, mit vollständigen Details zur Gensatzanreicherung in SI.
Auswirkung von ERG3 machen ERG6 Deletion im Hefetranskriptom
Der Vergleich der Transkriptome von Stämmen unter Kontrollbedingungen zeigt interessante Unterschiede zwischen den Mutantenstämmen und WT. Löschung von ERG6 führt zu einer größeren Anzahl von DEGs (n = 162) als ERG3 Löschung (n = 106). In beiden Mutanten sind hochregulierte DEGs mit Genen angereichert, die mit der Sterol-Biosynthese und dem Sterol-Transport zusammenhängen, einschließlich des Sterol-Sensors und des Transkriptionsaktivators UPC2 (Feige. S6). Im Gegensatz dazu sind nur sehr wenige Gene, die an der Sphingolipid- und Phospholipidversorgung beteiligt sind, hochreguliert (YSR3, RSB1, FAA2 machen ELO2 in beiden Stämmen und zusätzlich Surxnumx in erg6Δ; Feige. S7), obwohl für Sterol-Biosynthese-Mutanten große Veränderungen in der Sphingolipid-Zusammensetzung berichtet wurden31.
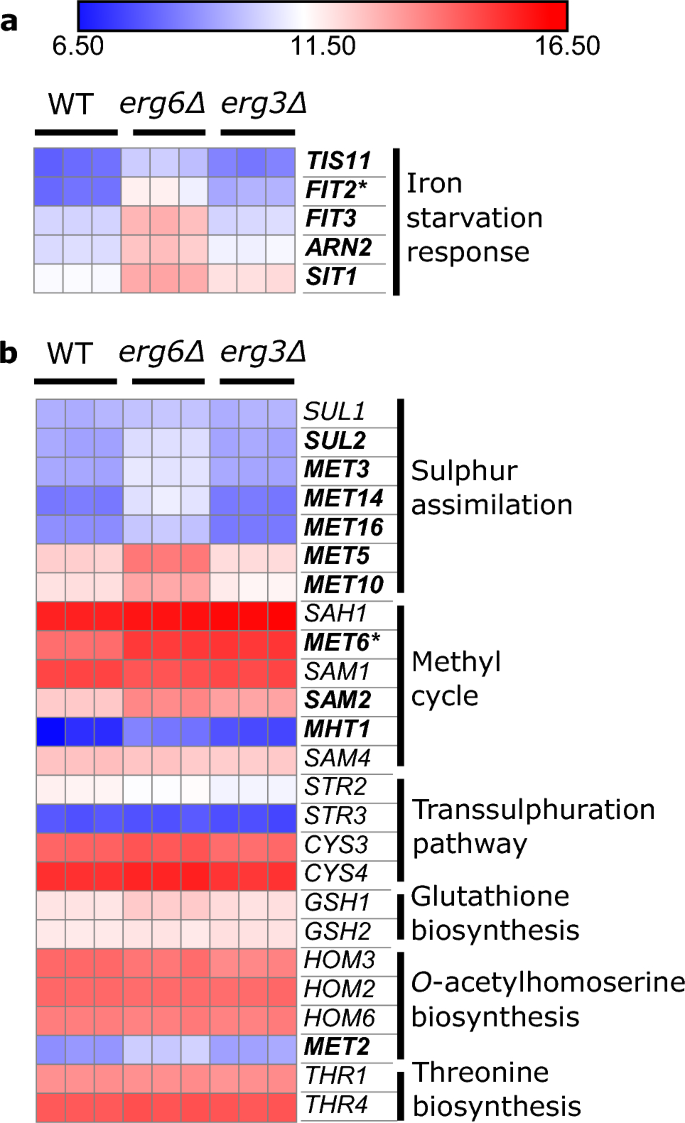
Auch die Mannoprotein-Gene der Zellwand der anaeroben Reaktion sind in beiden Stämmen stark hochreguliert (Abb. S8). Mehrere Schritte der Ergosterol-Biosynthese erfordern Sauerstoff und Eisen als Cofaktoren, und es gibt eine komplexe Wechselwirkung zwischen den regulatorischen Reaktionen auf Ergosterin, Sauerstoff und Eisen32,33,34,35. Eine Reihe von Genen, die auf Eisenmangel reagieren36 sind hochreguliert erg6Δ (FIT2, FIT3, RNA2, SIT1, TIS11), aber nicht erg3Δ, relativ zur WT (Abb. 3A). Kürzlich wurde berichtet, dass sich der eisenempfindliche Transkriptionsfaktor Aft1, der normalerweise zwischen Zytoplasma und Zellkern pendelt, in den Vakuolen von Ergosterin-verminderten Zellen ansammelt upc2Δ Zellen37. Unsere Daten deuten darauf hin, dass dieser Signalweg in unterschiedlichem Ausmaß beeinflusst werden kann erg6Δ und erg3Δ.
Das erg6Der Δ-Stamm weist eine erhöhte Expression von Schwefelassimilationsgenen und Eisenmangelreaktionsgenen auf. Expression von Genen im Zusammenhang mit (a), Eisenhomöostase und (b), der Methionin-Biosyntheseweg, unter Kontrollbedingungen Einheiten log2(CPM + 4). Deutlich; Gen um ≥ 2-fach hochreguliert erg6Δ gegen WT. Sternchen; Gen um ≥ 2-fach hochreguliert erg3Δ versus WT (FDR ≤ 0.1).
Auch die Gene des Methioninstoffwechsels sind hochreguliert erg6Δ, aber nicht erg3Δ (Abb. 3B). Insbesondere sind die hochregulierten Gene mit dem Schwefelassimilationsweg verbunden (SUL2, MET3, MET14, MET16, MET5, MET10) und der Methylzyklus (MET6, SAM2, MHT1), das S-Adenosylmethionin (SAM) erzeugt (Abb. S9). Das Erg6-Enzym nutzt den Methyldonor SAM, um Zymosterol zu methylieren38und ein Erg6-Es wurde zuvor festgestellt, dass ein defizitärer Stamm SAM ansammelt, was auf einen verringerten SAM-Verbrauch zurückgeführt wurde39.
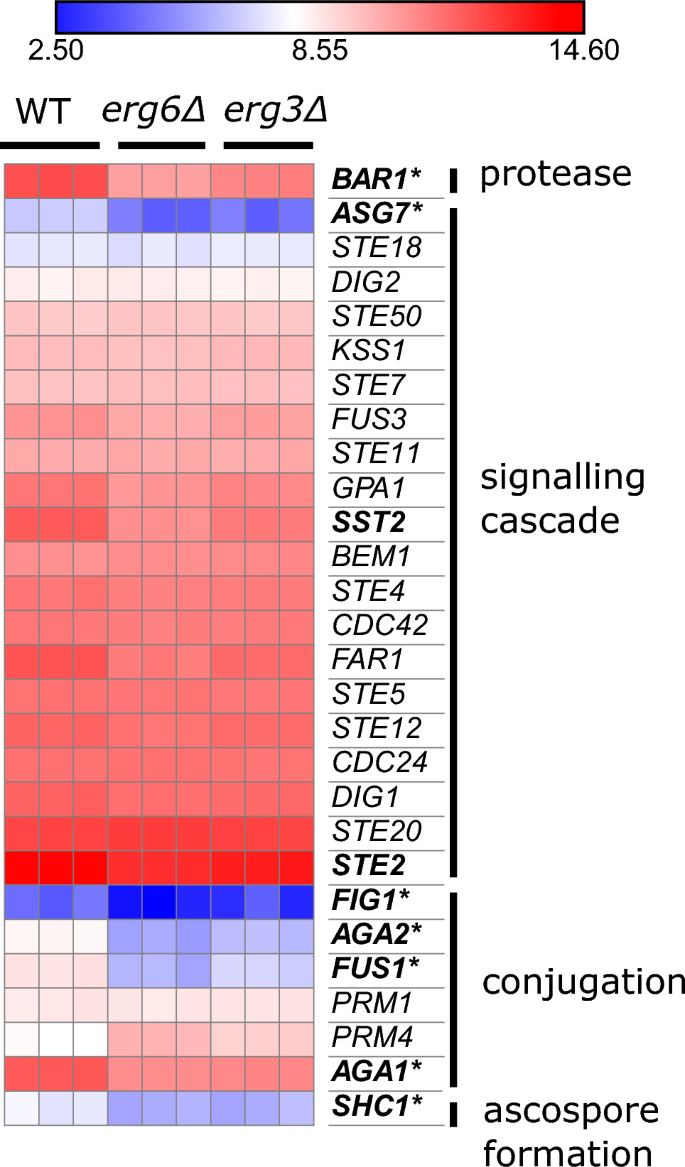
Beide Stämme regulieren eine Reihe von Genen herunter, die an der Paarung beteiligt sind (Abb. 4). Hierbei handelt es sich hauptsächlich um Gene, die an der Konjugation zwischen sich paarenden Schmoo-Spitzen beteiligt sind (FUS1, AGA1, AGA2, ABB1, SHC1) und eine deutlich verringerte Paarungseffizienz wurden bereits für Ergosterin-Biosynthese-Mutanten beschrieben40,41.
Das erg3Δ und erg6Δ-Stämme weisen eine verringerte Expression von Paarungsgenen auf. Expression von Paarungsgenen, Einheiten log2(CPM + 4). Deutlich; im Vergleich ≥ 2-fach herunterreguliert erg6Δ 0 µg/ml Escin gegenüber WT 0 µg/ml Escin. Sternchen; in den Vergleichen ≥ 2-fach herunterreguliert erg3Δ 0 µg/ml Escin versus WT 0 µg/ml Escin (FDR ≤ 0.1).
Beide Stämme exprimieren auch NCE102 auf einem niedrigeren Niveau als WT (42 % des WT-Niveaus in erg3Δ und 40 % des WT-Niveaus in erg6Δ). Obwohl Nce102 hauptsächlich an den Eisosomen der Plasmamembran lokalisiert ist, wurde kürzlich berichtet, dass es eine Rolle bei der Vakuolenfusion spielt42, und Vakuolen sind in vielen Ergosterol-Biosynthese-Mutantenstämmen stark fragmentiert, einschließlich erg3Δ, obwohl nicht erg6Δ43,44,45.
Es ist bemerkenswert, dass mehrere DEGs zwischen den Mutanten und WT eine unbekannte Funktion haben; 20 und 29 hochregulierte DEGs und 10 und 13 herunterregulierte DEGs erg3Δ und erg6Δ bzw.
Transkriptom-Reaktionen auf die Aescin-Behandlung
Als Reaktion auf 100 µg/ml Escin wurden der WT und erg6Δ-Stämme regulieren die Gene 473 bzw. 494 hoch (Abb. 2B). Diese DEGs sind mit Genen angereichert, die auch mit Reaktionen auf Austrocknung, osmotischen Stress, oxidativen Stress, Temperatur, chemische Behandlung und Veränderungen des Nährstoffgehalts (SI) verbunden sind. Behandlung von erg6Δ-Zellen mit 75 µg/ml Escin führten zu einer Hochregulierung von 199 Genen (Abb. 2C). Diese 199 DEGs sind mit Genen angereichert, die mit Reaktionen auf oxidativen Stress, chemischen Stress und Hunger (SI) verbunden sind.
Die Auswirkung der Aescin-Behandlung auf die Gene des zentralen Kohlenstoffstoffwechsels ist in Abb. dargestellt. S10. Als Reaktion auf 100 μg/ml Escin wurden sowohl WT als auch erg6Δ reguliert Gene im Zusammenhang mit der Biosynthese von Trehalose, Glykogen und Glycerin stark hoch. Es wird angenommen, dass das Glukosepolymer Glykogen in erster Linie als Speicherkohlenhydrat fungiert, das kaum Einfluss auf den inneren osmotischen Druck der Zelle hat46,47. Es wird angenommen, dass dem Disaccharid Trehalose eine Schlüsselrolle beim Schutz der Struktur von Lipidmembranen und Proteinen zukommt, indem es Wasser aus Lipiddoppelschichten und Proteinoberflächen verdrängt und die Aggregation denaturierter Proteine verhindert, die deren anschließende Neufaltung verhindern würden47. Glycerin fungiert als wichtiger Osmolyt bei hyperosmotischem Stress und bildet das Rückgrat von Phospholipiden und Speicherlipid-Triacylglycerinen48. Die Hochregulierung der Biosynthese von Trehalose, Glykogen und Glycerin ist Teil einer allgemeinen Reaktion auf Umweltveränderungen47,48,49. Darüber hinaus sind sowohl WT als auch erg6Δ-Stämme regulieren Gene hoch, die für Proteine des GID-Komplexes (Glucose Induced degradation Deficient) kodieren, der die Gluconeogenese zugunsten der Glykolyse negativ reguliert, indem er die Polyubiquitinierung und den Abbau der Fructose-1,6-bisphosphatase initiiert50 (Feige. S11).
Als Reaktion auf 100 μg/ml Escin wurden sowohl WT als auch erg6Δ reguliert auch Gene hoch, die Enzyme des γ-Aminobuttersäure (GABA)-Shunt-Signalwegs kodieren (Abb. S12), das Glutamat über GABA abbaut. Bei Hefen hat sich gezeigt, dass dieser Weg wichtig für die Toleranz gegenüber Hitze und oxidativem Stress ist51.
Die hochregulierten DEGs sind auch mit Genen angereichert, die mit der Zellwandorganisation, einschließlich Sporulation und Chitinbiosynthese, zusammenhängen (Abb. S13). Durch die Wechselwirkung zwischen den Pfaden der mitogenaktivierten Proteinkinase (MAPK) von High Osmolarity Glycerol 1 (Hog1) und Cell Wall Integrity (CWI) ist es schwierig, spezifische Belastungen zu erkennen, die die Hochregulierung dieser Gene auslösen, ohne defekte Mutanten zu verwenden in bestimmten Zweigen dieser Signalwege52,53,54.
Viele autophagiebezogene Gene sind in WT und hochreguliert erg6Δ als Reaktion auf die Aescin-Behandlung (Abb. S14), darunter zentrale Phagosomenkomponenten und Proteine, die am Weg vom Zytoplasma zur Vakuole beteiligt sind und Hydrolasen selektiv an die Vakuole abgeben55. Als Reaktion auf 100 μg/ml Escin werden mehr Autophagie-bezogene Gene um das ≥ 2-fache hochreguliert erg6Δ Dehnung als WT (11 vs. 7).
Das erg6Der Δ-Stamm reguliert auch mehr Gene als WT als Reaktion auf 100 μg/ml Escin herunter (159 vs. 50), und diese DEGs sind mit Genen angereichert, die mit der Ribosomenbiogenese und -translation zusammenhängen. Diese Prozesse erfordern erhebliche Mengen an Energie56, und ihre Herunterregulierung ist Teil einer allgemeinen Stressreaktion, bei der Ressourcen eher für die Stressresistenz als für die Zellproliferation verwendet werden57,58.
Wenn Aescin Ergosterol aus Zellen absondert oder die Sterol-Erkennung durch Wechselwirkung mit Ergosterol oder einem Sterol-Sensor hemmt, können wir mit einer Transkriptionsänderung in Sterol-Biosynthese-Genen rechnen, z ERG2 machen ERG11 als Reaktion auf eine Aescin-Behandlung. Diese Gene werden durch die Sterolsensoren und Transkriptionsaktivatoren Upc2 und Ecm22 als Reaktion auf den Ergosterolgehalt reguliert59,60. Eine differenzielle Expression von Ergosterol-Biosynthesegenen wird nicht beobachtet (Abb. S6), obwohl es eine statistisch signifikante, aber geringfügige Hochregulierung von gibt UPC2 in jedem Stamm (1.3-, 1.9- und 1.5-fach für WT, erg6Δ und erg3Δ bzw. Δ). Eine kleine Anzahl von Genen, die mit der Sphingolipidversorgung zusammenhängen, werden als Reaktion auf Aescin in WT und hochreguliert erg6Δ (Abb. S7), obwohl zu beachten ist, dass ein Großteil der Sphingolipid-Regulierung posttranslational erfolgt31.
Im Gegensatz zu den großen Transkriptomveränderungen, die für den WT und beobachtet wurden erg6Δ-Dehnungen, erg3Der Stamm Δ reguliert als Reaktion auf 19 µg/ml Escin nur 100 Gene hoch und reguliert 4 herunter (Abb. S15). Die meisten dieser DEGs werden auch im WT und/oder unterschiedlich ausgedrückt erg6Δ-Stamm als Reaktion auf die Aescin-Behandlung, mit Ausnahme von HAP4 (Kodierung eines Atmungsregulators), YOL163W (gilt als nicht funktionsfähig), YGL088W (unbekannte Funktion) und die Zellwand-Mannoprotein-Gene DAN1 machen DAN4.
Das erg3Das Δ-Transkriptom ist nicht auf den Stress der Aescin-Behandlung vorbereitet
Um zu untersuchen, ob die Escin-Reaktions-DEGs im WT-Stamm bereits unterschiedlich exprimiert werden erg3Δ unter Kontrollbedingungen, möglicherweise „Priming“ erg3Δ für den Stress der Escin-Behandlung haben wir DEGs in den Vergleichen WT 100 mit 0 µg/ml Escin verglichen und erg3Δ 0 µg/ml Escin versus WT 0 µg/ml Escin (Abb. S16A). Unter Kontrollbedingungen ist die erg3Der Δ-Stamm weist eine erhöhte Expression von 18 Genen auf, die im WT als Reaktion auf die Aescin-Behandlung ebenfalls hochreguliert werden, und eine geringere Expression von 5 Genen, die im WT als Reaktion auf die Aescin-Behandlung herunterreguliert werden. Eine Heatmap, die die Expressionsniveaus dieser überlappenden Gene zeigt, ist in Abb. enthalten. S16B. Die meisten dieser Gene werden auch in der DNA unterschiedlich reguliert erg6Δ Dehnung unter Kontrollbedingungen und die erg6Der Δ-Stamm weist nicht den Phänotyp der erhöhten Escin-Toleranz auf. Die Ausnahmen sind PAI3 (kodiert für einen zytoplasmatischen Proteinase-A-Inhibitor), SRL3 (kodiert für einen Zellzyklusregulator), HED1 (kodiert für ein Meiose-spezifisches Protein), MAL31 (kodiert für eine Maltosepermease), ARG3 (kodiert für Ornithin-Carbamoyltransferase), COS111 (kodiert für ein mitochondriales Protein), Gene YBR090C machen YKR075C unbekannter Funktion und Gene YJR037W machen YJL195C von denen es unwahrscheinlich ist, dass sie funktionelle Proteine kodieren.
Insgesamt zeigen diese Daten, dass Escin keinen Einfluss auf das Wachstum und einen vernachlässigbaren Einfluss auf das Transkriptom von hat erg3Δ Zellen. Daran dürfte es nicht liegen erg3Δ-Zellen sind bereits auf die durch die Aescin-Behandlung induzierten Belastungen vorbereitet, was möglicherweise auf die chemische Zusammensetzung ihrer Membransterole zurückzuführen ist.
Das Vormischen von Aescin mit Ergosterol, jedoch nicht mit Brassicasterol, verhindert eine durch Aescin vermittelte Wachstumshemmung
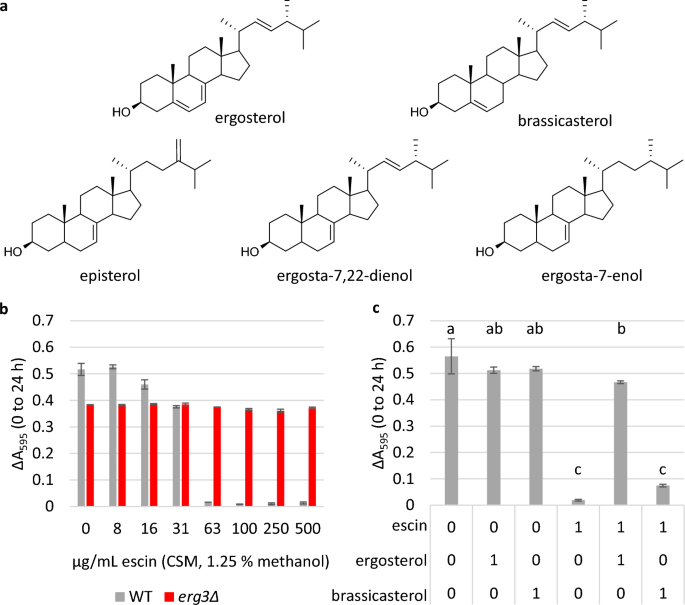
Um die Hypothese zu untersuchen, dass Aescin durch direkte Wechselwirkung mit Ergosterol Toxizität vermittelt, nicht jedoch mit den darin enthaltenen veränderten Sterolen erg3Δ, wir haben die Auswirkung der Behandlung von WT-Zellen mit Escin allein oder mit Escin, das mit Ergosterol im Molverhältnis 1:1 vorgemischt wurde, bewertet. Es wurde angenommen, dass, wenn Ergosterol direkt mit Aescin interagiert, eine Aescin-Ergosterol-Mischung aufgrund der geringeren Zugänglichkeit der Aescin-Saponine einen geringeren Einfluss auf das WT-Wachstum hätte als Aescin allein. Bemerkenswert ist, dass Hefezellen unter aeroben Bedingungen keine exogenen Sterole importieren37.
Das Experiment wurde unter Verwendung von Mikroplattenkulturen und CSM-Medien (Complete Supplement Mix) durchgeführt, im Gegensatz zu YPD, da der genaue Gehalt an CSM definiert ist, während YPD ein reichhaltiges Medium ist, das komplexe Moleküle aus Hefeextrakt enthält, die auch mit Aescin interagieren können und/oder Sterole. Frühere Studien haben gezeigt, dass Ergosta-7,22-dienol das vorherrschende Sterin in Hefen ohne Erg3 ist, wobei sich auch Episterol und Ergosta-7-enol anreichern31,43. Als der Sterolgehalt durch Gaschromatographie-Massenspektrometrie überprüft wurde (Abb. S2 machen S3), die relative Retentionszeit des größten Sterolpeaks im erg3Das Δ-Gesamtionenchromatogramm entsprach früheren Berichten für Ergosta-7,22-dienol, als der Stamm entweder in YPD oder CSM gezüchtet wurde.
Die MHK von Escin in CSM-Medien wurde für WT-Zellen auf 63 μg/ml bestimmt (Abb. 5B); 2.4-fach niedriger als bei YPD. Mittlerweile Wachstum der erg3Der Δ-Stamm war bis zur höchsten getesteten Konzentration (500 μg/ml) ungehemmt. Wenn Escin mit Ergosterol im Molverhältnis 1:1 vorgemischt und die Mischung den Zellen zugesetzt wurde, wurde das Wachstum des WT-Stammes in Gegenwart von 63 μg/ml Escin vollständig wiederhergestellt (Abb. 5C). Durch das Vormischen von Escin mit Ergosterol wurde auch der Prozentsatz der mit dem membranundurchlässigen Farbstoff Propidiumiodid gefärbten Zellen wiederhergestellt, um die Behandlungsmengen zu kontrollieren (Abb. S17), was auf eine verringerte Zellpermeabilität und/oder eine erhöhte Lebensfähigkeit der Zellen hinweist.
Wiederherstellung des Wachstums, wenn Aescin mit Ergosterol vorgemischt wird, jedoch nicht mit Brassicasterol. (a) Struktur von Ergosterol (dem vorherrschenden Sterol in S. cerevisiae Plasmamembran), Brassicasterol (ein Pflanzensterin) und Sterole, die sich nachweislich in der Plasmamembran ansammeln erg3Δ-Stamm (Episterol, Ergosta-7,22-dienol, Ergosta-7-enol). (b) und (c), Wachstum in Mikrotiterplattenkulturen, angezeigt durch ΔA595 (0–24 Std.). Drei biologische Replikate ± Standardabweichung (SD). In (b) beträgt die MHK von Escin in CSM-Medien 63 μg/ml für WT, was 2.4-fach niedriger ist als in reichem Komplexmedium. In (c) wurden WT-Zellen in Gegenwart von 0 oder 63 μg/ml Escin (1.25 % Methanol) allein oder vorgemischt mit Ergosterol oder Brassicasterol im Molverhältnis 1:1 gezüchtet. Nur Ergosterol- und Brassicasterol-Kontrollen wurden einbezogen. Statistik: einfaktorielle ANOVA mit Post-hoc-Tukey-HSD-Test, Bedingungen, die nicht durch denselben Buchstaben verbunden sind, unterscheiden sich signifikant (p ≤ 0.05).
Die darin enthaltenen Sterole erg3Δ sind nur begrenzt verfügbar, daher haben wir zum Vergleich auch das Wachstum beurteilt, wenn Escin mit dem Phytosterol Brassicasterol vorgemischt wurde, das ebenfalls eine Doppelbindung im B-Ring aufweist, obwohl sich diese zwischen C-5 und C- befindet. 6 (Abb. 5A). Im Gegensatz zu dem wiederhergestellten Wachstum, das beobachtet wurde, wenn Escin mit Ergosterol vorgemischt wurde, wurde keine Wiederherstellung des WT-Wachstums beobachtet, wenn Escin mit Brassicasterol im Molverhältnis 1:1 vorgemischt wurde (Abb. 5c).
Dies stützt die Hypothese, dass Aescin direkt mit Ergosterol interagiert und dass kleine Veränderungen in der Sterolstruktur einen großen Einfluss auf die Stärke der Sterol-Saponin-Wechselwirkungen haben können.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Automobil / Elektrofahrzeuge, Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- ChartPrime. Verbessern Sie Ihr Handelsspiel mit ChartPrime. Hier zugreifen.
- BlockOffsets. Modernisierung des Eigentums an Umweltkompensationen. Hier zugreifen.
- Quelle: https://www.nature.com/articles/s41598-023-40308-0