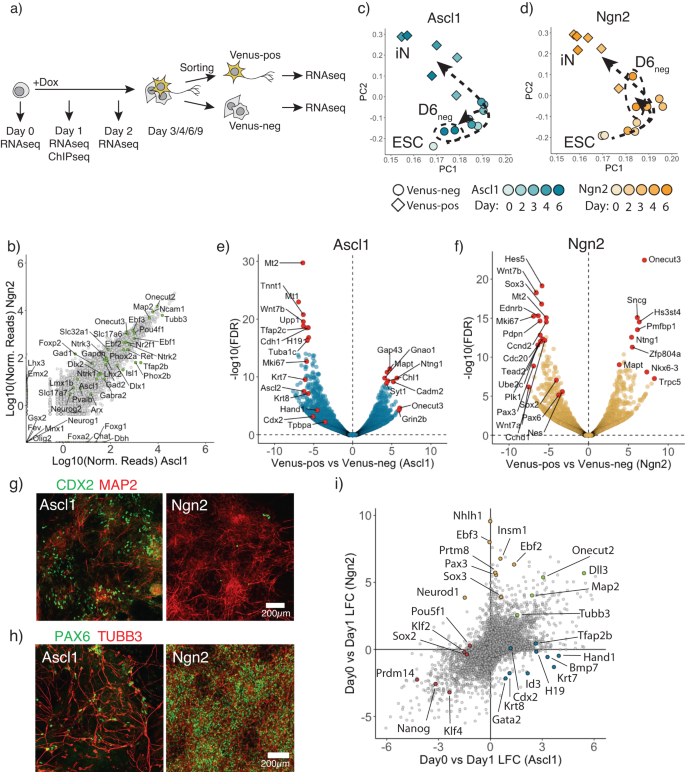
Ascl1 und Ngn2 konvertieren ESC in iN, erzeugen jedoch unterschiedliche Nebenlinien
Die ektopische Expression von Ascl1 oder Ngn2 in embryonalen Stammzellen (ESCs) der Maus reicht aus, um die terminale Differenzierung in Neuronen zu induzieren28. Dennoch sind die Unterschiede im Übergangsmechanismus zu Neuronen sowie möglichen Nebenpopulationen nicht gut charakterisiert6,31. Um diese Zelltypumwandlung im Detail zu untersuchen, haben wir klonale ESC-Zelllinien generiert, die rtTA und TetO-Ascl1 oder TetO-Ngn2 exprimieren27,28 (Ergänzende Abb. 1a). Nach der Zugabe von Doxycyclin (Dox) werden ESCs schnell in induzierte Neuronen umgewandelt: Ascl1 und Ngn2 produzieren Zellen, die den neuronalen Marker TUBB3 exprimieren und ab Tag 3 bzw. Tag 2 eine neuronale Morphologie aufweisen (ergänzende Abbildung). 1a). Um das neuronale Schicksal in diesen Zelllinien zu melden, haben wir das panneuronale Markergen Mapt an seinem C-Terminus endogen mit dem fluoreszierenden Protein Venus markiert27 (Ergänzende Abb. 1b) und führte eine zeitaufgelöste Massen-RNAseq nach Dox-Induktion durch. Die Zellen wurden in Venus-positive Neuronen und Venus-negative Zellpopulationen sortiert (Abb. 1a) ab Tag 3. Wie bereits berichtet28,30Sowohl Ascl1 als auch Ngn2 führen zu ähnlichen iN-Zellidentitäten (Abb. 1b; Ergänzende Abb. 1c–e). Daher sind die anfänglichen ESC- und terminalen iN-Zustände zwischen Ascl1- und Ngn2-induzierten Umwandlungen sehr ähnlich (ergänzende Abbildung). 1b unten). Dies steht im Einklang mit früheren Beobachtungen, dass Transkriptome trotz Unterschieden in der anfänglichen Transkriptionsreaktion zusammenlaufen, um die iN-Bildung voranzutreiben6 und folgen einer insgesamt ähnlichen Flugbahn zwischen ESC und iN in der PCA-Analyse (Abb. 1c, d; Ergänzende Abb. 1g).
a Schematischer Überblick über den Versuchsaufbau. b Streudiagramm zum Vergleich der Genexpression an Tag 6 zwischen Ascl1- und Ngn2-Venus-positiven Zellen mit verschiedenen neuronalen Subtyp-spezifischen Markern, die grün dargestellt sind. c, d Hauptkomponentenanalyse der zeitaufgelösten Massen-RNAseq nach Ascl1 (c) oder Ngn2 (d). Jeder Datenpunkt entspricht der einzelnen Zeitpunktreplikation. Die Farbintensität wird am Tag nach der Induktion angezeigt. Die Form entspricht der Mapt-Venus-Reporter-Hochregulierung. Pfeile zeigen die Flugbahn, die Zellen nach dem Ascl1 nehmen (c) oder Ngn2-Induktion (d). e, f Vulcano-Plot zum Vergleich der Genexpression zwischen Venus-positiven und -negativen Populationen am Tag 6 nach der Induktion von Ascl1 (e) oder Ngn2 (f). Rote Kreise kennzeichnen die am häufigsten hoch- oder herunterregulierten Gene sowie Beispielgene, die im Trophoblasten markiert sind (e) oder NSC-Linien (f). g Repräsentative immungefärbte Zellen für einen Trophoblastenmarker CDX2 und einen neuronalen Marker Map2 am Tag 6 nach der Induktion von Ascl1 oder Ngn2. Trophoblastenmarker wurden erst nach der Ascl1-Induktion exprimiert, nicht jedoch Ngn2. h Repräsentative immungefärbte Zellen für einen NSC-Marker PAX6 und einen neuronalen Marker TUBB3 am Tag 6 nach der Induktion von Ascl1 oder Ngn2. NSC-Marker wurden erst nach der Ngn2-Expression exprimiert, nicht jedoch Ascl1. i Streudiagramm zum Vergleich der Genexpressionsänderungen zwischen Ascl1 und Ngn2 am Tag 1 nach der Induktion. Hervorgehobene Kreise sind Beispielgene, die neuronale Marker sind, die in beiden exprimiert werden (grün), Trophoblasten-Ascl1-spezifische Marker (blau), NSC-Ngn2-spezifische Marker (gelb) und pluripotenzbezogene Gene (rot). Quelldaten werden als Quelldatendatei bereitgestellt.
Um Zellen, die kein iN bilden, genauer zu untersuchen, haben wir uns auf die Mapt-Venus-negativen Zellen konzentriert, die unvollständige oder alternative Differenzierungsergebnisse darstellen könnten. Mapt-Venus-negative Zellen, die durch Ascl1 erzeugt wurden, gruppieren sich näher an den anfänglichen ESC-Populationen in PCA-Plots als Ngn2-induzierte Mapt-negative Zellen (Abb. 1c). Dies ist jedoch nicht darauf zurückzuführen, dass eine Population undifferenzierter ESC erhalten bleibt, da nur eine marginale Anzahl von Zellen in der Endpopulation den ESC-Marker NANOG exprimieren (ergänzende Abbildung). 1g) und NANOG und OCT4 werden in der Mapt-Venus-Population nicht exprimiert (ergänzende Abbildung). 1h). Um den von Ascl1 generierten alternativen Zelltyp zu identifizieren, haben wir PanglaoDB verwendet32 unter Verwendung von Genen, die am Tag 6 unterschiedlich zwischen Venus-negativen und -positiven Populationen exprimiert werden (Abb. 1e, f; Ergänzende Abb. 2a). Interessanterweise produziert Ascl1 Zellen, die Trophoblastmarker exprimieren, wie z Hand1, CDx2, Tpbpa, Krt8 (Feige. 1e, g; Ergänzende Abb. 2b–g) mit mesenchymaler Morphologie, die in Ngn2-induzierten Kulturen nicht vorhanden waren (Abb. 1g, Ergänzende Abb. 2b, c, f). Wir haben diese Zellen als induzierte Trophoblasten-ähnliche Zellen (iT) bezeichnet. Interessanterweise sind viele davon Krt8, CDx2 Positive iTs schienen zweikernig zu sein, was auf eine Mehrkernbildung ähnlich der Entwicklung der Trophoblastenlinie in vivo zurückzuführen sein könnte (ergänzende Abb. 2e)33. Die Induktion von iT könnte darauf zurückzuführen sein, dass Ascl1 den bHLH-Transkriptionsfaktor Ascl2, einen Treiber für die Trophoblastenlinie, nachahmt. Sowohl Ascl1 als auch Ascl2 sind evolutionär nahe beieinander und teilen nahezu identische DNA-Bindungsdomänen und binden ähnliche E-Box-Motive (ergänzende Abb. 2h–k)33,34,35,36,37. Um schließlich klonale Effekte der verwendeten Zelllinie auszuschließen, wiederholten wir diese Experimente im Hintergrund einer alternativen Maus-ESC-Linie, E14. Wir führten rtTA über einen Piggybac-Transposonvektor ein und exprimierten Ascl1 über einen Dox-induzierbaren viralen Vektor27 und erzeugte 24 von Einzelzellen abgeleitete Klone. Alle Klone zeigten die Bildung von iN und iT, was darauf hindeutet, dass die Bildung von iTs ein reproduzierbares Nebenprodukt der ektopischen Ascl1-Expression in Maus-ESCs ist (ergänzende Abb. 3).
Im Gegensatz zur Ascl1-Induktion induziert Ngn2 Mapt-negative Zellen, die NSC-Marker exprimieren, wie z Sox2, Pax3, Pax6, Nes (Feige. 1f, h; Ergänzende Abb. 2a, 4a, b), wie zuvor beschrieben21,38. Darüber hinaus könnte die Ngn2-Reprogrammierung in Gegenwart von FGF2 und EGF im NSC-ähnlichen Zustand (iNSC) blockiert werden und ist vom Notch-Signalweg abhängig39,40 (Ergänzende Abb. 4c, d). Im Gegensatz dazu beobachteten wir keine Hochregulierung der NSC-Marker während der Ascl1-induzierten Differenzierung (Abb. 1h, Ergänzende Abb. 4b, c). Um zu sehen, ob Ngn2 den iNSC-Zustand als proliferatives Zwischenprodukt nutzen kann, haben wir ab Tag 4 nach der Induktion Zellen in Gegenwart oder Abwesenheit von Cytosin β-D-Arabinofuranosid (AraC) differenziert, um sich teilende Zellen zu inaktivieren (ergänzende Abb. 4e). Tatsächlich reduziert die Zugabe von AraC die von Ngn2 produzierten iNs drastisch, während Ascl1 gegenüber der AraC-Behandlung unempfindlich war, was darauf hindeutet, dass während der Ascl1-induzierten iN-Reprogrammierung kein kontinuierlich proliferatives Zwischenprodukt vorhanden ist (ergänzende Abbildung). 4e). Zusammenfassend lässt sich sagen, dass Ascl1 und Ngn2, obwohl Ascl1 und Ngn2 mESC in ähnliche iN-Subtypen umwandeln, unterschiedliche zusätzliche alternative Zelllinien produzieren, was darauf hindeutet, dass trotz identischer Anfangs- und Endpopulationen Unterschiede bestehen, die wir weiter verstehen wollten (ergänzende Abbildung). 1b unten).
Ascl1 und Ngn2 initiieren Pfade mit unterschiedlichen Transkriptionsprogrammen
Um ein besseres Verständnis der unterschiedlichen Transkriptionsreaktionen zu erhalten, die durch Ascl1 und Ngn2 hervorgerufen werden, führten wir am ersten Tag Bulk-RNAseq und ChiPseq durch (Abb. 1a). Sowohl Ascl1 als auch Ngn2 induzieren allgemeine neuronale Marker wie z Tubb3, Map2 und Onecut2 und allgemeine Pluripotenzmarker wie herunterregulieren Nanog, Klf4 (Feige. 1i). Darüber hinaus induziert Ascl1 stark nachgeschaltete Ziele Tfap2b, Lmx1b, während Ngn2 stark hochreguliert Neurod1, Nhlh1 (Feige. 1i). Darüber hinaus reguliert Ascl1 Trophoblasten-Abstammungsmarker hoch, z. Krt7/8, Hand1, während Ngn2 die Expression von NSC-bezogenen Genen hochreguliert Pax3 und Sox3 (Feige. 1i). Interessanterweise beobachteten wir, dass Zellen zu Beginn der Neuprogrammierung sowohl für neuronale als auch für Trophoblastenmarker positiv sind (ergänzende Abb. 5a, b). Darüber hinaus haben wir die verfügbaren scRNAseq-Daten erneut analysiert6 für Tag 2 der Umwandlung von ESC in iN durch Ascl1 und Ngn2 und konnte auch Zellen beobachten, die sowohl für neuronale als auch für Trophoblastenmarker positiv waren (ergänzende Abb. 5c, d). Dies legt nahe, dass Ascl1 beide Abstammungslinien gleichzeitig induzieren kann, die später in iN- oder iT-Zellen aufgelöst werden (ergänzende Abb. 5b).
Wie von Aydin und Kollegen berichtet6, Ascl1 und Ngn2 zeigen unterschiedliche Präferenzen für E-Box-Motive, was wiederum zur Aktivierung unterschiedlicher Untergruppen von Genen führt. Tatsächlich haben wir die unterschiedliche Bindung von Ascl1 und Ngn2 in ESC bestätigt (ergänzende Abbildung). 6a). Darüber hinaus sehen wir, dass Ascl1- und Ngn2-Zielgene unterschiedlich exprimiert werden, ebenso wie Gene, die an den verschiedenen alternativen Abstammungslinien beteiligt sind, z. B. Hand1, CDx2, Krt8 or Neurod1, Pax3bzw. (Ergänzende Abb. 6b). Interessanterweise bindet Ascl1 Trophoblasten-bezogene Gene auch in MEF, allerdings ohne iT-Induktion (ergänzende Abb. 7a, b). Zusätzlich zur iN-Induktion führt die Überexpression von Ascl1 in MEF jedoch zur Bindung von Ascl1 an die Gene der Skelettmuskulatur und zur Induktion von Myozyten7,16. Tatsächlich bindet Ascl1 in ESC auch stark an Gene, die mit der Abstammungslinie der Skelettmuskulatur in Zusammenhang stehen, z. Myod1, Myog, Myf3, Tnnt2 (Ergänzende Abb. 7c). Wir konnten jedoch keine Hochregulierung dieser Gene beobachten (ergänzende Abbildung). 7d). Daher ist es verlockend zu spekulieren, dass der zelluläre Kontext, wie z. B. zelltypspezifische Histonmodifikationen oder Transkriptionsfaktoren, die Wahl der induzierten alternativen Abstammungslinie beeinflusst. Somit werden zusätzlich zur Induktion des neuronalen Transkriptionsprogramms zusätzliche Gene gebunden und transkribiert, was zur Bildung alternativer Abstammungslinien führt. Dieses Transkriptionsprogramm außerhalb des Ziels hängt von der Überexpression des Transgens, z. B. Ascl1 oder Ngn2, sowie vom zellulären Kontext, z. B. ESC oder MEF, ab.
Um die allgemeinen Prinzipien der frühen Transkriptionsreaktion eingehender zu untersuchen, verwendeten wir die STRING-Datenbank, um das Genregulationsnetzwerk (GRN) von Ascl1- und Ngn2-hochregulierten differentiell exprimierten Genen (DEGs) einen Tag nach der Induktion zu untersuchen (ergänzende Abb. 8a, b). Sowohl Ascl1- als auch Ngn2-DEGs bilden Netzwerke, die drei verschiedene Gengruppen enthalten. Interessanterweise teilen Ascl1- und Ngn2-GRNs Gruppen mit Genen, die am RNA- und Sterolstoffwechsel beteiligt sind, was auf die Stoffwechselverschiebung während der Umwandlung zurückzuführen ist41,42. Darüber hinaus bildet Ngn2 ein stärker vernetztes Netzwerk als Ascl1, was darauf hindeutet, dass Ngn2 eine kohärentere Transkriptionsreaktion auslöst als Ascl1 (ergänzende Abbildung). 8c).
Um die Unterschiede genauer zu analysieren, haben wir uns die zentralsten verbundenen Knoten beider GRNs angesehen (ergänzende Abbildung). 8d, e). Im Gegensatz zu Ascl1 induziert Ngn2 Gene, die die neuronale Differenzierung steuern Neurog2, Neurod1, Neurog1, Lhx2/3, Otx2sowie Gene, die an der Differenzierung neuronaler Stammzellen beteiligt sind: Notch1, Hes5, Pax3, wobei GRN um sie herum zentriert wird (ergänzende Abb. 8e–g). Interessanterweise ist bekannt, dass Ngn2, Neurod1 und Ngn2 ESC in iN umwandeln können43. Somit kann eine solche positive Rückkopplungsschleife zusammen mit der Induktion starker Abstammungstreiber und stark vernetzter GRN eine robustere iN-Umwandlung und eine schnellere Unabhängigkeit von der anfänglichen Induktion der Kassette ermöglichen. Um diese Hypothese zu testen, induzierten wir Zellen für 2, 4 oder 6 Tage (ergänzende Abb. 8h). Tatsächlich beobachten wir, dass eine effiziente iN-Umwandlung mit Ascl1 auf der anhaltenden Expression von Ascl1 beruht, während Ngn2 die Umwandlung bereits nach 2 Tagen der Induktion effizient induziert (ergänzende Abbildung). 8h).
Zusammenfassend lässt sich sagen, dass Ascl1 und Ngn2 unterschiedliche Transkriptionsprofile und daraus resultierende mechanistische Unterschiede bei der Umwandlung von ESC in iN binden und hervorrufen. Während die Ascl1-Induktion auf der anhaltenden Expression von Ascl1 beruht, induziert Ngn2 ein insgesamt kohärenteres Netzwerk, das eine effiziente und schnelle Neuprogrammierung sowie die Induktion eines proliferativen Zwischenprodukts ermöglicht.
Funktionsverlust-CRISPR/Cas9-Screening zur Identifizierung genetischer Abhängigkeiten für die Umwandlung von ESC in iN
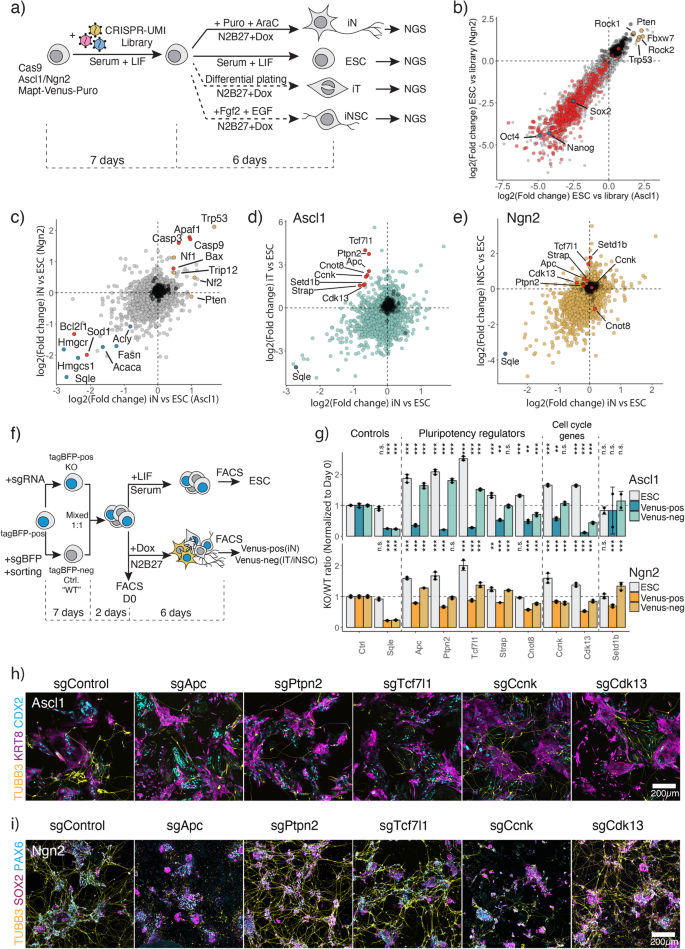
Um einen mechanistischen Überblick über die zugrunde liegenden Unterschiede zwischen Ascl1- und Ngn2-induzierter ESC-Reprogrammierung zu erhalten, führten wir ein CRISPR/Cas9-Funktionsverlust-Screening durch. Unser Ziel war es, zwei Aspekte dieser Umwandlung zu untersuchen: erstens die genetischen Abhängigkeiten der durch Ascl1 und Ngn2 induzierten neuronalen Umwandlung und zweitens die Gene aufzuklären, die an der Bildung alternativer Zustände beteiligt sind (Abb. 2a). Zu diesem Zweck wurden ESC mit einer retroviralen CRISPR-UMI-sgRNA-Bibliothek infiziert, die etwa 27,000 Leitfäden für 6630 Gene (4 Leitfäden pro Gen) und 108 nicht zielgerichtete Leitfäden enthielt44. Anschließend differenzierten wir ESC für 6 Tage und reichern es mit AraC und einer Puromycin-Behandlung auf iN an (Abb. 2a, Ergänzende Abb. 1b, 9a). Darüber hinaus haben wir Abhängigkeiten für die iT- und iNSC-Differenzierung bewertet (Abb. 2a, siehe „Methoden“). Für eine erste Bewertung der Screening-Ergebnisse verglichen wir die Erschöpfung bekannter wesentlicher Elemente in ESC mit der in der Bibliothek (Abb. 2b). Wir haben tatsächlich eine starke Erschöpfung der gemeinsamen essentiellen Gene beobachtet, wie sie von Hart et al. definiert wurden.45. Darüber hinaus sind Gene im zentralen Pluripotenznetzwerk (Nanog, Sox2, Pou5f1) gehören zu den wichtigsten Genen, während Tumorsuppressoren darunter sind Trp53, Fbxw7, Rock1 bereichern die ESC stark (Abb. 2b), was zeigt, dass unsere Bibliothek beim Gen-Targeting und der Aufdeckung von Gen-Knockout-Phänotypen wirksam ist.
a Experimenteller Überblick über den CRISPR-Cas9-Bildschirm. b Vergleichende Analyse der Gen-Knockout-Effekte zwischen Ascl1- und Ngn2-Transgenen, die ESC tragen. Der Unterschied in der Leithäufigkeit wurde zwischen nichtinduziertem ESC am Tag 13 nach der Bibliotheksinfektion berechnet (a) im Vergleich zum Bibliotheksplasmidpool. Punkte stellen Gene dar; Die Achse zeigt die Erschöpfung des LFC in jeder Zelllinie. Rot stellt essentielle Kerngene dar, wie von Hart et al. definiert.45, blau – Kernpluripotenzgene, gelb – Tumorsuppressoren. c Vergleichende Analyse der Gen-Knockout-Effekte von Ascl1- oder Ngn2-induziertem iN im Vergleich zu ESC. Rote Punkte stehen für Apoptose-bezogene Gene, gelbe für Tumorsuppressor-Gene und blaue für Cholesterin-Biosynthese-Gene. d Vergleichende Analyse der Gen-Knockout-Effekte von Ascl1-induziertem iN oder iT im Vergleich zu ESC. Rote Punkte stellen Gene dar, die zur Validierung ausgewählt wurden. Sqle ist eine Positivkontrolle für eine starke Erschöpfung. e Vergleichende Analyse der Gen-Knockout-Effekte von Ngn2-induziertem iN oder iNSC im Vergleich zu ESC. Rote Punkte stellen Gene dar, die zur Validierung ausgewählt wurden (d). Sqle ist eine Positivkontrolle für eine starke Erschöpfung. f Experimenteller Überblick über die Treffervalidierung von (d). g FACS-basierte Validierung der Treffer. Der Balken stellt das normalisierte Verhältnis zum anfänglichen Mischungsverhältnis am Tag 0 dar. N = 3 unabhängige biologische Replikate. Das Balkendiagramm zeigt den Mittelwert ± SD. p-Werte, die oben angegeben sind, wurden durch eine einfaktorielle ANOVA gefolgt von Dunnetts Mehrfachvergleichstest (zweiseitig) unter Verwendung des Strg-Verhältnisses als Kontrolle bestimmt. „ns“ nicht signifikant, „*“ p < 0.05, „**“ p < 0.01, „***“ p <0.001. h, i Immunfärbung der Knockout-Zellen für den neuronalen Marker Tubb3 und alternative Abstammungsmarker KRT8/CDX2 für Ascl1-induziertes iT (h) und SOX2/PAX6 für Ngn2-induziertes iNSC (i). Quelldaten werden als Quelldatendatei bereitgestellt.
Um häufige genetische Abhängigkeiten der iN-Bildung zu identifizieren, verglichen wir die Leithäufigkeit im ESC mit der iN bei D6 (Abb. 2c). Leitfäden, die den stärksten Abbau zeigen, zielen beispielsweise auf den Fettsäurestoffwechsel und die Sterolbiosynthese ab, wie z Fasn, Hmgcr, Hmgcs1, Platz, ein wesentlicher Bestandteil des neuronalen Stoffwechsels (Abb. 2c)41,42. Darüber hinaus unterliegen Zellen während der iN-Differenzierung einem hohen Stressniveau aufgrund der Lipidperoxidation und können durch Apoptose oder Ferroptose zum Zelltod führen46,47. Daher finden wir proapoptotische Gene, Casp3/9 und Bax anreichernd, während antiapoptotische Gene wie z Bcl2l1 und Sod1 nehmen an der Bevölkerung ab (Abb. 2c; Ergänzende Abb. 9b). Darüber hinaus kommt es zum Knockout von Tumorsuppressoren, z. Trp53, Nfxnumx, Nfxnumx, ermöglicht eine höhere iN-Umwandlung. Somit liefert dieser Bildschirm genetische Abhängigkeiten bei der Umwandlung von ESC in iN bei hoher Auflösung.
Um unterschiedliche genetische Abhängigkeiten zwischen iN und alternativen Zuständen aufzudecken, wurde die relative sgRNA-Häufigkeit mit einem besonderen Fokus auf Gene korreliert, die in iN abgereichert und in den alternativen Zuständen angereichert sind. Wir konzentrierten uns auf unterschiedliche Abhängigkeiten zwischen dem Ascl1-induzierten iN und iT (Abb. 2d). Wir haben Gene zur Validierung anhand der folgenden Kriterien ausgewählt: (1) Gene, die geringe Auswirkungen auf ESC zeigen (−2 <LFC <2); (2) Gene, die eine Anreicherung in iT zeigen (LFC > 1.5); (3) Gene mit vermindertem iN-Gehalt (LFC < −0.5). Wir haben festgestellt, dass Gene wichtig für die Regulierung des Pluripotenznetzwerks und die Differenzierung von ESC sind: Tcf7l1, Ptpn2, apc, Armbänder, Cnot848,49,50,51,52,53,54sowie Zellzyklusgene Ccnk und Cdk1355,56 sind für die Ascl1-induzierte iN-Umwandlung erforderlich und hemmen gleichzeitig die iT-Bildung. Im Gegensatz dazu hatte das Ausschalten dieser Treffer kaum oder gar keinen Einfluss auf die Ngn2-gesteuerte Umwandlung in Neuronen (Abb. 2d, e).
Um die Ergebnisse zu validieren, führten wir einen Konkurrenztest durch. Um eine interne Kontrolle zu implementieren, haben wir einen konstitutiven tagBFP-Vektor in unsere Zelllinie eingeführt und einen Klon mit einer stabilen tagBFP-Expression abgeleitet. Anschließend wurden die Zellen mit der sgRNA infiziert, die im Screen die stärkste Depletion zeigte. Unabhängig davon haben wir eine isogene Kontrollpopulation generiert, indem wir tagBFP mit einem Kontrollleitfaden gegen tagBFP gezielt und nach tagBFP sortiert habenneg Zellen. Anschließend wurden Knockout- und Kontrollzellen im Verhältnis 1:1 gemischt und für eine weitere Passage aufbewahrt, um sie an die gleichen Bedingungen anzupassen, bevor die Zellen zur Induktion ausplattiert wurden (Abb. 2f). Nach 6 Tagen der Induktion beurteilten wir die iN Mapt-Venus-positive Population sowie die Venus-negative Population, entsprechend der Bildung von iT/iNSC-Populationen (Abb. 2f, g). Darüber hinaus beurteilten wir die Eignung der Knockouts in den ESC-Zellen, während wir ESC weitere 6 Tage lang wachsen ließen (Abb. 2f). Wie vorausgesehen, Platz war in iN-, iT- oder iNSC-Populationen essentiell, nicht jedoch in ESC-Zellen, was die Screening-Ergebnisse widerspiegelt (Abb. 2c–e, g). Ebenso das Ausschalten pluripotenzbezogener Gene apc, Tcf7l1 und Ptpn2 verhinderte die Bildung von iNs nach Induktion mit Ascl1 und ermöglichte gleichzeitig die Bildung von iT-Zellen (Abb. 2g). Wir bestätigten den Phänotyp weiter durch Immunfärbung am Tag 6 nach der Induktion (Abb. 2h, ich). Das Ausschalten der Treffer führte dazu, dass Ascl3 keine TUBB1-positiven Neuronen erzeugte, während weiterhin CDX2+- und KRT8+-Zellen erzeugt wurden. Interessanterweise fehlt einigen Zellen, die neuronenspezifisches TUBB3 exprimieren, die neuronale Morphologie, was auf die Aktivierung neuronaler Gene hinweist, aber nicht in der Lage ist, einen endgültigen iN-Zustand zu etablieren (ergänzende Abbildung). 9c). Ebenso das Targeting von Ccnk or Cdk13 reduzierte die Anzahl der iNs bei der Ascl1-Induktion stark. Im Gegensatz zu Ascl1 wird die Ngn2-iN-Umwandlung bei Verlust von nicht beeinträchtigt Tcf7l1, Ptpn2 und Cdk13 und Ngn2 kann weiterhin sowohl Neuronen als auch neuronale Stammzellen erzeugen, die durch SOX2 und PAX6 gekennzeichnet sind (Abb. 2g–ich). Jedoch, apc und Ccnk Knockouts beeinflussten tatsächlich die durch Ngn2 induzierte Bildung von iN und iNSC. Bemerkenswerterweise haben wir zusätzlich mehrere andere Gene validiert, die unterschiedliche Abhängigkeiten zwischen der Ascl1- und Ngn2-Induktion zeigen (Supplementary Abb. 9d–h). Insgesamt haben unsere parallelen CRISPR/Cas9-Funktionsverlust-Screens mehrere gemeinsame und unterschiedliche Abhängigkeiten zwischen Ascl1- und Ngn2-induzierter gerichteter Differenzierung zu iNs aufgedeckt.
Schnelle Herunterregulierung des Pluripotenznetzwerks nach Ascl1-Induktion
Da das Screening Gene identifizierte, die an der Aufrechterhaltung des Pluripotenznetzwerks beteiligt sind, als wesentlich für die Ascl1-induzierte iN-Bildung, untersuchten wir genauer, wie Ascl1 und Ngn2 das Pluripotenznetzwerk zerlegen. Zu diesem Zweck verwendeten wir eine Ingenuity-Pathway-Analyse unter Verwendung aller differentiell exprimierten Gene einen Tag nach der Ascl1- oder Ngn2-Induktion (ergänzende Abb. 10a–d, Feige. 1i). Wie zuvor zeigte die Analyse der kanonischen Signalweganreicherung von Ingenuity, dass sowohl Ascl1 als auch Ngn2 Signalwege im Zusammenhang mit der Cholesterinbiosynthese induzieren (ergänzende Abbildung). 10b, d). Interessanterweise konzentriert sich die Transkriptionsreaktion von Ascl1 auf die Herunterregulierung des ESC-Pluripotenznetzwerks und die Selbsterneuerung (ergänzende Abbildung). 10a, b).
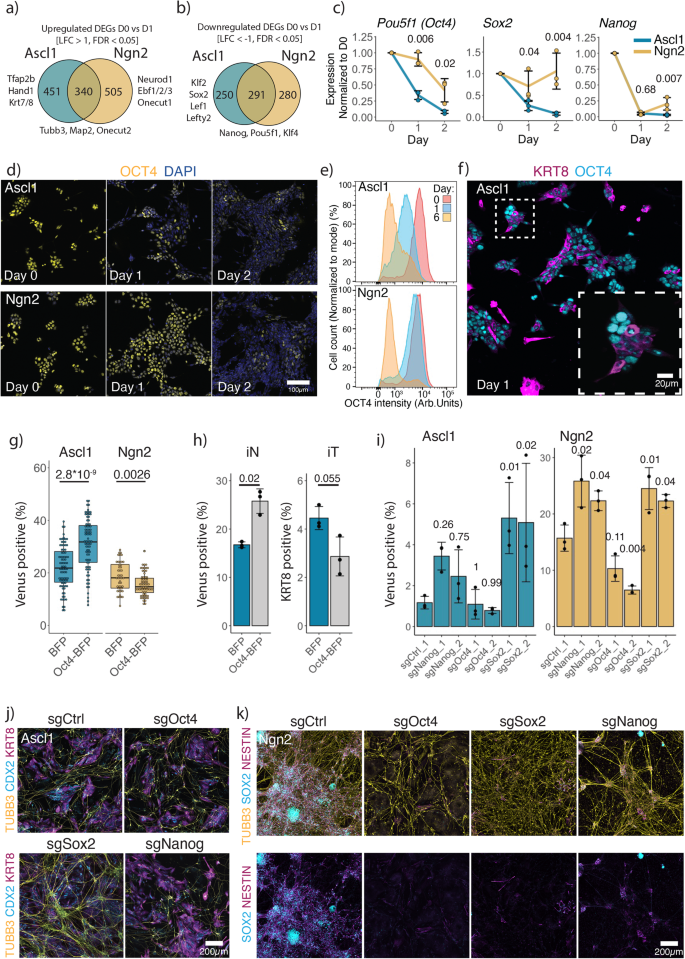
Anschließend haben wir die DEGs in Ascl1- oder Ngn2-spezifische oder gemeinsame DEGs unterteilt und eine KEGG-Signalweganreicherungsanalyse durchgeführt (Abb. 3a, b; Ergänzende Abb. 10e). Interessanterweise reguliert Ascl1 Gene hoch, die an der zellulären Seneszenz und dem Austritt aus dem Zellzyklus beteiligt sind, wie z Cdkn1a, Cebpa, Cebpb (Ergänzende Abb. 8d, g)57,58, und reguliert auch weitere Gene herunter, die an der Pluripotenz beteiligt sind, z. Pou5f1 (kodiert für Protein OCT4), Klf2, Sox2, Links1, Linkshänder2, im Vergleich zu Ngn2 (Abb. 3b, Feige. 1i, Ergänzende Abb. 10e). Als nächstes untersuchten wir daher die Dynamik des Abschaltens des Pluripotenznetzwerks (PPN) nach der Induktion von Ascl1 und Ngn2. Dabei haben wir uns auf die drei zentralen Pluripotenzgene konzentriert: Pou5f1 (4. Okt.), Sox2, Nanog (Feige. 3c, Feige. 1i). Nanog wird bei ähnlicher Kinetik zwischen Ascl1 und Ngn2 herunterreguliert (Abb. 3c, Feige. 1i, Ergänzende Abb. 11a, b). Im Gegensatz, Sox2 Die Expression bleibt nach der Ngn2-Induktion erhalten, da ein NSC-Genregulierungsnetzwerk aufgebaut wird, während Ascl1-induzierte Zellen die Expression von verlieren Sox2 (Feige. 3c, Feige. 1i, Ergänzende Abb. 11a, b). In ähnlicher Weise führt die Ascl1-Induktion zu einem schnellen Verlust der Oct4-Expression, während die Ngn2-Induktion zu einer allmählichen Herunterregulierung von Oct4 führt (Abb. 3c–e, Feige. 1i). Darüber hinaus korrespondiert der Verlust von Oct4 mit der Hochregulierung des Trophoblastenmarkers KRT8 (Abb. 3f).
a, b Anzahl der Ascl1-, Ngn2-spezifischen oder häufig hochregulierten (a) und herunterreguliert (b) Gene aus Abb. 1i. c qPCR-Daten der Expression der zentralen Pluripotenzgene (normalisiert auf die Expression von Actin und Tag 0). Es werden Linien durch den Mittelwert von gezogen n = 3 biologisch unabhängige Proben; Fehlerbalken zeigen ± SD an. Über, p-Werte des zweiseitigen Welch-T-Tests mit zwei Stichproben, der PPN-Gene vergleicht, die zum gegebenen Zeitpunkt zwischen Ascl1 und Ngn2 exprimiert wurden. d Repräsentative Immunfärbungen für die OCT4-Dynamik nach Ascl1- oder Ngn2-Induktion. e Quantifizierung der OCT4-Expression mittels intrazellulärer Immunfärbung, gefolgt von FACS. f Repräsentative Immunfärbung für den Pluripotenzmarker OCT4 und den Trophoblastmarker KRT8 am Tag 1 nach der Ascl1- oder Ngn2-Induktion. g Effizienz der iN-Bildung bei Vorliegen einer OCT4-Überexpression. Jeder Punkt stellt einen einzelnen ESC-Klon dar, der ein Überexpressionskonstrukt enthält. Die Effizienz wird anhand des Prozentsatzes der Mapt-Venus-Population gemessen. Boxplots geben das 25. und 75. Perzentil als Grenzen der Box mit der mittleren Mittellinie an; Whiskers zeigen Minima/Maxima in einem 1.5-fachen Abstand des IQR vom 25. und 75. Perzentil an. p-Wert des oben angegebenen zweiseitigen Welch-T-Tests bei zwei Stichproben. h Effizienz der iN-Bildung, gemessen am Prozentsatz der Mapt-Venus-exprimierenden Zellen, und iT-Bildung, Prozentsatz der auf KRT8 immungefärbten Zellen der polyklonalen Population, die Oct4-BFP überexprimiert. Das Balkendiagramm zeigt den Mittelwert von n = 3 unabhängige biologische Replikate mit ± SD; Die p-Werte wurden mithilfe des zweiseitigen Welch-T-Tests bei zwei Stichproben berechnet. i Effizienz der iN-Bildung bei akutem Ausfall der Kernpluripotenz. Die Effizienz wird anhand des Prozentsatzes der Mapt-Venus-Population gemessen. Das Balkendiagramm zeigt den Mittelwert von n = 3 biologisch unabhängige Proben mit ± SD. pDie oben angegebenen Werte wurden mithilfe einer einfaktoriellen ANOVA und anschließendem Mehrfachvergleichstest (zweiseitig) von Dunnett unter Verwendung von sgCtrl als Kontrolle berechnet. j Repräsentative Immunfärbungen von (i) für durch Ascl1 induziertes iN und iT. j Repräsentative Immunfärbungen von (i) für durch Ngn2 induziertes iN und iNSC. Quelldaten werden als Quelldatendatei bereitgestellt.
Um die funktionelle Relevanz des Verlusts von Oct4 zu testen, haben wir Oct4 konstitutiv überexprimiert und mehrere klonale mESC-Zelllinien generiert (Abb. 3g, h). Während die Überexpression von Oct4 zusammen mit Ngn2 zu einer Verringerung der iN-Population führt, erhöht die Koexpression von Oct4 und Ascl1 die Effizienz der iN-Bildung (Abb. 3g, h). Die iT-Bildung wiederum wird in Gegenwart von Oct4 behindert (Abb. 3h). Während der Entwicklung hemmt Oct4 die Differenzierung der Trophektoderm-Linie, und ein schneller Verlust von Oct4 ist mit der Hochregulierung der Trophektoderm-Marker verbunden59,60. Somit schafft der schnelle Verlust von Oct4 zulässige Bedingungen für die Bildung von iT-Linien, die mit der Bildung von iN konkurrieren.
Angesichts der kinetischen Unterschiede bei der Herunterregulierung von PPN nach der Ascl1- oder Ngn2-Induktion haben wir getestet, ob eine erzwungene Störung des PPN zusammen mit der Differenzierung die Umwandlung von ESC in iN beeinflussen würde. Als Pou5f1 (4. Okt.), Sox2 und Nanog sind für ESC essentiell (Abb. 2b), wir infizierte Zellen mit gezielten Leitfäden Pou5f1 (4. Okt.), Sox2 und Nanog 2 Tage vor der Induktion von Ascl1 oder Ngn2 (Abb. 3i–k). Interessanterweise führt die Ascl1-Induktion zur Bildung von iN und iT beim Ausschalten aller Pluripotenzfaktoren (Abb. 3j). Nach dem Targeting von Oct4 sehen wir kaum bis gar keine Wirkung, da Oct4 bei der Ascl1-Induktion schnell verloren geht (Abb. 3i). Allerdings Knockout von Nanog oder Sox2 begünstigt die iN-Bildung, was darauf hindeutet, dass eine zusätzliche Störung des PPN die Entstehung des iN-Zustands unterstützt (Abb. 3i, j). Im Gegenzug Knockout von Sox2 verhinderte die Bildung von iNSC durch Ngn2 und zeigte dies Sox2 wird vom PPN zum Genregulationsnetzwerk des NSC umfunktioniert (Abb. 3i, k). Ebenso eine Störung von beidem Nanog or Pou5f1 (4. Okt.) führte ebenfalls zum Verlust von iNSC (Abb. 3k). Stattdessen beobachteten wir die Bildung von primitivem Endoderm und Trophoblast nach dem Verlust von Nanog or Pou5f1 (4. Okt.) bzw. (Abb. 3k, Ergänzende Abb. 11c, d), im Einklang mit dem Ergebnis des Verlusts dieser Faktoren während der Entwicklung59,60,61.
Diese Daten zeigen zusammen mit den im CRISPR-Screen identifizierten unterschiedlichen Abhängigkeiten, dass die Induktion von Ascl1 und Ngn2 zu ausgeprägten funktionellen Unterschieden beim Verlassen des Pluripotenzzustands von ESC führt. Die Ascl1-Induktion führt zu einer effizienten Abschaltung des PPN und später zur Induktion des iN- oder iT-Gennetzwerks, während Ngn2 das PPN nicht vollständig herunterreguliert und stattdessen beide Netzwerke überlagert, wodurch Gene für die Induktion von NSCs umfunktioniert werden.
Tcf7l1 ist für den Zellzyklusausgang erforderlich, um Ascl1-iNs zu generieren
Als nächstes konzentrierten wir uns auf die Ascl1-induzierte PPN-Abschaltung. Bei der Ascl1-gesteuerten MEF-zu-iN-Transdifferenzierung erleichtert Myt1l die iN-Bildung durch Unterdrückung des anfänglichen Fibroblasten-GRN sowie der alternativen Myoblastenlinie16,62. Angesichts der schnellen Herunterregulierung von PPN haben wir getestet, ob Myt1l in ähnlicher Weise für die Umwandlung von ESC in iN erforderlich ist. Unser Bildschirm zeigte dies jedoch nicht an Myt1loder seine Paraloge Myt1 und St18, sind für die Umwandlung von ESC in iN erforderlich (ergänzende Abbildung). 12a) sowie Validierungsexperimente mit zusätzlichen sgRNAs (Ergänzende Abb. 12b). Im CRISPR-Bildschirm haben wir identifiziert Tcf7l1 (T-Zellfaktor/Lymphoid-Enhancer-Faktor, auch bekannt als Tcf3), ein Repressor von PPN, der für die iN-Bildung durch Ascl1 essentiell ist. Wir haben das bestätigt Tcf7l1 Knockout-Phänotyp in einem separaten E14-Hintergrund und schloss aus, dass der Verlust von Tcf7l1 die Expression des Ascl1-Transgens beeinflusste (ergänzende Abb. 12c–e). Tcf7l1 ist auf den mehreren Promotoren von PPN-verwandten Genen positioniert und unterdrückt deren Expression bei der ESC-Differenzierung schnell48,63,64und das Fehlen von Tcf7l1 stabilisiert den ESC-Zustand64. Daher wollten wir testen, ob Tcf7l1 vor oder nach Beginn der Zelltypkonvertierung benötigt wird, und sekundäre Effekte aufgrund der Stabilisierung des PPN vor der Konvertierung ausschließen. Zu diesem Zweck markierten wir Tcf7l1 N-terminal mit einem Auxin-induzierbaren Degron, infizierten Zellen mit einem lentiviralen Vektor, der osTIR1 (F-Box E3-Ubiquitin-Ligase, abgeleitet von Oryza sativa) trug, und erzeugten von Einzelzellen abgeleitete Klone (ergänzende Abb. 12f)65. Anschließend haben wir Tcf7l1 vor oder zu Beginn der Umwandlung durch Zugabe von Auxin abgereichert (ergänzende Abbildung). 12 g). Während der Abbau von Tcf7l1 vor der Induktion nur einen geringen Einfluss auf die Bildung von iN hatte, verringerte der Abbau von Tcf7l1 zu Beginn der Ascl1-Induktion die Anzahl der von Ascl1 produzierten Neuronen erheblich (ergänzende Abbildung). 12 g). Dies weist darauf hin, dass Tcf7l1 nach der Induktion wirkt und nicht das PPN stabilisiert.
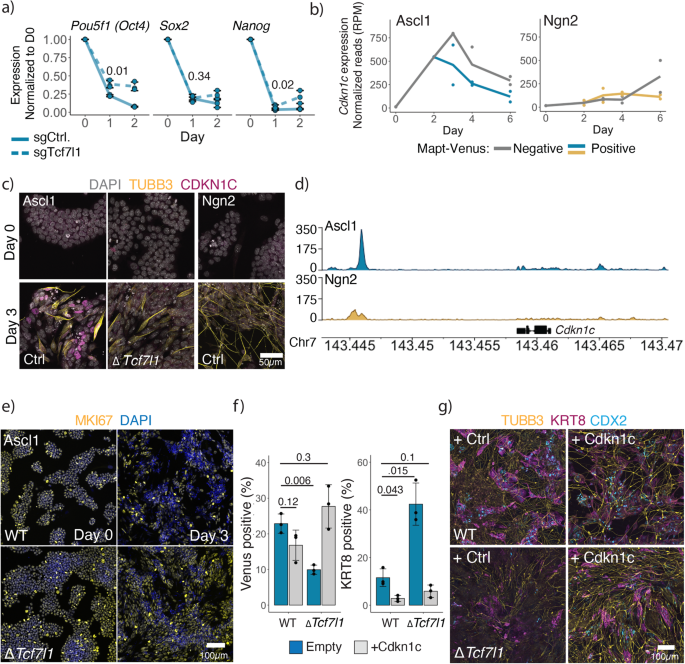
Um die Rolle von Tcf7l1 bei der Ascl1-induzierten ESC-zu-iN-gerichteten Differenzierung zu verstehen, haben wir von Einzelzellen abgeleitete Klone mit einem homozygoten Gen generiert Tcf7l1 Knockout und führte Massen-RNAseq am Tag 1 nach der Induktion durch (ergänzende Abb. 13a). Da Tcf7l1 während der ESC-Differenzierung als Pluripotenznetzwerk-Repressor fungiert, haben wir uns zunächst mit der Abschaltung der Pluripotenz befasst Tcf7l1 Knockout-Zellen. Überraschenderweise war das PPN nach der Ascl1-Induktion in Abwesenheit von Tcf7l1 immer noch herunterreguliert (Abb. 4a; Ergänzende Abb. 13a). Wir beobachteten jedoch eine Gruppe von Genen, die nicht exprimiert wurden ΔTcf7l1 Klone (ergänzende Abb. 13a). Interessant, Cdkn1c ist nach Ascl1-Induktion im Vergleich zu stark hochreguliert ΔTcf7l1 (Ergänzende Abb. 13a), und seine Expression ist spezifisch für die Ascl1-Induktion und erreicht ihren Höhepunkt am dritten Tag der Umwandlung von ESC in iN (Abb. 4b, c; Ergänzende Abb. 13b). Darüber hinaus zeigt Ascl1 eine starke Bindung in der Nähe Cdkn1c Locus im Vergleich zu Ngn2 (Abb. 4d).
a qPCR der Kernpluripotenz-Genexpression nach Induktion in WT und Tcf7l1 KO-Zellen. Expression normalisiert auf Actin und Tag 0. Es werden Linien durch den Mittelwert von gezogen n = 3 biologisch unabhängige Proben; Fehlerbalken zeigen ± SD an. p-Werte des zweiseitigen Welch-T-Tests mit zwei Stichproben, der sgTcf1l1 mit sgControl am oben angegebenen Tag 1 vergleicht. b Cdkn1c Ausdruck während der ESC-zu-iN-Konvertierung (Abb. 1a). c Immunfärbungen von CDKN1C am Tag 0 und Tag 3 der ESC-zu-iN-Umwandlung durch Ascl1 WT oder Tcf7l1 KO- oder Ngn2-exprimierende Zellen. d Bindung des Flag-Ascl1 oder Flag-Ngn2 im Cdkn1c Locus am Tag 1 nach der Induktion. Daten, die kombinierte Lesevorgänge von vier Replikaten zeigen. e Immunfärbungen des Proliferationsmarkers MKI67 nach Induktion von Ascl1 in WT oder Tcf7l1 KO ESC-Zellen. f FACS-Daten der Cdkn1c-Überexpression mit Ascl1 in WT oder Tcf7l1 KO ESCs. Die Effizienz der iN-Bildung wird anhand des Prozentsatzes der Mapt-Venus-Population gemessen, die Effizienz der iT-Bildung wird anhand des Prozentsatzes der auf KRT8 immungefärbten Zellen gemessen. Das Balkendiagramm zeigt den Mittelwert von n = 3 unabhängige biologische Replikate mit ± SD. p-Wert des oben angegebenen zweiseitigen Welch-T-Tests bei zwei Stichproben. g Repräsentative Bilder von immungefärbten Zellen für neuronale TUBB3- und Trophoblasten-CDX2/KRT8-Marker am Tag 6 nach der Induktion von WT oder Tcf7l1 KO-ESCs mit Ascl1 und Cdkn1c. Quelldaten werden als Quelldatendatei bereitgestellt.
Die durch Ascl1 gesteuerte Differenzierung ist zellzyklusabhängig
Cdkn1c ist ein Cyclin-abhängiger Kinase-Inhibitor der Cip/Kip-Familie, der den Stillstand des Zellzyklus in G1 reguliert66. Cdkn1c ist für die Embryonalentwicklung essentiell, und Mäuse, denen Cdkn1c fehlt, sterben perinatal mit mehreren Entwicklungsstörungen. Darüber hinaus ist Cdkn1c wichtig für die Entwicklung der frühen Plazenta, um die Endoreplikation von Trophoblasten einzuleiten66. Kürzlich wurde Cdkn1c auch an der Unterdrückung der Pluripotenz bei Mäuse-ESC beteiligt67. Wir sehen außerdem, dass Ascl1-induzierte GRN-Zentralknoten mehrere Gene enthalten, die an der Regulierung des Zellzyklus beteiligt sind, z. B. Cdkn1a, Cdkn1b, Cebpa, Cebpb (Ergänzende Abb. 8d, g). Daher haben wir die Hypothese aufgestellt, dass in ΔTcf7l1 Zellen ist Ascl1 nicht in der Lage, den Zellzyklus anzuhalten. Tatsächlich beobachten wir eine verlängerte Expression von MKI67 sowie einen höheren Prozentsatz sich teilender Zellen nach der Induktion von Ascl1 im ΔTcf7l1 Zellen (Abb. 4e, Ergänzende Abb. 13c). Um zu testen, ob die Expression von Cdkn1c ausreicht, um die iN-Bildung in Abwesenheit von Tcf7l1 zu induzieren, haben wir Cdkn1c zusammen mit Ascl1 coexprimiert. Tatsächlich sehen wir eine teilweise Wiederherstellung der iN-Bildung sowie einen Rückgang der iT-Erzeugung (Abb. 4f, g). Darüber hinaus sehen wir, dass die Expression von Cdkn1c auch in WT-Populationen bis zum 6. Tag einen reiferen neuronalen und Trophoblasten-Phänotyp erzeugt (Abb. 4g). Bemerkenswert ist, dass Cdkn1c für die Bildung von Ascl1-induzierten iNs nicht wesentlich ist, was darauf hindeutet, dass eine Gruppe von Zellzyklusregulatoren und nicht Cdkn1c allein für den Stillstand des Zellzyklus verantwortlich ist (ergänzende Abb. 13d).
Zusammenfassend zeigen unsere Daten, dass bei der Ascl1-induzierten Umwandlung von ESC in iN der Stillstand des Zellzyklus ein Hindernis nach dem Verlassen des Pluripotenzzustands darstellt und dass Cdkn1c ausreicht, um dieses Hindernis zu überwinden. Im Gegensatz dazu wird diese Abhängigkeit bei der Ngn2-abhängigen iN-Bildung nicht beobachtet, wo der Zellzyklus aufrechterhalten wird und einige ESCs zu NSCs übergehen.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Automobil / Elektrofahrzeuge, Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- ChartPrime. Verbessern Sie Ihr Handelsspiel mit ChartPrime. Hier zugreifen.
- BlockOffsets. Modernisierung des Eigentums an Umweltkompensationen. Hier zugreifen.
- Quelle: https://www.nature.com/articles/s41467-023-40803-y