17 de enero de 2024
(Noticias de Nanowerk) Investigadores del Nano Life Science Institute (WPI-NanoLSI) de la Universidad de Kanazawa informan en Nano Letters (“Dinámica estructural del dominio HECT de la ligasa E6AP E3 y participación del bucle de bisagra flexible en el mecanismo de síntesis de la cadena de ubiquitina”) cómo la flexibilidad de una bisagra proteica desempeña un papel crucial en la transferencia de proteínas en procesos celulares clave.
Imagen llamativa: observación y simulación en tiempo real de cambios conformacionales del dominio E6AP HECT.
El dominio HECT repite conformaciones esféricas y elípticas a través de la región flexible. Los resultados de la simulación por computadora muestran que la forma esférica corresponde a la estructura catalítica y la forma ovalada corresponde a la estructura en forma de L. La eficiencia de formación del dímero de ubiquitina (Ub2) cambia significativamente dependiendo del estado conformacional de la región de bisagra flexible. La ubiquitinación (la adición de la proteína ubiquitina) es una etapa clave en muchos procesos celulares, como la degradación de proteínas, la reparación del ADN y la transducción de señales. Utilizando microscopía de fuerza atómica de alta velocidad (HS-AFM) y modelos moleculares, investigadores dirigidos por Hiroki Konno y Holger Flechsig en WPI-NanoLSI, Universidad de Kanazawa, han identificado cómo la movilidad de una bisagra enzimática relacionada con la ubiquitinación permite que se produzca la ubiquitinación. Estudios anteriores han identificado una serie de enzimas que facilitan la ubiquitinación, incluida una enzima que activa la ubiquitina (E1), una enzima que la conjuga (E2) y una enzima que cataliza la unión de la proteína ubiquitina (es decir, una ligasa, E3) a la proteína objetivo ( Figura 1).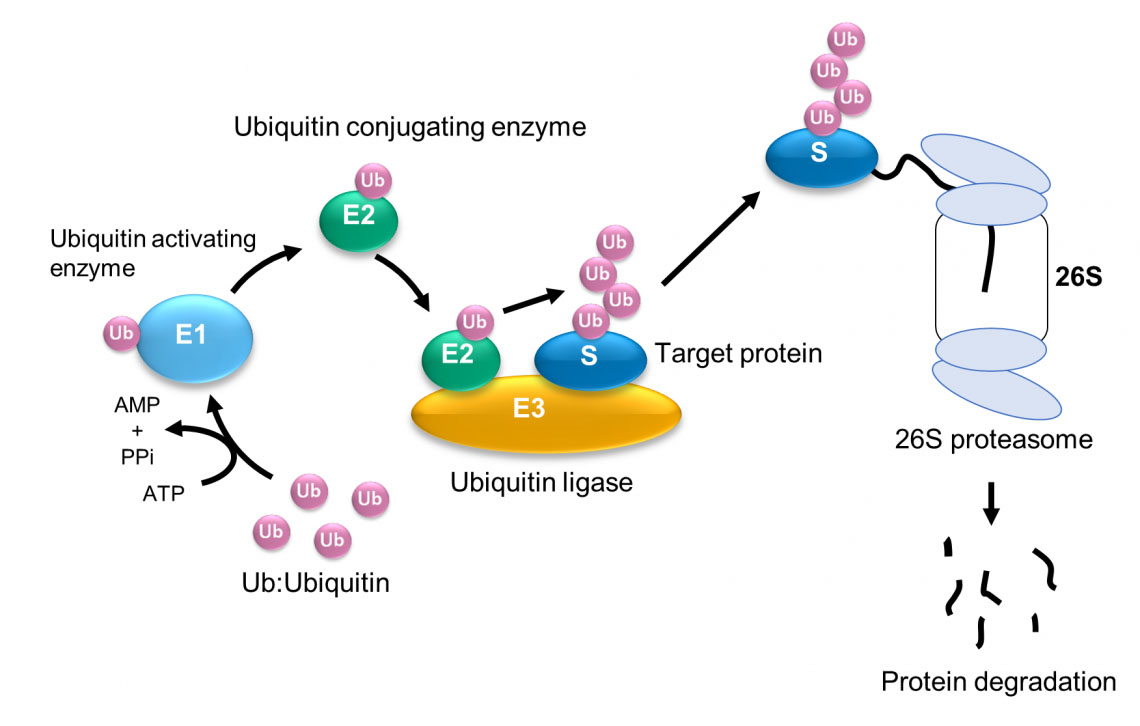 Fig. 1: Ubiquitinación y degradación de proteínas por el sistema ubiquitina-proteosoma. (© 2023 Takeda)
La ligasa E3 de tipo HECT se caracteriza por un dominio HECT que comprende un lóbulo N con el sitio de unión E2 y un lóbulo C con un residuo catalítico Cys. Una bisagra flexible conecta los dos lóbulos, lo que lleva a la hipótesis de que la ubiquitinación se ve facilitada por la reorganización de la proteína alrededor de esta bisagra. Konno y sus colaboradores desplegaron su microscopio de fuerza atómica de alta velocidad para buscar evidencia de que este fuera el caso.
Los investigadores observaron que cuando el dominio HECT se cristalizó con un tipo de enzima E2, formó una forma de L tal que la distancia entre el residuo catalítico Cys del dominio HECT y el Cys catalítico de la enzima E2 era de 41 Å, demasiado lejos para la transferencia de ubiquitina. Sin embargo, en su conformación catalítica, el dominio HECT tiene una forma diferente donde la distancia entre los dos residuos catalíticos de Cys es mucho más cercana (solo 8 Å), por lo que se cree que se trata de una "conformación catalítica".
El análisis de imágenes HS-AFM de un dominio HECT de tipo salvaje de E6AP reveló dos conformaciones, una de las cuales parecía esférica y la otra ovalada (Fig. 2). Utilizando simulaciones AFM, atribuyeron las formas ovaladas a la conformación L y las formas esféricas son la conformación catalítica o la llamada conformación T invertida, que se había observado en otro tipo de dominio HECT donde la distancia entre los residuos Cys es de 16 Å ( Figura 2). Para superar las limitaciones de resolución espacio-temporal de las imágenes, los experimentos se complementaron con modelos moleculares para visualizar los movimientos conformacionales del dominio HECT a nivel atomístico. Se utilizó AFM de simulación para generar una pseudo película AFM correspondiente, que mostraba claramente el cambio de la topografía esférica a la ovalada (Fig. 2).
Fig. 1: Ubiquitinación y degradación de proteínas por el sistema ubiquitina-proteosoma. (© 2023 Takeda)
La ligasa E3 de tipo HECT se caracteriza por un dominio HECT que comprende un lóbulo N con el sitio de unión E2 y un lóbulo C con un residuo catalítico Cys. Una bisagra flexible conecta los dos lóbulos, lo que lleva a la hipótesis de que la ubiquitinación se ve facilitada por la reorganización de la proteína alrededor de esta bisagra. Konno y sus colaboradores desplegaron su microscopio de fuerza atómica de alta velocidad para buscar evidencia de que este fuera el caso.
Los investigadores observaron que cuando el dominio HECT se cristalizó con un tipo de enzima E2, formó una forma de L tal que la distancia entre el residuo catalítico Cys del dominio HECT y el Cys catalítico de la enzima E2 era de 41 Å, demasiado lejos para la transferencia de ubiquitina. Sin embargo, en su conformación catalítica, el dominio HECT tiene una forma diferente donde la distancia entre los dos residuos catalíticos de Cys es mucho más cercana (solo 8 Å), por lo que se cree que se trata de una "conformación catalítica".
El análisis de imágenes HS-AFM de un dominio HECT de tipo salvaje de E6AP reveló dos conformaciones, una de las cuales parecía esférica y la otra ovalada (Fig. 2). Utilizando simulaciones AFM, atribuyeron las formas ovaladas a la conformación L y las formas esféricas son la conformación catalítica o la llamada conformación T invertida, que se había observado en otro tipo de dominio HECT donde la distancia entre los residuos Cys es de 16 Å ( Figura 2). Para superar las limitaciones de resolución espacio-temporal de las imágenes, los experimentos se complementaron con modelos moleculares para visualizar los movimientos conformacionales del dominio HECT a nivel atomístico. Se utilizó AFM de simulación para generar una pseudo película AFM correspondiente, que mostraba claramente el cambio de la topografía esférica a la ovalada (Fig. 2).
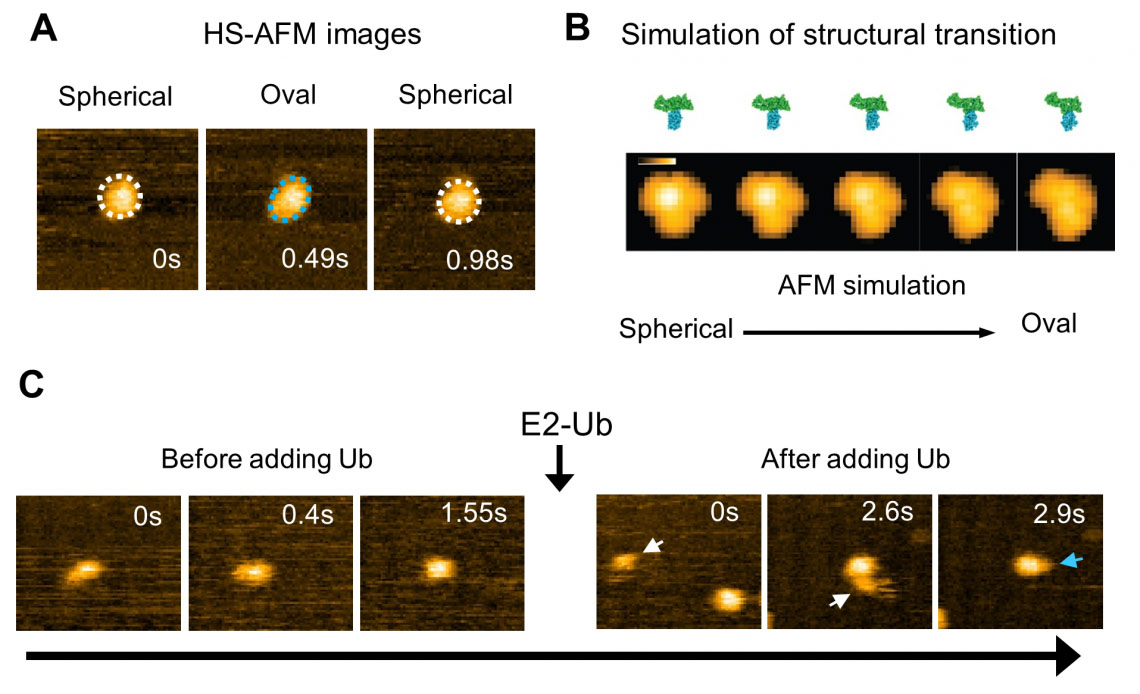 Fig. 2: Observación AFM de alta velocidad y simulación de la dinámica estructural del dominio HECT de E6AP. (A) Imagen HS-AFM del dominio HECT. Se observaron estados conformacionales esféricos (línea punteada blanca) y ovalados (línea punteada azul). (B) Proceso de transición simulado del estado estructural del dominio HECT y su pseudo imagen AFM. (C) La ubiquitina (Ub) se transfiere de E2 a E3 (dominio HECT). Después de la adición de E2 que contiene ubiquitina (E2-Ub), E2-Ub (flecha blanca) se une al dominio HECT y después de que E2 se disocia, se agrega una pequeña partícula (flecha azul) que parece ser ubiquitina al dominio HECT. . (© 2023 Takeda)
“Aunque las limitaciones experimentales no nos permiten resolver las conformaciones intermedias”, explican los investigadores en su informe del trabajo. “El modelado realizado proporciona evidencia de que las transiciones entre las formas de dominio HECT esféricas y ovaladas observadas bajo HS-AFM corresponden a movimientos conformacionales funcionales bajo los cuales el lóbulo C gira en relación con el lóbulo N, permitiendo así el cambio entre la forma catalítica y la forma L. conformaciones HECT”.
Experimentos adicionales con dominios HECT mutantes con menos flexibilidad en la bisagra no revelaron cambios entre conformaciones: los dominios HECT mutantes estaban bloqueados en la conformación catalítica. También descubrieron que, si bien estos dominios HECT mutantes podían formar dos proteínas de ubiquitina, se unían de manera más eficiente que el tipo salvaje.
E6AP, el E3 de tipo HECT en este estudio, interactúa con la proteína E6 derivada del virus del papiloma humano (VPH) y ubiquitina p53, una proteína supresora de tumores. También se sabe que la ubiquitinación de p53 por E6AP y E6 es una causa importante de cáncer de cuello uterino. Sin embargo, el mecanismo de ubiquitinación de p53 mediante la interacción de las proteínas E6AP y E6 aún no está claro. En el futuro, dilucidaremos la dinámica estructural de E6AP/E6 y el complejo E6AP/E6/p53 con HS-AFM, y aclararemos cómo E6 aumenta la actividad de ubiquitinación de p53 por parte de E6AP.
Fig. 2: Observación AFM de alta velocidad y simulación de la dinámica estructural del dominio HECT de E6AP. (A) Imagen HS-AFM del dominio HECT. Se observaron estados conformacionales esféricos (línea punteada blanca) y ovalados (línea punteada azul). (B) Proceso de transición simulado del estado estructural del dominio HECT y su pseudo imagen AFM. (C) La ubiquitina (Ub) se transfiere de E2 a E3 (dominio HECT). Después de la adición de E2 que contiene ubiquitina (E2-Ub), E2-Ub (flecha blanca) se une al dominio HECT y después de que E2 se disocia, se agrega una pequeña partícula (flecha azul) que parece ser ubiquitina al dominio HECT. . (© 2023 Takeda)
“Aunque las limitaciones experimentales no nos permiten resolver las conformaciones intermedias”, explican los investigadores en su informe del trabajo. “El modelado realizado proporciona evidencia de que las transiciones entre las formas de dominio HECT esféricas y ovaladas observadas bajo HS-AFM corresponden a movimientos conformacionales funcionales bajo los cuales el lóbulo C gira en relación con el lóbulo N, permitiendo así el cambio entre la forma catalítica y la forma L. conformaciones HECT”.
Experimentos adicionales con dominios HECT mutantes con menos flexibilidad en la bisagra no revelaron cambios entre conformaciones: los dominios HECT mutantes estaban bloqueados en la conformación catalítica. También descubrieron que, si bien estos dominios HECT mutantes podían formar dos proteínas de ubiquitina, se unían de manera más eficiente que el tipo salvaje.
E6AP, el E3 de tipo HECT en este estudio, interactúa con la proteína E6 derivada del virus del papiloma humano (VPH) y ubiquitina p53, una proteína supresora de tumores. También se sabe que la ubiquitinación de p53 por E6AP y E6 es una causa importante de cáncer de cuello uterino. Sin embargo, el mecanismo de ubiquitinación de p53 mediante la interacción de las proteínas E6AP y E6 aún no está claro. En el futuro, dilucidaremos la dinámica estructural de E6AP/E6 y el complejo E6AP/E6/p53 con HS-AFM, y aclararemos cómo E6 aumenta la actividad de ubiquitinación de p53 por parte de E6AP.
El dominio HECT repite conformaciones esféricas y elípticas a través de la región flexible. Los resultados de la simulación por computadora muestran que la forma esférica corresponde a la estructura catalítica y la forma ovalada corresponde a la estructura en forma de L. La eficiencia de formación del dímero de ubiquitina (Ub2) cambia significativamente dependiendo del estado conformacional de la región de bisagra flexible. La ubiquitinación (la adición de la proteína ubiquitina) es una etapa clave en muchos procesos celulares, como la degradación de proteínas, la reparación del ADN y la transducción de señales. Utilizando microscopía de fuerza atómica de alta velocidad (HS-AFM) y modelos moleculares, investigadores dirigidos por Hiroki Konno y Holger Flechsig en WPI-NanoLSI, Universidad de Kanazawa, han identificado cómo la movilidad de una bisagra enzimática relacionada con la ubiquitinación permite que se produzca la ubiquitinación. Estudios anteriores han identificado una serie de enzimas que facilitan la ubiquitinación, incluida una enzima que activa la ubiquitina (E1), una enzima que la conjuga (E2) y una enzima que cataliza la unión de la proteína ubiquitina (es decir, una ligasa, E3) a la proteína objetivo ( Figura 1).
 Fig. 1: Ubiquitinación y degradación de proteínas por el sistema ubiquitina-proteosoma. (© 2023 Takeda)
La ligasa E3 de tipo HECT se caracteriza por un dominio HECT que comprende un lóbulo N con el sitio de unión E2 y un lóbulo C con un residuo catalítico Cys. Una bisagra flexible conecta los dos lóbulos, lo que lleva a la hipótesis de que la ubiquitinación se ve facilitada por la reorganización de la proteína alrededor de esta bisagra. Konno y sus colaboradores desplegaron su microscopio de fuerza atómica de alta velocidad para buscar evidencia de que este fuera el caso.
Los investigadores observaron que cuando el dominio HECT se cristalizó con un tipo de enzima E2, formó una forma de L tal que la distancia entre el residuo catalítico Cys del dominio HECT y el Cys catalítico de la enzima E2 era de 41 Å, demasiado lejos para la transferencia de ubiquitina. Sin embargo, en su conformación catalítica, el dominio HECT tiene una forma diferente donde la distancia entre los dos residuos catalíticos de Cys es mucho más cercana (solo 8 Å), por lo que se cree que se trata de una "conformación catalítica".
El análisis de imágenes HS-AFM de un dominio HECT de tipo salvaje de E6AP reveló dos conformaciones, una de las cuales parecía esférica y la otra ovalada (Fig. 2). Utilizando simulaciones AFM, atribuyeron las formas ovaladas a la conformación L y las formas esféricas son la conformación catalítica o la llamada conformación T invertida, que se había observado en otro tipo de dominio HECT donde la distancia entre los residuos Cys es de 16 Å ( Figura 2). Para superar las limitaciones de resolución espacio-temporal de las imágenes, los experimentos se complementaron con modelos moleculares para visualizar los movimientos conformacionales del dominio HECT a nivel atomístico. Se utilizó AFM de simulación para generar una pseudo película AFM correspondiente, que mostraba claramente el cambio de la topografía esférica a la ovalada (Fig. 2).
Fig. 1: Ubiquitinación y degradación de proteínas por el sistema ubiquitina-proteosoma. (© 2023 Takeda)
La ligasa E3 de tipo HECT se caracteriza por un dominio HECT que comprende un lóbulo N con el sitio de unión E2 y un lóbulo C con un residuo catalítico Cys. Una bisagra flexible conecta los dos lóbulos, lo que lleva a la hipótesis de que la ubiquitinación se ve facilitada por la reorganización de la proteína alrededor de esta bisagra. Konno y sus colaboradores desplegaron su microscopio de fuerza atómica de alta velocidad para buscar evidencia de que este fuera el caso.
Los investigadores observaron que cuando el dominio HECT se cristalizó con un tipo de enzima E2, formó una forma de L tal que la distancia entre el residuo catalítico Cys del dominio HECT y el Cys catalítico de la enzima E2 era de 41 Å, demasiado lejos para la transferencia de ubiquitina. Sin embargo, en su conformación catalítica, el dominio HECT tiene una forma diferente donde la distancia entre los dos residuos catalíticos de Cys es mucho más cercana (solo 8 Å), por lo que se cree que se trata de una "conformación catalítica".
El análisis de imágenes HS-AFM de un dominio HECT de tipo salvaje de E6AP reveló dos conformaciones, una de las cuales parecía esférica y la otra ovalada (Fig. 2). Utilizando simulaciones AFM, atribuyeron las formas ovaladas a la conformación L y las formas esféricas son la conformación catalítica o la llamada conformación T invertida, que se había observado en otro tipo de dominio HECT donde la distancia entre los residuos Cys es de 16 Å ( Figura 2). Para superar las limitaciones de resolución espacio-temporal de las imágenes, los experimentos se complementaron con modelos moleculares para visualizar los movimientos conformacionales del dominio HECT a nivel atomístico. Se utilizó AFM de simulación para generar una pseudo película AFM correspondiente, que mostraba claramente el cambio de la topografía esférica a la ovalada (Fig. 2).
 Fig. 2: Observación AFM de alta velocidad y simulación de la dinámica estructural del dominio HECT de E6AP. (A) Imagen HS-AFM del dominio HECT. Se observaron estados conformacionales esféricos (línea punteada blanca) y ovalados (línea punteada azul). (B) Proceso de transición simulado del estado estructural del dominio HECT y su pseudo imagen AFM. (C) La ubiquitina (Ub) se transfiere de E2 a E3 (dominio HECT). Después de la adición de E2 que contiene ubiquitina (E2-Ub), E2-Ub (flecha blanca) se une al dominio HECT y después de que E2 se disocia, se agrega una pequeña partícula (flecha azul) que parece ser ubiquitina al dominio HECT. . (© 2023 Takeda)
“Aunque las limitaciones experimentales no nos permiten resolver las conformaciones intermedias”, explican los investigadores en su informe del trabajo. “El modelado realizado proporciona evidencia de que las transiciones entre las formas de dominio HECT esféricas y ovaladas observadas bajo HS-AFM corresponden a movimientos conformacionales funcionales bajo los cuales el lóbulo C gira en relación con el lóbulo N, permitiendo así el cambio entre la forma catalítica y la forma L. conformaciones HECT”.
Experimentos adicionales con dominios HECT mutantes con menos flexibilidad en la bisagra no revelaron cambios entre conformaciones: los dominios HECT mutantes estaban bloqueados en la conformación catalítica. También descubrieron que, si bien estos dominios HECT mutantes podían formar dos proteínas de ubiquitina, se unían de manera más eficiente que el tipo salvaje.
E6AP, el E3 de tipo HECT en este estudio, interactúa con la proteína E6 derivada del virus del papiloma humano (VPH) y ubiquitina p53, una proteína supresora de tumores. También se sabe que la ubiquitinación de p53 por E6AP y E6 es una causa importante de cáncer de cuello uterino. Sin embargo, el mecanismo de ubiquitinación de p53 mediante la interacción de las proteínas E6AP y E6 aún no está claro. En el futuro, dilucidaremos la dinámica estructural de E6AP/E6 y el complejo E6AP/E6/p53 con HS-AFM, y aclararemos cómo E6 aumenta la actividad de ubiquitinación de p53 por parte de E6AP.
Fig. 2: Observación AFM de alta velocidad y simulación de la dinámica estructural del dominio HECT de E6AP. (A) Imagen HS-AFM del dominio HECT. Se observaron estados conformacionales esféricos (línea punteada blanca) y ovalados (línea punteada azul). (B) Proceso de transición simulado del estado estructural del dominio HECT y su pseudo imagen AFM. (C) La ubiquitina (Ub) se transfiere de E2 a E3 (dominio HECT). Después de la adición de E2 que contiene ubiquitina (E2-Ub), E2-Ub (flecha blanca) se une al dominio HECT y después de que E2 se disocia, se agrega una pequeña partícula (flecha azul) que parece ser ubiquitina al dominio HECT. . (© 2023 Takeda)
“Aunque las limitaciones experimentales no nos permiten resolver las conformaciones intermedias”, explican los investigadores en su informe del trabajo. “El modelado realizado proporciona evidencia de que las transiciones entre las formas de dominio HECT esféricas y ovaladas observadas bajo HS-AFM corresponden a movimientos conformacionales funcionales bajo los cuales el lóbulo C gira en relación con el lóbulo N, permitiendo así el cambio entre la forma catalítica y la forma L. conformaciones HECT”.
Experimentos adicionales con dominios HECT mutantes con menos flexibilidad en la bisagra no revelaron cambios entre conformaciones: los dominios HECT mutantes estaban bloqueados en la conformación catalítica. También descubrieron que, si bien estos dominios HECT mutantes podían formar dos proteínas de ubiquitina, se unían de manera más eficiente que el tipo salvaje.
E6AP, el E3 de tipo HECT en este estudio, interactúa con la proteína E6 derivada del virus del papiloma humano (VPH) y ubiquitina p53, una proteína supresora de tumores. También se sabe que la ubiquitinación de p53 por E6AP y E6 es una causa importante de cáncer de cuello uterino. Sin embargo, el mecanismo de ubiquitinación de p53 mediante la interacción de las proteínas E6AP y E6 aún no está claro. En el futuro, dilucidaremos la dinámica estructural de E6AP/E6 y el complejo E6AP/E6/p53 con HS-AFM, y aclararemos cómo E6 aumenta la actividad de ubiquitinación de p53 por parte de E6AP.
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- PlatoData.Network Vertical Generativo Ai. Empodérate. Accede Aquí.
- PlatoAiStream. Inteligencia Web3. Conocimiento amplificado. Accede Aquí.
- PlatoESG. Carbón, tecnología limpia, Energía, Ambiente, Solar, Gestión de residuos. Accede Aquí.
- PlatoSalud. Inteligencia en Biotecnología y Ensayos Clínicos. Accede Aquí.
- Fuente: https://www.nanowerk.com/news2/biotech/newsid=64429.php



