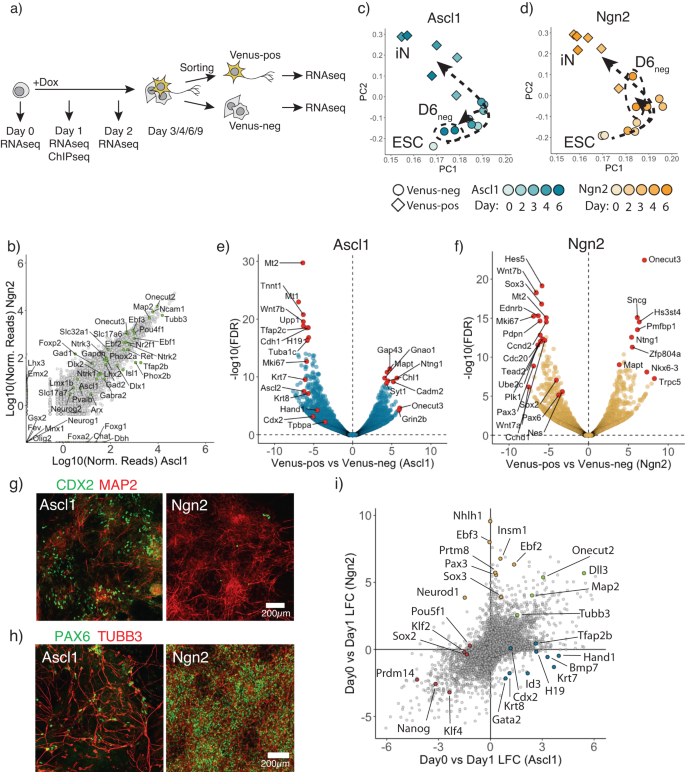
Ascl1 y Ngn2 convierten ESC a iN pero generan linajes laterales diferentes
La expresión ectópica de Ascl1 o Ngn2 en células madre embrionarias de ratón (ESC) es suficiente para inducir la diferenciación terminal en neuronas.28. Sin embargo, las diferencias en los mecanismos de transición hacia las neuronas, así como las posibles poblaciones laterales, no están bien caracterizadas.6,31. Para examinar esta conversión de tipo celular en detalle, generamos líneas celulares clonales ESC que expresan rtTA y TetO-Ascl1 o TetO-Ngn2.27,28 (Fig. complementaria. 1a). Después de la adición de doxiciclina (Dox), las ESC se convierten rápidamente en neuronas inducidas: Ascl1 y Ngn2 producen células que expresan el marcador neuronal TUBB3 y muestran morfología neuronal desde el día 3 y el día 2 en adelante, respectivamente (Fig. 1a). Para informar el destino neuronal en estas líneas celulares, etiquetamos endógenamente el gen marcador panneuronal Mapt en su extremo C con la proteína fluorescente Venus.27 (Fig. complementaria. 1b) y realizó RNAseq masivo resuelto en el tiempo tras la inducción de Dox. Las células se clasificaron en neuronas positivas para Venus y poblaciones de células negativas para Venus (Fig. 1a) a partir del día 3 en adelante. Como se informó antes28,30, tanto Ascl1 como Ngn2 dan lugar a identidades de células iN similares (Fig. 1b; Figura complementaria. 1c-e). Por lo tanto, los estados inicial de ESC y terminal iN son muy similares entre las conversiones inducidas por Ascl1 y Ngn2 (Figura complementaria). 1b abajo). Esto está en línea con observaciones previas de que los transcriptomas convergen para impulsar la formación de iN a pesar de las diferencias en la respuesta transcripcional inicial.6 y sigue una trayectoria general similar entre ESC e iN en el análisis de PCA (Fig. 1c, d; Figura complementaria. 1g).
a Descripción esquemática del diseño experimental. b Diagrama de dispersión que compara la expresión génica en el día 6 entre células Ascl1 y Ngn2 Venus positivas con varios marcadores específicos de subtipo neuronal indicados en verde. c, d Análisis de componentes principales de RNAseq masivo resuelto en el tiempo después de Ascl1 (c) o Ngn2 (d). Cada punto de datos corresponde a la réplica del único punto de tiempo. La intensidad del color muestra el día posterior a la inducción. La forma corresponde a la regulación positiva del reportero Mapt-Venus. Las flechas muestran la trayectoria que toman las células después de Ascl1 (c) o inducción de Ngn2 (d). e, f Gráfico de Vulcano que compara la expresión genética entre poblaciones positivas y negativas de Venus en el día 6 después de la inducción de Ascl1 (e) o Ngn2 (f). Los círculos rojos indican los principales genes significativamente regulados hacia arriba o hacia abajo, así como genes de ejemplo marcados en el trofoblasto (e) o linajes NSC (f). g Células inmunoteñidas representativas para un marcador de trofoblasto CDX2 y un marcador neuronal Map2 en el día 6 después de la inducción de Ascl1 o Ngn2. Los marcadores de trofoblasto se expresaron sólo después de la inducción de Ascl1, pero no de Ngn2. h Células inmunoteñidas representativas para un marcador NSC PAX6 y un marcador neuronal TUBB3 en el día 6 después de la inducción de Ascl1 o Ngn2. Los marcadores NSC se expresaron solo después de la expresión de Ngn2, pero no de Ascl1. i Diagrama de dispersión que compara los cambios en la expresión génica entre Ascl1 y Ngn2 el día 1 después de la inducción. Los círculos resaltados son genes de ejemplo que son marcadores neuronales expresados en ambos (verde), marcadores específicos de trofoblasto Ascl1 (azul), marcadores específicos de NSC Ngn2 (amarillo), genes relacionados con la pluripotencia (rojo). Los datos de origen se proporcionan como un archivo de datos de origen.
Para investigar con más detalle las células que no producen iN, nos centramos en las células negativas para Mapt-Venus, que podrían representar resultados de diferenciación incompletos o alternativos. Las células Mapt-Venus negativas generadas por Ascl1 se agrupan más cerca de las poblaciones iniciales de ESC en los gráficos de PCA que las células Mapt negativas inducidas por Ngn2 (Fig. 1c). Sin embargo, esto no se debe a la retención de una población de ESC indiferenciadas, ya que solo un número marginal de células expresan el marcador ESC NANOG en la población terminal (Fig. 1g), y NANOG y OCT4 no se expresan en la población Mapt-Venus (Fig. 1h). Para identificar el tipo alternativo de células generadas por Ascl1, utilizamos PanglaoDB32 utilizando genes expresados diferencialmente entre poblaciones positivas y negativas de Venus en el día 6 (Fig. 1e,f; Figura complementaria. 2a). Curiosamente, Ascl1 produce células que expresan marcadores de trofoblasto como Mano1, cdx2, tpbpa, krt8 (Higo. 1e, g; Figura complementaria. 2b-g) con morfología mesenquimatosa, que no estaban presentes en los cultivos inducidos por Ngn2 (Fig. 1g, Fig. Suplementaria 2b, c, f). A estas células las denominamos células similares al trofoblasto inducidas (iT). Curiosamente, muchos de los krt8, cdx2 Los iT positivos parecían binucleados, lo que podría ser el resultado de una multinucleación similar al desarrollo del linaje del trofoblasto in vivo (Fig. 2e)33. La inducción de iT podría deberse a que Ascl1 imita el factor de transcripción bHLH Ascl2, un controlador del linaje del trofoblasto. Tanto Ascl1 como Ascl2 son evolutivamente cercanos y comparten dominios de unión al ADN casi idénticos y se unen a motivos de caja E similares (Fig. 2h–k)33,34,35,36,37. Por último, para excluir los efectos clonales de la línea celular utilizada, repetimos estos experimentos en el fondo de una línea ESC de ratón alternativa, E14. Introdujimos rtTA a través de un vector transposón piggybac y expresamos Ascl1 a partir de un vector viral inducible por Dox.27 y generó 24 clones derivados de una sola célula. Todos los clones mostraron la formación tanto de iN como de iT, lo que sugiere que la formación de iTs es un producto secundario reproducible de la expresión ectópica de Ascl1 en ESC de ratón (Fig. 3).
A diferencia de la inducción de Ascl1, Ngn2 induce células Mapt negativas que expresan marcadores NSC, como medias2, pax3, pax6, Nes (Higo. 1f, h; Figura complementaria. 2a, 4a, b), como se describió previamente21,38. Además, la reprogramación de Ngn2 podría bloquearse en el estado similar a NSC (iNSC) en presencia de FGF2 y EGF y depende de la vía de Notch.39,40 (Fig. complementaria. 4c, d). Por el contrario, no observamos marcadores NSC regulados positivamente durante la diferenciación inducida por Ascl1 (Fig. 1h, Fig. Suplementaria 4b, c). Para ver si Ngn2 puede usar el estado de iNSC como intermediario proliferativo, diferenciamos las células en presencia o ausencia de citosina β-D-arabinofuranósido (AraC) desde el día 4 después de la inducción para inactivar las células en división (Fig. 4e). De hecho, la adición de AraC reduce drásticamente las iN producidas por Ngn2, mientras que Ascl1 fue insensible al tratamiento con AraC, lo que sugiere que no hay ningún intermediario proliferativo continuo presente durante la reprogramación de iN inducida por Ascl1 (Figura complementaria. 4e). En resumen, a pesar de que Ascl1 y Ngn2 convierten mESC en subtipos iN similares, Ascl1 y Ngn2 producen distintos linajes celulares alternativos adicionales, lo que sugiere que a pesar de poblaciones iniciales y terminales idénticas, existen diferencias que intentamos comprender mejor (Figura complementaria). 1b abajo).
Ascl1 y Ngn2 inician caminos con diferentes programas transcripcionales
Para comprender mejor las diferentes respuestas transcripcionales invocadas por Ascl1 y Ngn2, realizamos RNAseq y ChiPseq en masa el día 1 (Fig. 1a). Tanto Ascl1 como Ngn2 inducen marcadores neuronales generales como tubb3, Map2 y Onecut2 y regular a la baja los marcadores generales de pluripotencia como nanog, klf4 (Higo. 1i). Además, Ascl1 induce fuertemente objetivos posteriores. Tfap2b, Lmx1b, mientras que Ngn2 regula fuertemente neurod1, Nhlh1 (Higo. 1i). Además, Ascl1 regula positivamente los marcadores del linaje del trofoblasto, por ejemplo, Krt7/8, Mano1, mientras que Ngn2 regula positivamente la expresión de genes relacionados con NSC, como pax3 y medias3 (Higo. 1i). Curiosamente, observamos que en las primeras etapas de la reprogramación las células son positivas para ambos marcadores, neuronales y trofoblásticos (Fig. 5a, b). Además, volvimos a analizar los datos de scRNAseq disponibles.6 para el día 2 de la conversión de ESC a iN por Ascl1 y Ngn2 y también se pudieron observar células positivas para marcadores neuronales y de trofoblasto (Figura complementaria. 5c, d). Esto sugiere que Ascl1 puede inducir ambos linajes simultáneamente, que luego se resuelven en células iN o iT (Fig. 5b).
Según lo informado por Aydin y sus colegas6, Ascl1 y Ngn2 muestran diferentes preferencias por motivos de caja E, que a su vez resultan en la activación de diferentes subconjuntos de genes. De hecho, confirmamos la unión diferencial de Ascl1 y Ngn2 en ESC (Fig. 6a). Además, vemos que Ascl1 y Ngn2 se dirigen a genes expresados diferencialmente entre ellos, así como a genes implicados en los diferentes linajes alternativos, por ejemplo, Mano1, cdx2, krt8 or neurod1, pax3, respectivamente (Fig. 6b). Curiosamente, Ascl1 se une a genes relacionados con el trofoblasto también en MEF, aunque sin inducción de iT (Figura complementaria. 7a, b). Sin embargo, además de la inducción de iN, la sobreexpresión de Ascl1 en MEF conduce a la unión de Ascl1 a los genes del músculo esquelético y a la inducción de miocitos.7,16. De hecho, en ESC Ascl1 también se une fuertemente a genes relacionados con el linaje del músculo esquelético, por ejemplo, miod1, miog, mif3, tnt2 (Fig. complementaria. 7c). Sin embargo, no observamos una regulación positiva de estos genes (Figura complementaria. 7d). Por lo tanto, es tentador especular que el contexto celular, como las modificaciones de histonas específicas del tipo de célula o los factores de transcripción, afecta la elección del linaje alternativo inducido. Así, además de la inducción del programa transcripcional neuronal, se unen y transcriben genes adicionales, lo que lleva a la formación de linajes alternativos. Este programa transcripcional "fuera del objetivo" depende de la sobreexpresión del transgén, por ejemplo, Ascl1 o Ngn2, así como del contexto celular, por ejemplo, ESC o MEF.
Para investigar los principios generales de la respuesta transcripcional temprana con más profundidad, utilizamos la base de datos STRING para examinar la red reguladora de genes (GRN) de los genes expresados diferencialmente (DEG) regulados positivamente por Ascl1 y Ngn2 un día después de la inducción (Fig. 8a, b). Tanto los DEG Ascl1 como los Ngn2 forman redes que contienen tres grupos de genes distintos. Curiosamente, los GRN Ascl1 y Ngn2 comparten grupos que contienen genes implicados en el metabolismo del ARN y los esteroles, lo que puede atribuirse al cambio metabólico durante la conversión.41,42. Además, Ngn2 forma una red más interconectada que Ascl1, lo que sugiere que Ngn2 invoca una respuesta transcripcional más coherente que Ascl1 (Figura complementaria). 8c).
Para analizar las diferencias con más detalle, observamos los nodos centrales más conectados de ambos GRN (Figura complementaria). 8d, e). A diferencia de Ascl1, Ngn2 induce genes que impulsan la diferenciación neuronal neurog2, neurod1, neurog1, Lhx2/3, otx2, así como genes implicados en la diferenciación de células madre neurales: Notch1, hes5, pax3, centrando GRN alrededor de ellos (Fig. 8e-g). Curiosamente, se sabe que Ngn2, Neurod1 y Ngn2 pueden convertir ESC en iN.43. Por lo tanto, un circuito de retroalimentación positiva junto con la inducción de controladores de linaje fuertes y GRN altamente interconectados pueden permitir una conversión iN más robusta y una independencia más rápida de la inducción inicial del casete. Para probar esta hipótesis, indujimos células durante 2, 4 o 6 días (Fig. 8h). De hecho, observamos que la conversión eficiente de iN con Ascl1 depende de la expresión sostenida de Ascl1, mientras que Ngn2 induce eficientemente la conversión ya después de 2 días de inducción (Fig. 8h).
En resumen, Ascl1 y Ngn2 se unen e invocan diferentes perfiles transcripcionales y diferencias mecanísticas posteriores de la conversión de ESC a iN. Mientras que la inducción de Ascl1 se basa en la expresión sostenida de Ascl1, Ngn2 induce una red general más coherente que permite una reprogramación rápida y eficiente, así como la inducción de un intermediario proliferativo.
Pantalla CRISPR/Cas9 de pérdida de función para identificar dependencias genéticas para la conversión de ESC a iN
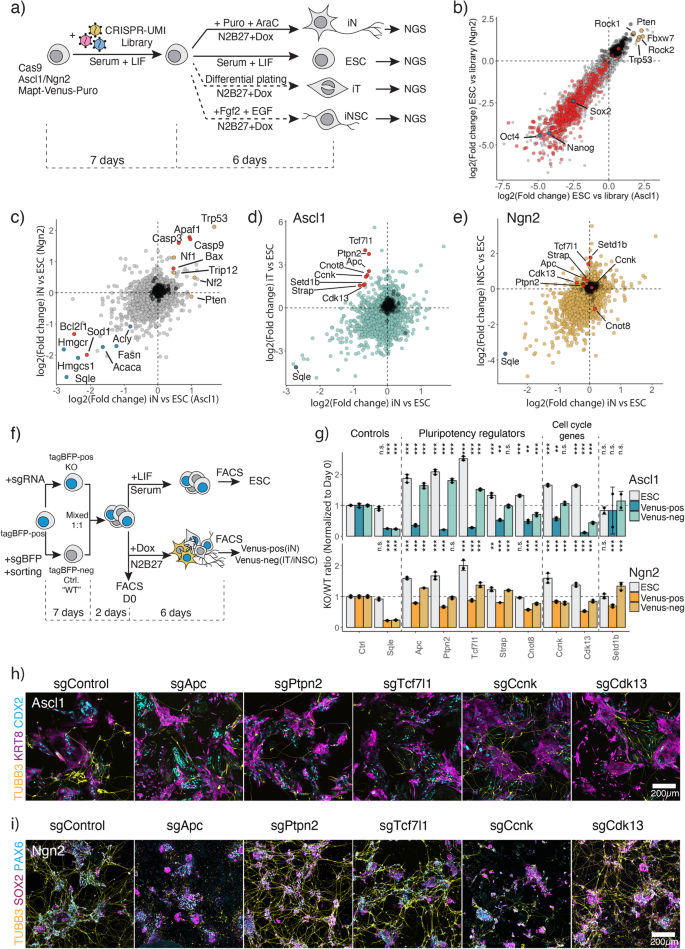
Para establecer un control mecanicista de las diferencias subyacentes entre la reprogramación de ESC inducida por Ascl1 y Ngn2, realizamos una evaluación de pérdida de función CRISPR/Cas9. Nuestro objetivo fue investigar dos aspectos de esta conversión: primero, las dependencias genéticas de la conversión neuronal inducida por Ascl1 y Ngn2, y segundo, dilucidar los genes involucrados en la formación de estados alternativos (Fig. 2a). Para esto, las ESC se infectaron con una biblioteca de sgRNA CRISPR-UMI retroviral que contiene ~27,000 6630 guías dirigidas a 4 genes (108 guías por gen) y XNUMX guías no dirigidas.44. Luego diferenciamos ESC durante 6 días y lo enriquecimos para iN usando AraC y tratamiento con puromicina (Fig. 2a, Fig. Suplementaria 1b, 9a). Además, evaluamos las dependencias para la diferenciación de iT e iNSC (Fig. 2a, ver “Métodos”). Para una evaluación inicial de los resultados de la evaluación, comparamos el agotamiento de los elementos esenciales conocidos en ESC versus la Biblioteca (Fig. 2b). De hecho, observamos un fuerte agotamiento en genes esenciales comunes según lo definido por Hart et al.45. Además, los genes de la red central de pluripotencia (nanog, medias2, pou5f1) se encuentran entre los genes más esenciales, mientras que los supresores de tumores, incluidos Trp53, Fbxw7, Rock1 son fuertemente enriquecedores en ESC (Fig. 2b), lo que demuestra que nuestra biblioteca es eficaz para apuntar a genes y revelar fenotipos de genes knockout.
a Esquema experimental de la pantalla CRISPR-Cas9. b Análisis comparativo de los efectos de eliminación de genes entre los transgenes Ascl1 y Ngn2 que portan ESC. La diferencia en la abundancia de guías se calculó entre ESC no inducidas el día 13 después de la infección de la biblioteca (a) frente al conjunto de plásmidos de la biblioteca. Los puntos representan genes; El eje muestra el agotamiento de LFC en cada línea celular. El rojo representa genes esenciales centrales según lo definido por Hart et al.45, azul: genes centrales de pluripotencia, amarillo: supresores de tumores. c Análisis comparativo de los efectos de eliminación de genes de iN inducida por Ascl1 o Ngn2 versus ESC. Los puntos rojos representan genes relacionados con la apoptosis, los amarillos, genes supresores de tumores, y los azules, genes de biosíntesis de colesterol. d Análisis comparativo de los efectos de eliminación de genes de iN o iT inducidos por Ascl1 versus ESC. Los puntos rojos representan genes elegidos para la validación. Sqle es un control positivo para un fuerte agotamiento. e Análisis comparativo de los efectos de eliminación de genes de iN o iNSC inducidos por Ngn2 versus ESC. Los puntos rojos representan genes elegidos para la validación en (d). Sqle es un control positivo para un fuerte agotamiento. f Esquema experimental de validación de visitas de (d). g Validación de los aciertos basada en FACS. La barra representa la proporción normalizada con respecto a la proporción de mezcla inicial en el día 0. N = 3 réplicas biológicas independientes. El gráfico de barras muestra la media ± DE. pLos valores indicados anteriormente se determinaron mediante ANOVA unidireccional seguido de la prueba de comparación múltiple de Dunnett (bilateral) utilizando la relación Ctrl como control. “ns” no significativo, “*” p < 0.05, “**” p < 0.01, “***” p <0.001. h, i Inmunotinción de las células knockout para el marcador neuronal Tubb3 y marcadores de linaje alternativos KRT8/CDX2 para iT inducida por Ascl1 (h), y SOX2/PAX6 para iNSC inducido por Ngn2 (i). Los datos de origen se proporcionan como un archivo de datos de origen.
Para identificar dependencias genéticas comunes de la formación de iN, comparamos la abundancia de guías en ESC versus iN en D6 (Fig. 2c). Las guías que muestran el mayor agotamiento se centran, por ejemplo, en el metabolismo de los ácidos grasos y la biosíntesis de esteroles, como Fasn, Hmgcr, Hmgcs1, cuadrado, un componente esencial para el metabolismo neuronal (Fig. 2c)41,42. Además, durante la diferenciación iN, las células sufren altos niveles de estrés debido a la peroxidación lipídica y pueden sufrir la muerte celular mediante apoptosis o ferroptosis.46,47. Por tanto, encontramos genes proapoptóticos, Casp3/9 y Bax enriquecedores, mientras que los genes antiapoptóticos como bcl2l1 y Césped1 se están agotando de la población (Fig. 2c; Figura complementaria. 9b). Además, la eliminación de supresores de tumores, por ejemplo, Trp53, Nf1, Nf2, permite una mayor conversión iN. Por lo tanto, esta pantalla produce dependencias genéticas en ESC a la conversión iN en alta resolución.
Para descubrir dependencias genéticas diferenciales entre iN y estados alternativos, la abundancia relativa de sgRNA se correlacionó con un enfoque particular en genes agotados en iN y enriquecidos en los estados alternativos. Nos centramos en las dependencias diferenciales entre iN e iT inducidas por Ascl1 (Fig. 2d). Elegimos genes para la validación según los siguientes criterios: (1) genes que muestran pocos efectos en ESC (−2 <LFC <2); (2) genes que muestran enriquecimiento en iT (LFC > 1.5); (3) genes agotados en iN (LFC <-0.5). Notamos que los genes importantes en la regulación de la red de pluripotencia y la diferenciación de ESC: Tcf7l1, ptpn2, apc, Correa, cnot848,49,50,51,52,53,54, así como genes del ciclo celular. Cnk y Cdk1355,56 son necesarios para la conversión de iN inducida por Ascl1 al tiempo que se inhibe la formación de iT. Por el contrario, la eliminación de estos golpes tuvo poca o ninguna influencia en la conversión a neuronas impulsada por Ngn2 (Fig. 2d, e).
Para validar los hallazgos, realizamos un ensayo de competencia. Para implementar un control interno, introdujimos un vector tagBFP constitutivo en nuestra línea celular y derivamos un clon con una expresión tagBFP estable. Luego, las células se infectaron con el sgRNA que mostró el agotamiento más fuerte en la pantalla. Por separado, generamos una población de control isogénica, apuntando a tagBFP con una guía de control contra tagBFP y clasificando por tagBFP.negativo células. Posteriormente, las células knockout y de control se mezclaron en una proporción de 1:1 y se mantuvieron durante un pase adicional para adaptarlas a las mismas condiciones, antes de sembrar las células para la inducción (Fig. 2f). Después de 6 días de inducción, evaluamos la población iN Mapt-Venus positiva, así como la población Venus negativa, correspondiente a la formación de poblaciones iT/iNSC (Fig. 2f, gramo). Además, evaluamos la aptitud de los knockouts en las células ESC mientras cultivamos ESC durante 6 días adicionales (Fig. 2f). Como se esperaba, cuadrado fue esencial en poblaciones iN, iT o iNSC, mientras que no en células ESC, reflejando los resultados de la pantalla (Fig. 2c-e, gramo). De manera similar, la eliminación de genes relacionados con la pluripotencia apc, Tcf7l1 y ptpn2 abolió la formación de iN tras la inducción con Ascl1, al tiempo que permitió la formación de células iT (Fig. 2g). Además, confirmamos el fenotipo mediante inmunotinción el día 6 después de la inducción (Fig. 2h, yo). La eliminación de los éxitos provocó que Ascl3 no generara neuronas positivas para TUBB1 mientras aún generaba células CDX2+ y KRT8+. Curiosamente, algunas células que expresan TUBB3 específico de neurona carecen de morfología neuronal, lo que indica activación de genes neuronales pero no logran establecer un estado iN final (Figura complementaria. 9c). Asimismo, la focalización de Cnk or Cdk13 redujo fuertemente el número de iN tras la inducción de Ascl1. A diferencia de Ascl1, la conversión de Ngn2 iN no se ve afectada por la pérdida de Tcf7l1, ptpn2y Cdk13 y Ngn2 todavía puede generar tanto neuronas como células madre neurales, marcadas por SOX2 y PAX6 (Fig. 2g–yo) Sin embargo, apc y Cnk Los knockouts afectaron la formación de iN e iNSC inducida por Ngn2. No obstante, también validamos muchos otros genes que muestran dependencias diferenciales entre la inducción de Ascl1 y Ngn2 (suplementario). Figura 9d-h). En conjunto, nuestras pantallas paralelas de pérdida de función CRISPR/Cas9 revelaron múltiples dependencias comunes y diferenciales entre la diferenciación dirigida a iN inducida por Ascl1 y Ngn2.
Rápida regulación a la baja de la red de pluripotencia tras la inducción de Ascl1
A medida que la pantalla identificó genes involucrados en el mantenimiento de la red de pluripotencia como esenciales para la formación de iN inducida por Ascl1, investigamos cómo Ascl1 y Ngn2 desmontan la red de pluripotencia con más profundidad. Para esto, utilizamos un análisis de vías de ingenio utilizando todos los genes expresados diferencialmente un día después de la inducción de Ascl1 o Ngn2 (Figura complementaria). 10a–d, Higo. 1i). Como antes, el ingenioso análisis de enriquecimiento de la vía canónica mostró que tanto Ascl1 como Ngn2 inducen vías relacionadas con la biosíntesis del colesterol (Figura complementaria. 10b,d). Curiosamente, la respuesta transcripcional de Ascl1 se centra en la regulación negativa de la red de pluripotencia ESC y la autorrenovación (Fig. 10a, b).
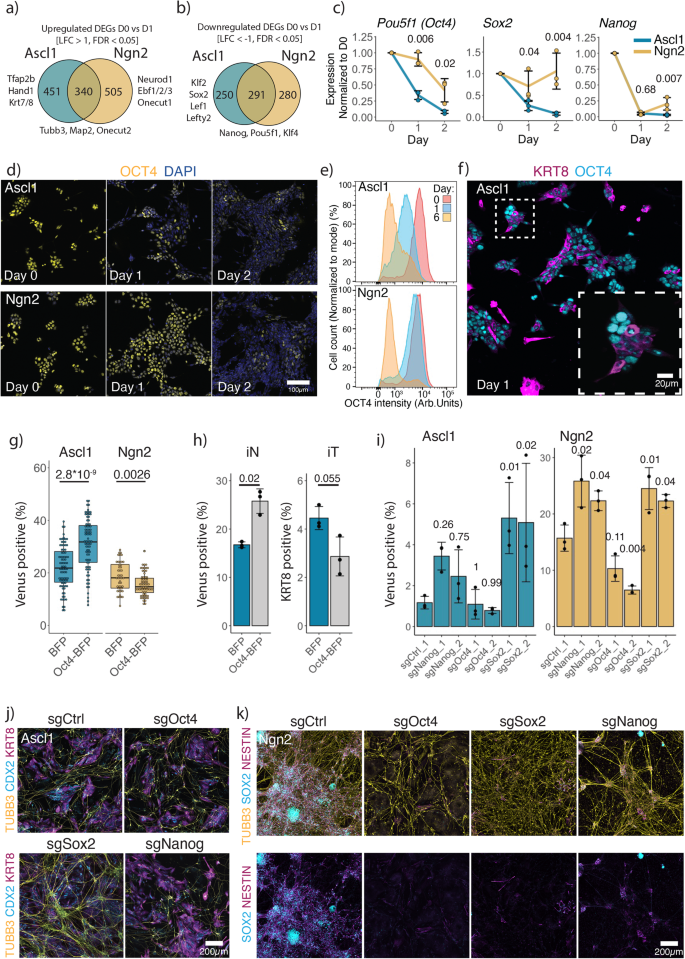
Luego dividimos los DEG en específicos de Ascl1 o Ngn2, o comunes, y realizamos un análisis de enriquecimiento de la vía KEGG (Fig. 3a, b; Figura complementaria. 10e). Curiosamente, Ascl1 regula positivamente los genes implicados en la senescencia celular y la salida del ciclo celular, como Cdkn1a, Cebpa, Cebbb (Fig. complementaria. 8d, gramo)57,58, así como también regula a la baja más genes implicados en la pluripotencia, por ejemplo, pou5f1 (que codifica la proteína OCT4), klf2, medias2, Lef1, Zurdo2, en comparación con Ngn2 (Fig. 3b, Higo. 1i, Fig. Suplementaria 10e). Por lo tanto, a continuación analizamos la dinámica del cierre de la red de pluripotencia (PPN) tras la inducción de Ascl1 y Ngn2. Para ello, nos centramos en los tres genes centrales de la pluripotencia: pou5f1 (4 de octubre), medias2, nanog (Higo. 3c, Higo. 1i). nanog está regulado negativamente con una cinética similar entre Ascl1 y Ngn2 (Fig. 3c, Higo. 1i, Fig. Suplementaria 11a, b). A diferencia de, medias2 La expresión se conserva después de la inducción de Ngn2 a medida que se establece una red reguladora del gen NSC, mientras que las células inducidas por Ascl1 pierden la expresión de medias2 (Higo. 3c, Higo. 1i, Fig. Suplementaria 11a, b). De manera similar, la inducción de Ascl1 conduce a una rápida pérdida de la expresión de Oct4, mientras que la inducción de Ngn2 conduce a una regulación negativa gradual de Oct4 (Fig. 3c-e, Higo. 1i). Además, la pérdida de Oct4 se corresponde con la regulación positiva del marcador trofoblástico KRT8 (Fig. 3f).
a, b Número de Ascl1, Ngn2 específicos o comunes regulados al alza (a) y regulado a la baja (b) genes de la Fig. 1i. c Datos de qPCR de la expresión de los genes centrales de pluripotencia (normalizados a la expresión de actina y el día 0). Las rectas se trazan por la media de n = 3 muestras biológicamente independientes; las barras de error indican ± SD. Arriba, p-valores de la prueba t de dos muestras de Welch bilateral que compara los genes PPN expresados entre Ascl1 y Ngn2 en el momento dado. d Inmunotinciones representativas de la dinámica de OCT4 después de la inducción de Ascl1 o Ngn2. e Cuantificación de la expresión de OCT4 mediante inmunotinción intracelular seguida de FACS. f Inmunotinción representativa para el marcador de pluripotencia OCT4 y el marcador de trofoblasto KRT8 el día 1 después de la inducción de Ascl1 o Ngn2. g Eficiencia de la formación de iN en presencia de sobreexpresión de OCT4. Cada punto representa un clon ESC individual que contiene una construcción de sobreexpresión. La eficiencia se mide por el porcentaje de población de Mapt-Venus. Los diagramas de caja indican los percentiles 25 y 75 como límites de la caja con la línea central mediana; los bigotes indican mínimos/máximos de una distancia de 1.5 veces del IQR desde los percentiles 25 y 75. p-valor de la prueba t de Welch bilateral para dos muestras indicada anteriormente. h Eficiencia de la formación de iN, medida por el porcentaje de células que expresan Mapt-Venus, y formación de iT, porcentaje de células inmunoteñidas para KRT8 de la población policlonal que sobreexpresa Oct4-BFP. El gráfico de barras muestra la media de n = 3 réplicas biológicas independientes con ± DE; el pLos valores se calcularon utilizando la prueba t de dos muestras de Welch bilateral. i Eficiencia de la formación de iN tras la eliminación aguda de la pluripotencia central. La eficiencia se mide por el porcentaje de población de Mapt-Venus. El diagrama de barras muestra la media de n = 3 muestras biológicamente independientes con ± DE. p-Los valores indicados anteriormente se calcularon utilizando ANOVA unidireccional seguido de la prueba de comparación múltiple de Dunnett (bilateral) utilizando sgCtrl como control. j Inmunotinciones representativas de (i) para iN e iT inducidos por Ascl1. j Inmunotinciones representativas de (i) para iN e iNSC inducidos por Ngn2. Los datos de origen se proporcionan como un archivo de datos de origen.
Para probar la relevancia funcional de la pérdida de Oct4, sobreexpresamos constitutivamente Oct4 y generamos múltiples líneas celulares clonales de mESC (Fig. 3g, h). Mientras que la sobreexpresión de Oct4 junto con Ngn2 conduce a una reducción de la población de iN, la coexpresión de Oct4 y Ascl1 aumenta la eficiencia de la formación de iN (Fig. 3g, h). A su vez, la formación de iT se ve obstaculizada en presencia de Oct4 (Fig. 3h). Durante el desarrollo, Oct4 inhibe la diferenciación del linaje del trofectodermo y la rápida pérdida de Oct4 se asocia con la regulación positiva de los marcadores del trofectodermo.59,60. Por lo tanto, la rápida pérdida de Oct4 genera condiciones permisivas para la formación del linaje iT, que compite con la formación de iN.
Dadas las diferencias cinéticas en la regulación negativa de PPN después de la inducción de Ascl1 o Ngn2, probamos si la interrupción forzada de PPN junto con la diferenciación afectaría la conversión de ESC a iN. Como pou5f1 (4 de octubre), medias2 y nanog son esenciales para la ESC (Fig. 2b), nosotros células infectadas con las guías dirigidas pou5f1 (4 de octubre), medias2 y nanog 2 días antes de la inducción de Ascl1 o Ngn2 (Fig. 3i-k). Curiosamente, la inducción de Ascl1 conduce a la formación tanto de iN como de iT en la eliminación de todos los factores de pluripotencia (Fig. 3j). Vemos poco o ningún efecto después de apuntar a Oct4, ya que Oct4 se pierde rápidamente tras la inducción de Ascl1 (Fig. 3i). Sin embargo, el nocaut de Nanog o medias2 favorece la formación de iN, lo que sugiere que una interrupción adicional de la PPN favorece la instalación del estado iN (Fig. 3i,j). A su vez, nocaut de medias2 abolió la formación de iNSC por Ngn2, mostrando que medias2 se reutiliza desde la PPN a la red reguladora de genes NSC (Fig. 3i,k). De manera similar, la interrupción de cualquiera de las dos nanog or pou5f1 (4 de octubre) también resultó en la pérdida de iNSC (Fig. 3k). En cambio, observamos la formación de endodermo y trofoblasto primitivos tras la pérdida de nanog or pou5f1 (4 de octubre), respectivamente (Fig. 3k, Fig. Suplementaria 11c, d), en alineación con el resultado de la pérdida de estos factores durante el desarrollo59,60,61.
Estos datos, junto con las dependencias diferenciales identificadas en la pantalla CRISPR, muestran que la inducción de Ascl1 y Ngn2 conduce a diferencias funcionales pronunciadas al salir del estado de pluripotencia de ESC. La inducción de Ascl1 conduce a un cierre eficiente de PPN y posterior inducción de la red de genes iN o iT, mientras que Ngn2 no regula completamente la PPN y, en cambio, superpone ambas redes, reutilizando genes para la inducción de NSC.
Se requiere Tcf7l1 para que la salida del ciclo celular genere Ascl1 iN
A continuación, nos centramos en el cierre de PPN inducido por Ascl1. En la transdiferenciación de MEF a iN impulsada por Ascl1, Myt1l facilita la formación de iN al reprimir el GRN de fibroblastos inicial, así como el linaje de mioblastos alternativo.16,62. Dada la rápida regulación a la baja de PPN, probamos si Myt1l es igualmente necesario para la conversión de ESC a iN. Sin embargo, nuestra pantalla no reveló eso. Mit1l, o sus parálogos mit1 y St18, son necesarios para la conversión de ESC a iN (Fig. 12a), así como experimentos de validación utilizando sgRNA adicionales (Fig. 12b). En la pantalla CRISPR identificamos Tcf7l1 (factor de células T/factor potenciador linfoide, también conocido como Tcf3), un represor de PPN, esencial para la formación de iN por Ascl1. Confirmamos el Tcf7l1 fenotipo knockout en un fondo E14 separado y descartó que la pérdida de Tcf7l1 afectara la expresión del transgén Ascl1 (Fig. 12c-e). Tcf7l1 está preparado para los múltiples promotores de genes relacionados con PPN y reprime rápidamente su expresión tras la diferenciación de ESC48,63,64, y la ausencia de Tcf7l1 estabiliza el estado ESC64. Por lo tanto, queríamos probar si se requiere Tcf7l1 antes o después del inicio de la conversión del tipo celular, así como excluir efectos secundarios debidos a la estabilización del PPN antes de la conversión. Para esto, etiquetamos Tcf7l1 en el extremo N-terminal con un degron inducible por auxina, infectamos células con un vector lentiviral que porta osTIR1 (F-box E3-ubiquitina ligasa, derivada de Oryza sativa) y generamos clones derivados de células individuales (Fig. 12f)65. Luego agotamos Tcf7l1 antes o al inicio de la conversión mediante la adición de auxina (Figura complementaria). 12g). Si bien el agotamiento de Tcf7l1 antes de la inducción tuvo solo un efecto leve en la formación de iN, la degradación de Tcf7l1 al inicio de la inducción de Ascl1 redujo severamente el número de neuronas producidas por Ascl1 (Figura complementaria. 12g). Esto indica que Tcf7l1 actúa después de la inducción en lugar de estabilizar el PPN.
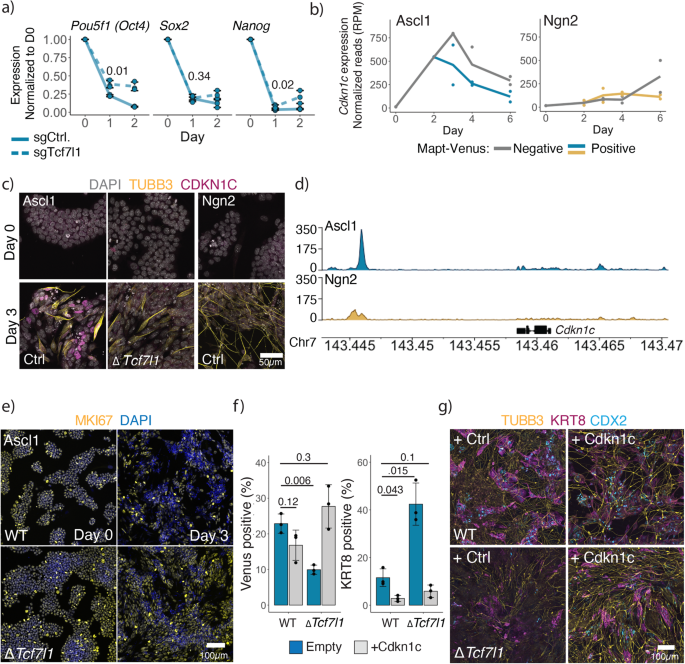
Para comprender el papel de Tcf7l1 en la ESC inducida por Ascl1 para la diferenciación dirigida por iN, generamos clones derivados de células individuales con un homocigoto. Tcf7l1 knockout y realizó RNAseq en masa el día 1 después de la inducción (Fig. 13a). Como Tcf7l1 actúa como un represor de la red de pluripotencia durante la diferenciación de ESC, primero observamos el cierre de la pluripotencia en el Tcf7l1 células knockout. Sorprendentemente, el PPN todavía estaba regulado negativamente después de la inducción de Ascl1 en ausencia de Tcf7l1 (Fig. 4a; Figura complementaria. 13a). Sin embargo, observamos un grupo de genes que no lograron expresarse en ΔTcf7l1 clones (Fig. 13a). Curiosamente, cdkn1c está altamente regulado positivamente después de la inducción de Ascl1 en comparación con ΔTcf7l1 (Fig. complementaria. 13a), y su expresión es específica para la inducción de Ascl1 y alcanza su punto máximo en el día 3 de la conversión de ESC a iN (Fig. 4b, c; Figura complementaria. 13b). Además, Ascl1 muestra una fuerte unión cerca cdkn1c locus en comparación con Ngn2 (Fig. 4d).
a qPCR de la expresión del gen central de pluripotencia después de la inducción en WT y Tcf7l1 Células KO. Expresión normalizada a actina y día 0. Las líneas se dibujan a través de la media de n = 3 muestras biológicamente independientes; las barras de error indican ± SD. p-valores de la prueba t de dos muestras de Welch bilateral que compara sgTcf1l1 con sgControl en el día 1 indicado anteriormente. b cdkn1c expresión durante la conversión de ESC a iN (Fig. 1a). c Inmunotinciones de CDKN1C el día 0 y el día 3 de la conversión de ESC a iN mediante Ascl1 WT o Tcf7l1 Células que expresan KO o Ngn2. d Enlace de Flag-Ascl1 o Flag-Ngn2 en el cdkn1c locus en el día 1 después de la inducción. Datos que muestran lecturas combinadas de cuatro réplicas. e Inmunotinciones del marcador de proliferación MKI67 después de la inducción de Ascl1 en WT o Tcf7l1 Células KO ESC. f Datos FACS de sobreexpresión de Cdkn1c con Ascl1 en WT o Tcf7l1 KO ESC. La eficiencia de la formación de iN se mide por el porcentaje de población de Mapt-Venus, la eficiencia de la formación de iT se mide por el porcentaje de células inmunoteñidas para KRT8. El diagrama de barras muestra la media de n = 3 réplicas biológicas independientes con ± DE. p-valor de la prueba t de Welch bilateral para dos muestras indicada anteriormente. g Imágenes representativas de células inmunoteñidas para marcadores neuronales TUBB3 y trofoblasto CDX2/KRT8 el día 6 después de la inducción de WT o Tcf7l1 KO ESC con Ascl1 y Cdkn1c. Los datos de origen se proporcionan como un archivo de datos de origen.
La diferenciación dirigida por Ascl1 depende del ciclo celular
Cdkn1c es un inhibidor de la quinasa dependiente de ciclina de la familia cip/kip que regula la detención del ciclo celular en G166. Cdkn1c es esencial para el desarrollo embrionario y los ratones que carecen de Cdkn1c mueren perinatalmente con múltiples defectos de desarrollo. Además, Cdkn1c es importante para el desarrollo temprano de la placenta para iniciar la endorreplicación de los trofoblastos.66. Recientemente, Cdkn1c también estuvo implicado en la supresión de la pluripotencia en ESC de ratón.67. Además, vemos que los nodos centrales GRN inducidos por Ascl1 contienen múltiples genes implicados en la regulación del ciclo celular, por ejemplo, cdkn1a, cdkn1b, cebpa, Cebbb (Fig. complementaria. 8d, gramo). Así, planteamos la hipótesis de que en ΔTcf7l1 células, Ascl1 es incapaz de detener el ciclo celular. De hecho, observamos una expresión prolongada de MKI67, así como un mayor porcentaje de células en división después de la inducción de Ascl1 en el ΔTcf7l1 células (Fig. 4e, Fig. Suplementaria 13c). Para probar si la expresión de Cdkn1c es suficiente para inducir la formación de iN en ausencia de Tcf7l1, coexpresamos Cdkn1c junto con Ascl1. De hecho, vemos un rescate parcial de la formación de iN así como una disminución en la generación de iT (Fig. 4f, gramo). Además, vemos que la expresión de Cdkn1c genera un fenotipo neuronal y trofoblástico más maduro también en poblaciones WT en el día 6 (Fig. 4g). Es de destacar que Cdkn1c no es esencial para la formación de iN inducidas por Ascl1, lo que sugiere que un grupo de reguladores del ciclo celular, en lugar de Cdkn1c solo, es responsable de la detención del ciclo celular (Fig. 13d).
En conjunto, nuestros datos muestran que en la conversión de ESC a iN inducida por Ascl1, la detención del ciclo celular es un obstáculo después de salir del estado de pluripotencia y que Cdkn1c es suficiente para superar este obstáculo. Por el contrario, esta dependencia no se observa en la formación de iN dependiente de Ngn2, donde el ciclo celular se mantiene y algunas ESC transitan a NSC.
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- PlatoData.Network Vertical Generativo Ai. Empodérate. Accede Aquí.
- PlatoAiStream. Inteligencia Web3. Conocimiento amplificado. Accede Aquí.
- PlatoESG. Automoción / vehículos eléctricos, Carbón, tecnología limpia, Energía, Ambiente, Solar, Gestión de residuos. Accede Aquí.
- PlatoSalud. Inteligencia en Biotecnología y Ensayos Clínicos. Accede Aquí.
- ChartPrime. Eleve su juego comercial con ChartPrime. Accede Aquí.
- Desplazamientos de bloque. Modernización de la propiedad de compensaciones ambientales. Accede Aquí.
- Fuente: https://www.nature.com/articles/s41467-023-40803-y