El aprendizaje no supervisado es una rama del aprendizaje automático en la que los modelos aprenden patrones a partir de los datos disponibles en lugar de proporcionarlos con la etiqueta real. Dejamos que el algoritmo proponga las respuestas.
En el aprendizaje no supervisado, existen dos técnicas principales; agrupamiento y reducción de dimensionalidad. La técnica de agrupamiento utiliza un algoritmo para aprender el patrón para segmentar los datos. Por el contrario, la técnica de reducción de dimensionalidad intenta reducir el número de características manteniendo la información real intacta tanto como sea posible.
Un algoritmo de ejemplo para el agrupamiento es K-Means, y para la reducción de dimensionalidad es PCA. Estos fueron los algoritmos más utilizados para el aprendizaje no supervisado. Sin embargo, rara vez hablamos de las métricas para evaluar el aprendizaje no supervisado. Tan útil como es, todavía necesitamos evaluar el resultado para saber si la salida es precisa.
Este artículo discutirá las métricas utilizadas para evaluar algoritmos de aprendizaje automático no supervisados y se dividirá en dos secciones; Métricas de algoritmos de agrupamiento y métricas de reducción de dimensionalidad. Entremos en ello.
No discutiremos en detalle sobre el algoritmo de agrupamiento ya que no es el punto principal de este artículo. En su lugar, nos centraríamos en ejemplos de las métricas utilizadas para la evaluación y cómo evaluar el resultado.
Este artículo utilizará el Conjunto de datos de vino de Kaggle como nuestro ejemplo de conjunto de datos. Primero leamos los datos y usemos el algoritmo K-Means para segmentar los datos.
import pandas as pd
from sklearn.cluster import KMeans
df = pd.read_csv('wine-clustering.csv') kmeans = KMeans(n_clusters=4, random_state=0)
kmeans.fit(df)Inicio el clúster como 4, lo que significa que segmentamos los datos en 4 clústeres. ¿Es el número correcto de grupos? ¿O hay algún número de clúster más adecuado? Comúnmente, podemos usar la técnica llamada método del codo para encontrar el clúster apropiado. Déjame mostrar el código a continuación.
wcss = []
for k in range(1, 11): kmeans = KMeans(n_clusters=k, random_state=0) kmeans.fit(df) wcss.append(kmeans.inertia_) # Plot the elbow method
plt.plot(range(1, 11), wcss, marker='o')
plt.xlabel('Number of Clusters (k)')
plt.ylabel('WCSS')
plt.title('Elbow Method')
plt.show() 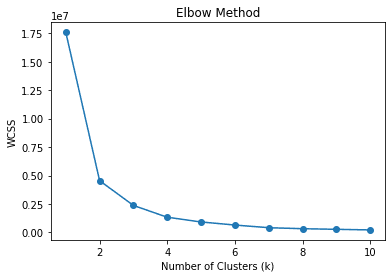
En el método del codo, usamos WCSS o la suma de cuadrados dentro del grupo para calcular la suma de las distancias al cuadrado entre los puntos de datos y los respectivos centroides de grupo para varios k (grupos). Se espera que el mejor valor de k sea el que tenga la mayor disminución de WCSS o el codo en la imagen de arriba, que es 2.
Sin embargo, podemos expandir el método del codo para usar otras métricas para encontrar la mejor k. ¿Qué tal si el algoritmo encuentra automáticamente el número de grupo sin depender del centroide? Sí, también podemos evaluarlos usando métricas similares.
Como nota, podemos asumir un centroide como la media de los datos para cada grupo aunque no usemos el algoritmo K-Means. Por lo tanto, cualquier algoritmo que no se basara en el centroide al segmentar los datos aún podría usar cualquier evaluación métrica que se base en el centroide.
Coeficiente de silueta
Silhouette es una técnica de agrupamiento para medir la similitud de los datos dentro del clúster en comparación con el otro clúster. El coeficiente de Silhouette es una representación numérica que va de -1 a 1. El valor 1 significa que cada grupo difiere completamente de los demás, y el valor -1 significa que todos los datos se asignaron al grupo incorrecto. 0 significa que no hay grupos significativos de los datos.
Podríamos usar el siguiente código para calcular el coeficiente de silueta.
# Calculate Silhouette Coefficient
from sklearn.metrics import silhouette_score sil_coeff = silhouette_score(df.drop("labels", axis=1), df["labels"])
print("Silhouette Coefficient:", round(sil_coeff, 3))Coeficiente de silueta: 0.562
Podemos ver que nuestra segmentación anterior tiene un coeficiente de silueta positivo, lo que significa que existe un grado de separación entre los grupos, aunque todavía se produce cierta superposición.
Índice de Calinski-Harabasz
El índice de Calinski-Harabasz o el criterio de relación de varianza es un índice que se utiliza para evaluar la calidad del conglomerado midiendo la relación entre la dispersión entre conglomerados y la dispersión dentro del conglomerado. Básicamente, medimos las diferencias entre la suma de la distancia al cuadrado de los datos entre el grupo y los datos dentro del grupo interno.
Cuanto mayor sea la puntuación del Índice de Calinski-Harabasz, mejor, lo que significa que los grupos estaban bien separados. Sin embargo, no hay límites superiores para la puntuación, lo que significa que esta métrica es mejor para evaluar diferentes números k en lugar de interpretar el resultado tal como es.
Usemos el código de Python para calcular la puntuación del Índice de Calinski-Harabasz.
# Calculate Calinski-Harabasz Index
from sklearn.metrics import calinski_harabasz_score ch_index = calinski_harabasz_score(df.drop('labels', axis=1), df['labels'])
print("Calinski-Harabasz Index:", round(ch_index, 3))Índice Calinski-Harabasz: 708.087
Otra consideración para la puntuación del Índice de Calinski-Harabasz es que la puntuación es sensible al número de grupos. Un mayor número de grupos también podría conducir a una puntuación más alta. Por lo tanto, es una buena idea usar otras métricas junto con el índice Calinski-Harabasz para validar el resultado.
Índice Davies-Bouldin
El índice Davies-Bouldin es una métrica de evaluación de agrupamiento medida mediante el cálculo de la similitud promedio entre cada grupo y su más similar. La relación entre las distancias dentro del grupo y las distancias entre grupos calcula la similitud. Esto significa que cuanto más separados estén los grupos y menos dispersos, se obtendrán mejores puntajes.
A diferencia de nuestras métricas anteriores, el Índice Davies-Bouldin tiene como objetivo tener una puntuación lo más baja posible. Cuanto más bajo era el puntaje, más separado estaba cada grupo. Usemos un ejemplo de Python para calcular la puntuación.
# Calculate Davies-Bouldin Index
from sklearn.metrics import davies_bouldin_score dbi = davies_bouldin_score(df.drop('labels', axis=1), df['labels'])
print("Davies-Bouldin Index:", round(dbi, 3))Índice de Davies-Bouldin: 0.544
No podemos decir que la puntuación anterior sea buena o mala porque, al igual que las métricas anteriores, todavía necesitamos evaluar el resultado utilizando varias métricas como apoyo.
A diferencia del agrupamiento, la reducción de la dimensionalidad tiene como objetivo reducir la cantidad de características mientras se preserva la información original tanto como sea posible. Por eso, muchas de las métricas de evaluación en la reducción de la dimensionalidad tenían que ver con la preservación de la información. Reduzcamos la dimensionalidad con PCA y veamos cómo funciona la métrica.
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler #Scaled the data
scaler = StandardScaler()
df_scaled = scaler.fit_transform(df) pca = PCA()
pca.fit(df_scaled)En el ejemplo anterior, ajustamos el PCA a los datos, pero aún no hemos reducido el número de características. En cambio, queremos evaluar la reducción de la dimensionalidad y la compensación de la varianza con el Varianza acumulada explicada. Es la métrica común para la reducción de dimensionalidad para ver cómo permanece la información con cada reducción de características.
#Calculate Cumulative Explained Variance
cev = np.cumsum(pca.explained_variance_ratio_) plt.plot(range(1, len(cev) + 1), cev, marker='o')
plt.xlabel('Number of PC')
plt.ylabel('CEV')
plt.title('CEV vs. Number of PC')
plt.grid() 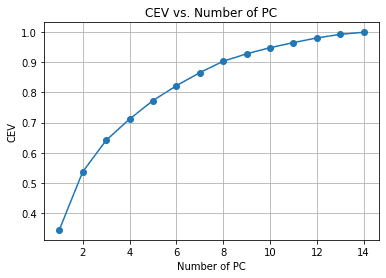
Podemos ver en el gráfico anterior la cantidad de PC retenida en comparación con la variación explicada. Como regla general, a menudo elegimos alrededor del 90-95% retenido cuando intentamos hacer una reducción de la dimensionalidad, por lo que alrededor de 14 características se reducen a 8 si seguimos el gráfico anterior.
Veamos las otras métricas para validar nuestra reducción de dimensionalidad.
Integridad
La confiabilidad es una medida de la calidad de la técnica de reducción de dimensionalidad. Esta métrica midió qué tan bien la dimensión reducida preservó los datos originales del vecino más cercano.
Básicamente, la métrica trata de ver qué tan bien la técnica de reducción de dimensiones preservó los datos manteniendo la estructura local de los datos originales.
La métrica de Confiabilidad oscila entre 0 y 1, donde los valores más cercanos a 1 significan que el vecino que está cerca de los puntos de datos de dimensión reducida también está cerca en su mayoría en la dimensión original.
Usemos el código de Python para calcular la métrica de confiabilidad.
from sklearn.manifold import trustworthiness # Calculate Trustworthiness. Tweak the number of neighbors depends on the dataset size.
tw = trustworthiness(df_scaled, df_pca, n_neighbors=5)
print("Trustworthiness:", round(tw, 3))Confiabilidad: 0.87
Mapeo de Sammon
El mapeo de Sammon es una técnica de reducción de dimensionalidad no lineal para preservar la distancia por pares de alta dimensionalidad cuando se reduce. El objetivo es utilizar la función de estrés de Sammon para calcular la distancia por pares entre los datos originales y el espacio de reducción.
Cuanto menor sea la puntuación de la función de estrés de Sammon, mejor porque indica una mejor conservación por parejas. Intentemos usar el ejemplo del código de Python.
Primero, instalaríamos un paquete adicional para Sammon's Mapping.
pip install sammon-mappingEntonces usaríamos el siguiente código para calcular la tensión de Sammon.
# Calculate Sammon's Stress
from sammon import sammon pca_res, sammon_st = sammon.sammon(np.array(df)) print("Sammon's Stress:", round(sammon_st, 5))El estrés de Sammon: 1e-05
El resultado mostró un puntaje de Sammon bajo, lo que significa que la preservación de los datos estaba allí.
El aprendizaje no supervisado es una rama del aprendizaje automático que intenta aprender el patrón de los datos. En comparación con el aprendizaje supervisado, es posible que la evaluación de resultados no discuta mucho. En este artículo, tratamos de aprender algunas métricas de aprendizaje no supervisado, que incluyen:
- Cuadrado de suma dentro del grupo
- Coeficiente de silueta
- Índice de Calinski-Harabasz
- Índice Davies-Bouldin
- Varianza acumulada explicada
- Integridad
- Mapeo de Sammon
Cornelio Yudha Wijaya es subgerente de ciencia de datos y escritor de datos. Mientras trabaja a tiempo completo en Allianz Indonesia, le encanta compartir consejos sobre Python y datos a través de las redes sociales y los medios de escritura.
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- Platoblockchain. Inteligencia del Metaverso Web3. Conocimiento amplificado. Accede Aquí.
- Acuñando el futuro con Adryenn Ashley. Accede Aquí.
- Fuente: https://www.kdnuggets.com/2023/04/exploring-unsupervised-learning-metrics.html?utm_source=rss&utm_medium=rss&utm_campaign=exploring-unsupervised-learning-metrics




