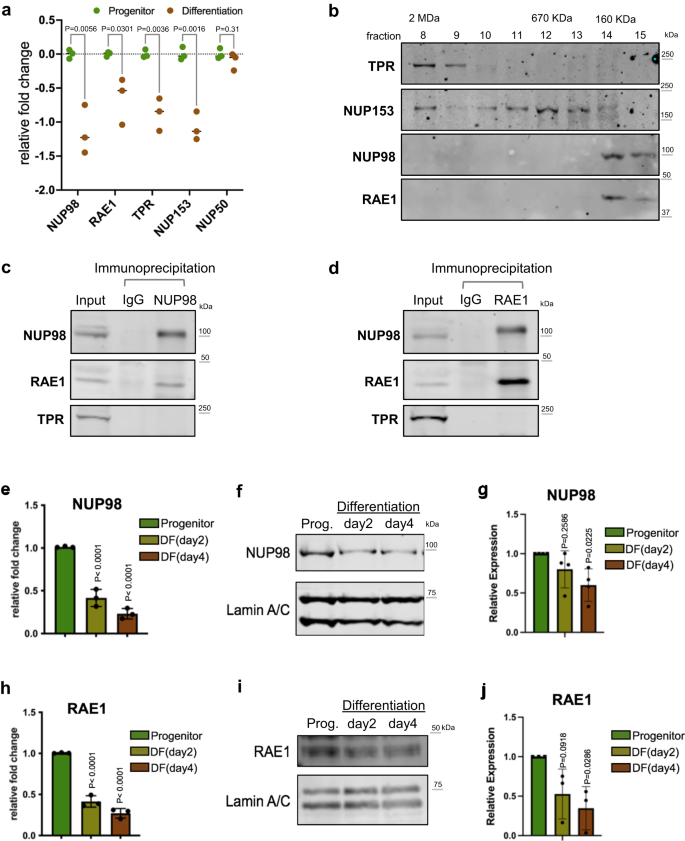
NUP98 y RAE1 están enriquecidos en progenitores y constituyen un complejo distinto
Entre los queratinocitos en estado progenitor versus los queratinocitos en estado de diferenciación, identificamos que 4 de 5 NUP de canasta nuclear están significativamente regulados a la baja en la diferenciación (Fig. 1a), aprovechando los datos de RNA-seq que generamos recientemente20. Para determinar cómo el enriquecimiento de estos NUP de canasta nuclear puede influir en el mantenimiento de los progenitores, preguntamos si estos NUP existen en otros complejos dentro del núcleo. Extrajimos las fracciones solubles de los núcleos de los queratinocitos en estado progenitor y realizamos una cromatografía de exclusión por tamaño. Mientras que TPR y NUP153 eluyeron en un rango de fracciones anteriores, correspondientes a complejos proteicos de mayor tamaño hasta 2MDa, NUP98 y RAE1 solo eluyeron en las fracciones posteriores alrededor del marcador de 160 KDa (Fig. 1b). La coelución de NUP98 y RAE1, en las mismas fracciones correspondientes a pequeños complejos proteicos, sugiere que estas dos proteínas pueden asociarse entre sí independientemente de las NUP. Para probar esto, realizamos coinmunoprecipitación utilizando extracción nuclear de queratinocitos en estado progenitor. El anticuerpo NUP98 co-inmunoprecipitó tanto NUP98 como RAE1; el anticuerpo RAE1 también co-inmunoprecipitó tanto RAE1 como NUP98. Ambas co-inmunoprecipitaciones no enriquecieron otras nucleoporinas como TPR (Fig. 1c, d). Dado que el ARN podría mediar en las interacciones de NUP98 con otras proteínas23, investigamos esto comparando co-inmunoprecipitaciones NUP98-RAE1 con o sin tratamiento con RNasa. Curiosamente, la asociación entre NUP98 y RAE1 se vio mínimamente afectada por la RNasa (Fig. 1a), lo que sugiere que la interacción NUP98-RAE1 no requiere un componente de ARN en este contexto. Además, probamos las proteínas inmunoprecipitadas por el anticuerpo NUP98 o RAE1 usando mAb414, que reconoce varios NUP que contienen dominios FG. Aunque mAb414 detectó múltiples bandas en el lisado de entrada, este patrón de bandas no se observó en la inmunoprecipitación por el anticuerpo NUP98 o RAE1 (Fig. 1b). Estos datos sugieren que NUP98 y RAE1 pueden desempeñar un papel en el estado progenitor independiente de otros NUP.
a Expresión relativa de ARNm de NUP de canasta nuclear, comparando el estado progenitor versus los queratinocitos humanos primarios diferenciados (día 4), según los datos de ARN-seq (prueba t no emparejada múltiple, N = 3 réplicas biológicas). b Transferencias de Western que muestran la distribución de NUP de canasta nuclear en las fracciones de cromatografía de exclusión por tamaño (SEC), utilizando la extracción soluble de los núcleos de queratinocitos en estado progenitor. Las fracciones correspondientes a los estándares de proteína para SEC están etiquetadas en la parte superior. c, d Western blots que muestran la co-inmunoprecipitación entre NUP98 y RAE1 en la extracción soluble de los núcleos (queratinocitos en estado progenitor). NUP98 y RAE1 co-inmunoprecipitaron entre sí, pero no otras subunidades de poro nuclear como TPR. e RT-qPCR que compara la expresión relativa de NUP98 a nivel de ARNm en el estado progenitor, el estado de diferenciación temprano (día 2) y medio (día 4) de los queratinocitos (ANOVA unidireccional con prueba post-hoc, N = 3 réplicas biológicas, los datos se representan como media ± desviación estándar). f, g Western blot y cuantificaciones que comparan la expresión de proteína NUP98 en la diferenciación de queratinocitos, con Lamin A/C utilizado como control de carga (ANOVA unidireccional con prueba post-hoc, N = 4, los datos se representan como media ± desviación estándar). h RT-qPCR que compara la expresión relativa del ARNm de RAE1 en la diferenciación de queratinocitos (ANOVA unidireccional con prueba post-hoc, los datos se representan como media ± desviación estándar). i, j Western blot y cuantificaciones que comparan los niveles de expresión de proteína RAE1 en la diferenciación de queratinocitos, con Lamin A/C utilizado como control de carga (ANOVA unidireccional con prueba post-hoc, N = 3, los datos de cuantificación se representan como media ± desviación estándar).
Para comprender mejor la expresión temporal de NUP98 y RAE1, en transición desde el estado progenitor hacia la diferenciación terminal, realizamos qRT-PCR y transferencia de Western para cuantificar sus niveles de ARNm y proteínas en el curso temporal de la diferenciación de queratinocitos inducida por calcio. Se detectó una reducción significativa de NUP98 y RAE1 en el día 2 (diferenciación temprana), y esta reducción se extendió hasta el día 4 (diferenciación media) del transcurso del tiempo de diferenciación (Fig. 1e-j). Estos datos indican que la regulación a la baja de NUP98 y RAE1 es un evento temprano en la diferenciación de queratinocitos, que puede desempeñar un papel en la regulación del cambio del estado progenitor a la diferenciación.
La caída de NUP98 o RAE1 afecta la capacidad regenerativa de los progenitores
Para determinar si la regulación a la baja de NUP98 o RAE1 promueve el cambio del estado progenitor hacia la diferenciación, aprovechamos la eliminación mediada por shRNA en los queratinocitos del estado progenitor, ajustando su expresión a un nivel comparable al estado de diferenciación. Se validaron tres shRNA independientes para NUP98 o RAE1 en los niveles de mRNA y proteína (Fig. 2a, b, Fig. Suplementaria 2a–d). La reducción de NUP98 o RAE1 por estos 6 shRNA individualmente fue suficiente para disminuir la clonogenicidad de los queratinocitos (Fig. 2c–f). Para evaluar los roles de NUP98 o RAE1 en la influencia de la capacidad regenerativa de los progenitores, realizamos una competencia de progenitores en la regeneración epidérmica de la piel. Se sembró una mezcla 50:50 de queratinocitos en estado progenitor que expresan GFP o DsRed sobre dermis humana desvitalizado, criados en interfaz líquido-aire. Este proceso de regeneración organotípico se completa en una semana, formando una epidermis de piel humana arquitectónicamente fiel18,19. Los queratinocitos que expresan GFP coexpresaron el shRNA no dirigido al control; los queratinocitos que expresan DsRed coexpresaron uno de los tres shRNA: shRNA de control no dirigido, shRNA dirigido a NUP98 o shRNA dirigido a RAE1. Para la epidermis regenerada usando queratinocitos que expresan los shRNA de control no dirigidos, los queratinocitos fluorescentes rojos y verdes mostraron una representación comparable en el tejido, lo que indica que la expresión de GFP o DsRed no influyó de manera diferente en la capacidad regenerativa de los progenitores. Por el contrario, los queratinocitos rojos que coexpresan NUP98 o RAE1 shRNA fueron superados por los queratinocitos verdes que coexpresan el shRNA de control, con una representación disminuida en el compartimento progenitor basal (Fig. 2g, h). Estos hallazgos sugieren que el alto nivel de expresión tanto de NUP98 como de RAE1 es esencial para el mantenimiento de progenitores.
a, b RT-qPCR que muestra la eficiencia de eliminación de 3 shRNA independientes dirigidos a NUP98 o RAE1 (ANOVA unidireccional con prueba post-hoc, N = 3, los datos se representan como media ± desviación estándar). c–f Imágenes representativas y cuantificación del ensayo de clonogenicidad que compara queratinocitos con NUP98 o RAE1 knockdown versus control (ANOVA unidireccional con prueba post-hoc, N = 3, los datos de cuantificación se representan como media ± desviación estándar). g Imágenes representativas del ensayo de competición en regeneración de tejido epidérmico. Un número igual de queratinocitos que expresan DsRed o GFP se mezclaron y sembraron en dermis humana. Los queratinocitos que expresan GFP coexpresan el shRNA de control y los queratinocitos que expresan DsRed coexpresan el shRNA de control o el shRNA dirigido a NUP98 o RAE1 (barra de escala = 125 μm). h Cuantificación de queratinocitos que expresan DsRed o GFP en la capa basal de la epidermis regenerada, los datos se representan como media ± desviación estándar. i Diagrama de Venn que muestra la superposición de los genes expresados diferencialmente de NUP98 y RAE1 (prueba exacta de Fisher, p = 1 × 10-321). j Mapa de calor que muestra la expresión relativa de genes compartidos con la eliminación de NUP98 o RAE1. k Los tres términos principales de ontología génica (GO) para los genes regulados al alza o a la baja compartidos por la eliminación de NUP98 y RAE1, identificados mediante análisis de RNA-seq.
Para identificar los procesos celulares clave influenciados por la caída de NUP98 o RAE1, realizamos un perfil de transcriptoma utilizando RNA-seq. En total, identificamos 1493 genes modificados significativamente con la eliminación de NUP98 y 1401 genes modificados significativamente con la eliminación de RAE1 (p < 0.05, dos colas, prueba de Wald, cambio de pliegue promedio para tres shRNA independientes > 2 y cambio de pliegue de shRNA individual > 1.5, datos complementarios 1, Dato suplementario 2). Estos dos conjuntos comparten 597 genes (p = 1 × 10-321, prueba exacta de Fisher), que se modifican en la misma dirección (regulados al alza o a la baja) con la caída de NUP98 o RAE1 (Fig. 2i,j). Los principales términos de ontología génica (GO) de los genes regulados al alza compartidos incluyen desarrollo epidérmico y diferenciación de queratinocitos; Los principales términos GO de los genes regulados a la baja compartidos están relacionados con la división celular (Fig. 2k). Además, identificamos que el 77% de estos genes compartidos también están significativamente alterados en la diferenciación inducida por calcio (Fig. S2e). Además, investigamos si la eliminación de NUP98 o RAE1 fue suficiente para desencadenar la apoptosis, aprovechando dos tintes diferentes que son sensibles al potencial mitocondrial. Mientras que la tinción se eliminó en los queratinocitos de control positivo tratados con H2O2, los queratinocitos con eliminación de NUP98 o RAE1 retuvieron la tinción de manera similar a la eliminación de control (Fig. 2f, gramo), lo que sugiere que la estrategia de eliminación no desencadenó la apoptosis. Por lo tanto, estos hallazgos sugieren que el enriquecimiento de NUP98 y RAE1 en el estado progenitor es esencial para mantener la proliferación y reprimir la diferenciación.
NUP98 se une cerca de los TSS de reguladores clave en el mantenimiento de progenitores
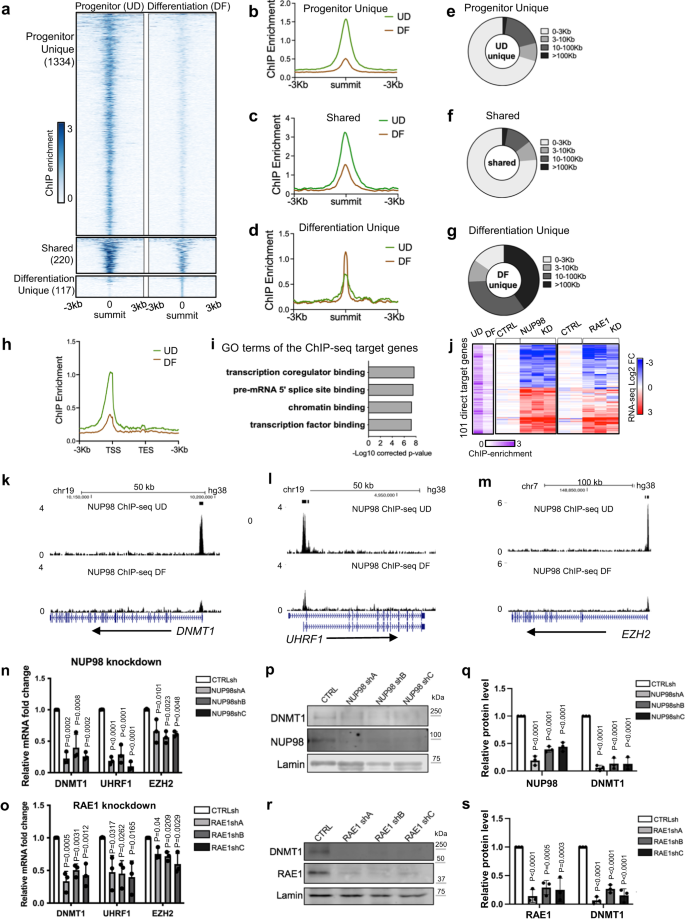
Se ha informado que NUP98 se une a diferentes regiones genómicas en el contexto de diferentes tipos de células14,24. Para investigar cómo la unión genómica de NUP98 podría influir en la expresión génica en el mantenimiento de progenitores epidérmicos, realizamos NUP98 ChIP-seq en queratinocitos. En los queratinocitos en estado progenitor, identificamos un total de 1554 picos de ChIP NUP98. Sin embargo, en el estado de diferenciación, la unión de NUP98 se reduce en todas estas regiones. La mayoría (86 %, 1334 picos) de estos picos NUP98 ChIP-seq son únicos para el estado progenitor, pero no para el estado de diferenciación. Solo el 14% de estos picos (220 picos) también se llamaron en el estado de diferenciación, sin embargo, el enriquecimiento de ChIP en estas regiones también se reduce en la diferenciación. Se identificó un pequeño número (117) de picos de ChIP NUP98 como exclusivos del estado de diferenciación, la mayoría de los cuales (74.3 %) se encuentran al menos a 10 kb de distancia de los TSS. Por el contrario, la mayoría de los picos (71%) identificados en el estado progenitor se encuentran dentro de las 3 kb desde los sitios de inicio de la transcripción (Fig. 3a-g, Dato suplementario 3). Por lo tanto, el cambio del estado progenitor a la diferenciación implicó una reducción general de la unión genómica de NUP98, especialmente cerca de los sitios de inicio de la transcripción (Fig. 3h).
a–d Mapas de calor centrados en la cumbre y diagramas promedio que comparan el enriquecimiento de ChIP NUP98 entre el estado progenitor (UD) y el estado de diferenciación (DF). e–g Gráficos circulares que muestran la distribución de los picos de ChIP-seq de NUP98, en función de sus distancias a los sitios de inicio transcripcional (TSS) más cercanos. h Diagrama promedio que compara el enriquecimiento de NUP98 ChIP-seq cerca de los sitios de inicio y finalización de la transcripción de los genes objetivo. i Los términos Top Gene Ontology (GO) de los genes más cercanos asociados con los picos NUP98 ChIP-seq. j Mapa de calor que muestra los 101 genes, con enriquecimiento de NUP98 ChIP-seq y se modifican significativamente con la eliminación de NUP98. El enriquecimiento relativo de NUP98 ChIP en UD y DF asociado con estos genes, y su expresión relativa con la eliminación de NUP98 o RAE1, se incluyen para cada uno de estos genes uno al lado del otro. k–m Ejemplos de seguimiento del navegador del enriquecimiento NUP98 ChIP-seq, comparando UD con DF. n, o Validación de qRT-PCR de genes diana representativos con eliminación de NUP98 o RAE1 (ANOVA unidireccional con prueba post-hoc, N = 3 réplicas biológicas, los datos se representan como media ± desviación estándar). p–s Western blot y cuantificación que comparan los niveles de proteína DNMT1 con NUP98 o RAE1 knockdown, con Lamin utilizado como control de carga (ANOVA unidireccional con prueba post-hoc, N = 3, los datos de cuantificación se representan como media ± desviación estándar).
Posteriormente, anotamos estos genes, que están asociados con picos de unión NUP98 ChIP-seq en el estado progenitor. Los términos principales de GO están relacionados con la unión del co-regulador de la transcripción, la unión de la cromatina y la unión del factor de transcripción (Fig. 3i), lo que sugiere que estos genes podrían ser reguladores aguas arriba de procesos biológicos clave. La intersección de los objetivos de ChIP-seq con los datos de NUP98 RNA-seq identificó un total de 101 genes objetivo directos. La mayoría de estos 101 genes objetivo directos también muestran una regulación ascendente o descendente similar con la eliminación de RAE1 (Fig. 3j). Curiosamente, los genes objetivo directos regulados a la baja incluyen reguladores clave que rigen el mantenimiento del progenitor epidérmico, incluida la ADN metiltransferasa DNMT1 y su reclutador URHF16, la proteína del grupo polycomb EZH23 (Higo. 3k–o), además de los reguladores de replicación del ADN CDT1 y RRM2. Estos genes objetivo directos también incluyen genes regulados al alza como DUSP10 y JARID2, que están menos caracterizados en el contexto de la diferenciación de queratinocitos (Datos complementarios 4).
Para determinar si RAE1 se une a la cromatina junto con NUP98, generamos una construcción RAE1 etiquetada con HA y la expresamos en queratinocitos, ya que los anticuerpos disponibles comercialmente que examinamos no produjeron datos ChIP-seq de alta calidad. HA-RAE1 coinmunoprecipitó NUP98 en los queratinocitos en estado progenitor (Fig. 3a), lo que confirma que la etiqueta HA no interfiere con la asociación de RAE1 con NUP98. Usando ChIP-seq de doble entrecruzamiento, identificamos que RAE1 está enriquecido en el 83% de las regiones pico de NUP98 ChIP-seq. Los picos de ChIP-seq superpuestos de NUP98-RAE1, pero no los picos únicos de NUP98, se encuentran predominantemente cerca de los TSS (Fig. 3b–f). Entre los 101 genes objetivo directos de NUP98, 96 de estos genes también están asociados con el enriquecimiento de RAE1 ChIP-seq, incluidos DNMT1 y EZH2 (Fig. 3g–yo). Estos hallazgos sugieren que NUP98 y RAE1 se unen directamente cerca de los TSS para regular la expresión génica.
DNMT1 se ha caracterizado previamente como un regulador clave para el mantenimiento de progenitores epidérmicos humanos6. Confirmamos la drástica regulación a la baja de DNMT1 en la diferenciación de queratinocitos mediante transferencia Western (Fig. 4a, b). Aprovechamiento de los datos de perfil de transcriptoma publicados de la caída de DNMT1 en queratinocitos en estado progenitor6, comparamos los genes expresados diferencialmente en la eliminación de DNMT1 con los genes expresados diferencialmente en la eliminación de NUP98 o RAE1. La intersección de estos conjuntos de datos identificó un total de 236 genes compartidos (Fig. 4c, d). Los principales términos GO de los genes compartidos regulados positivamente están asociados con el desarrollo epidérmico y la diferenciación de queratinocitos; los genes regulados a la baja están relacionados con la división celular y la replicación del ADN (Fig. 4e,f). Además, validamos mediante transferencia de Western que los niveles de proteína DNMT1 se reducen drásticamente con la eliminación de NUP98 o RAE1, en queratinocitos cultivados en la condición de progenitor (Fig. 3p-s). Por lo tanto, la regulación negativa de DNMT1 explica, al menos parcialmente, la inducción de la diferenciación y la inhibición de la proliferación observada con la eliminación de NUP98 o RAE1. En conjunto, estos datos sugieren que la unión de cromatina de NUP98 y RAE1 está involucrada en el mantenimiento de progenitores, mediante el control directo de la expresión de reguladores clave de proliferación/diferenciación como DNMT1.
NUP98 colocaliza con HDAC1 en la cromatina y coopera con HDAC en la regulación de genes
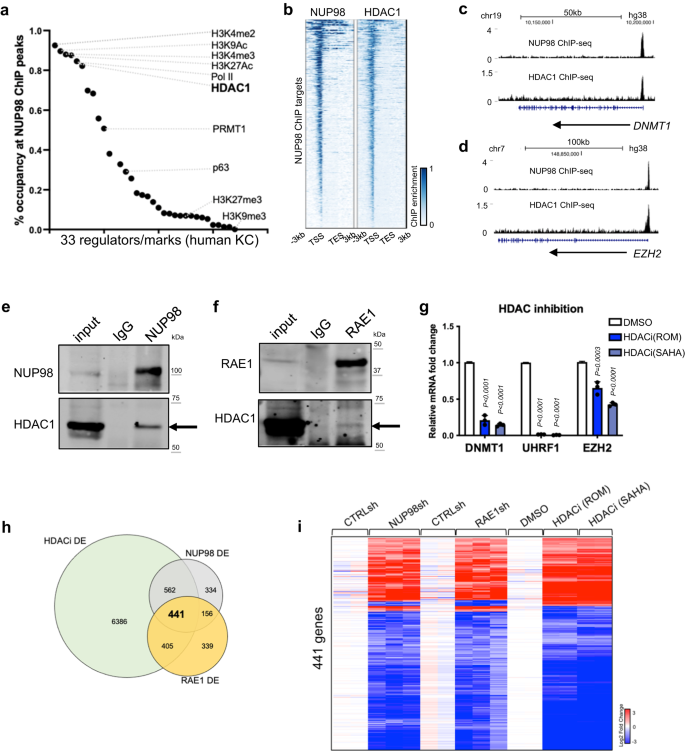
Dada la unión de la cromatina de NUP98 a los genes que codifican los reguladores clave del proceso de proliferación/diferenciación, investigamos los posibles mecanismos que facilitan la unión de la cromatina de NUP98 a estas regiones genómicas específicas. La búsqueda de motivos no descubrió factores de transcripción específicos que puedan explicar la mayoría de los sitios de unión genómicos. Luego comparamos los picos de NUP98 ChIP-seq con los archivos de picos de ChIP-seq de otros reguladores transcripcionales y marcas de histonas generadas usando el mismo tipo de célula de queratinocitos humanos primarios (Fig. 4a, Dato suplementario 5). En consonancia con la unión de NUP98 cerca de los sitios de inicio de la transcripción, las regiones pico de NUP98 ChIP-seq están muy ocupadas (>80 %) por las marcas de histonas (H3K4me2/3, H3K27Ac, H3K9Ac) y Pol II. Curiosamente, HDAC1 también ocupa el 82 % de todos los picos de NUP98 ChIP-seq, comparable a Pol II (85 %) y mucho más alto que el factor de transcripción p63 específico del linaje (29 %). Al igual que NUP98, HDAC1 también se une cerca de los TSS de los genes diana de NUP98, como DNMT1 y EZH2 (Fig. 4b–d). Por lo tanto, HDAC1 se destacó como un candidato que podría cooperar con NUP98 en la unión a cromatina y la regulación génica.
a Ocupación de otras marcas epigenéticas o reguladores en las regiones pico de NUP98 ChIP-seq. b Mapa de calor que compara NUP98 ChIP-seq y HDAC1 ChIP-seq en los picos de NUP98. c, d Pistas del navegador que muestran la coocupación de NUP98 y HDAC1 en genes diana representativos. e, f Co-inmunoprecipitación de entrecruzamiento usando NUP98 o anticuerpo RAE1 para detectar HDAC1. g qRT-PCR que muestra la expresión relativa de los genes diana RAE1-NUP98 con inhibición de HDAC (ANOVA unidireccional con prueba post-hoc, N = 3, los datos se representan como media ± desviación estándar). hDiagrama de Venn que muestra la superposición de los genes expresados diferencialmente NUP98, RAE1 y HDACi. i Mapa de calor que muestra la expresión de los genes expresados diferencialmente compartidos entre la eliminación de NUP98, la eliminación de RAE1 y la inhibición de HDAC.
Esta superposición extensa de los picos ChIP-seq de NUP98 y HDAC1 sugirió que estas dos proteínas podrían asociarse físicamente entre sí. Confirmamos esto usando inmunoprecipitación de entrecruzamiento, que el anticuerpo NUP98 o RAE1 coinmunoprecipitó HDAC1 en lisado de queratinocitos (Fig. 4e,f). Para determinar si HDAC influye en la expresión génica similar a NUP98 y RAE1, aprovechamos los inhibidores de HDAC Romidepsin (ROM) y SAHA. Cuando se agregaron a los queratinocitos cultivados en el estado progenitor, estos inhibidores regularon negativamente de manera constante los genes diana directos NUP98 representativos, como DNMT1, UHRF1 y EZH2 (Fig. 4g). Usando RNA-seq, identificamos además que el 74% de los genes diana NUP98-RAE1 también se alteran significativamente por la inhibición de HDAC (Fig. 4h, yo, Dato suplementario 6). Dado que p300 también se identificó como una proteína que interactúa con proteínas de fusión NUP98 en neoplasias malignas hematopoyéticas25, encontramos que la inhibición de p300 no alteró drásticamente la expresión del gen objetivo de NUP98 en queratinocitos (Fig. 5a), lo que respalda que HDAC está específicamente involucrado en la modulación de los genes diana controlados por NUP98 y RAE1 en el mantenimiento de progenitores epidérmicos.
Sobre la base de los cambios en la expresión génica de los genes diana de NUP98 y RAE1 inducidos por la inhibición de HDAC, investigamos más a fondo las funciones de HDAC1. Diseñamos y validamos un total de 3 shRNA independientes dirigidos a HDAC1. Curiosamente, estos tres shRNA regularon a la baja constantemente los genes objetivo de NUP98 y RAE1, como DNMT1 y UHRF1 (Fig. 5b). Por lo tanto, HDAC1 participa en la regulación de la expresión del gen objetivo de NUP98 y RAE1 en el estado progenitor. En el proceso de diferenciación de queratinocitos, el nivel de proteína de HDAC1 se reduce ligeramente, con una expresión relativa promedio del 54 % en el día 4 de diferenciación en comparación con el estado progenitor (Fig. 5c). De manera consistente, el enriquecimiento de HDAC1 ChIP solo se redujo moderadamente en el estado de diferenciación (Fig. 5d). Además, comparamos el enriquecimiento de HDAC1 ChIP-seq en los sitios de unión de NUP98 entre el estado progenitor y el estado de diferenciación (día 4, Fig. 5e-h). La mayoría (92.1%) de estos sitios no mostró una reducción drástica de la unión de HDAC1 en la diferenciación. Solo en una fracción muy pequeña (7.7%) de estos sitios, el enriquecimiento de HDAC1 mostró una reducción significativa (cambio de pliegue >2, p <0.05). Por lo tanto, el enriquecimiento de HDAC1 ChIP solo se redujo modestamente en la diferenciación en los sitios de unión de NUP98, en contraste con la reducción drástica de NUP98 en estos sitios de diferenciación, lo que sugiere que NUP98 no es necesario para mantener la unión de cromatina de HDAC1 en sus sitios de unión compartidos.
La unión de cromatina NUP98 depende de la actividad HDAC
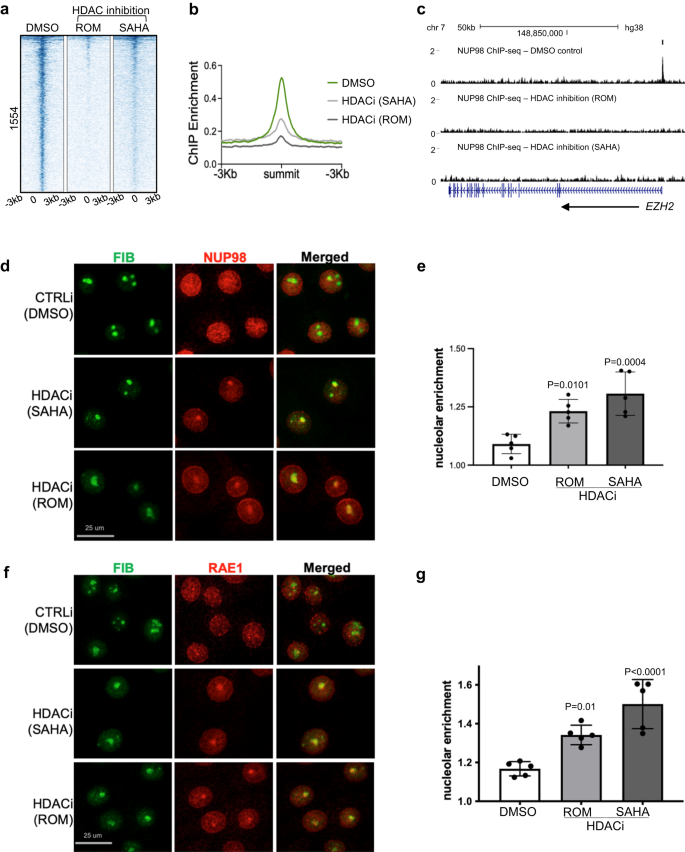
Dada la superposición entre NUP98 y HDAC1 en los sitios de unión genómica y en la regulación génica, preguntamos si la actividad de HDAC funciona aguas arriba para influir en la unión genómica de NUP98. Realizamos NUP98 ChIP-seq en queratinocitos con o sin inhibición de HDAC. Sorprendentemente, las señales de NUP98 ChIP-seq disminuyeron con la inhibición de HDAC utilizando SAHA o Romidepsin (Fig. 5a – c), lo que sugiere que la unión genómica de NUP98 a sus genes objetivo depende de la actividad de HDAC.
a Mapa de calor que muestra el enriquecimiento de ChIP NUP98 con inhibición de HDAC (usando ROM o SAHA) en comparación con el control DMSO. b Perfil promedio que muestra el enriquecimiento de ChIP NUP98 con inhibición de HDAC (usando ROM o SAHA) en comparación con el control DMSO. c Pista del navegador que muestra un gen objetivo representativo EZH2, comparando el enriquecimiento de NUP98 ChIP con inhibición de HDAC frente al control de DMSO, en queratinocitos cultivados en estado progenitor. d Imágenes representativas que muestran queratinocitos inmunoteñidos conjuntamente con anticuerpos dirigidos a NUP98 y el marcador de nucléolo fibrilarina (FIB), comparando la inhibición de HDAC con el control de DMSO (barra de escala = 25 μm). e Cuantificación del enriquecimiento relativo de NUP98 en el nucléolo en la inhibición de HDAC frente al control de DMSO (ANOVA unidireccional con prueba post-hoc, N = 5, los datos se representan como media ± desviación estándar). f, g Coinmunotinción de RAE1 y FIB en queratinocitos tratados con inhibidores de HDAC frente al control de DMSO (barra de escala = 25 μm) y cuantificación (ANOVA unidireccional con prueba post-hoc, N = 5, los datos de cuantificación se representan como media ± desviación estándar).
Para determinar si la localización subcelular de NUP98 también se vio afectada por la inhibición de HDAC, realizamos una tinción de inmunofluorescencia de NUP98 en queratinocitos con inhibición de HDAC en comparación con el control de DMSO. Si bien NUP98 mostró una tinción difusa dentro del núcleo en la condición de control, además de su enriquecimiento de la periferia nuclear, la inhibición de HDAC por SAHA o Romidespin resultó consistentemente en 1 o 2 focos agregados de NUP98 en cada núcleo. La tinción conjunta con el marcador de nucléolo fibrilina confirmó que el NUP98 se localizó en el nucléolo (Fig. 5d, e). Similar a NUP98, RAE1 también se agregó al nucléolo tras la inhibición de HDAC (Fig. 5f, gramo).
Un artículo reciente describió la orientación de FUS al nucléolo bajo estrés transcripcional.26. Preguntamos si FUS está involucrado en el enriquecimiento del nucléolo de NUP98 y RAE1. La tinción de coinmunofluorescencia se realizó tanto para NUP98 como para FUS con inhibición de HDAC, en comparación con el control. Si bien NUP98 se enriqueció constantemente en el nucléolo con inhibición de HDAC, no se detectó enriquecimiento de FUS (Fig. 6). Estos datos sugieren que el direccionamiento nucleolar de NUP98 y RAE1 es independiente de los mecanismos involucrados en el direccionamiento de FUS al nucleolo.
La localización nucleolar de NUP98 y RAE1 está equilibrada por las actividades de HDAC y HAT
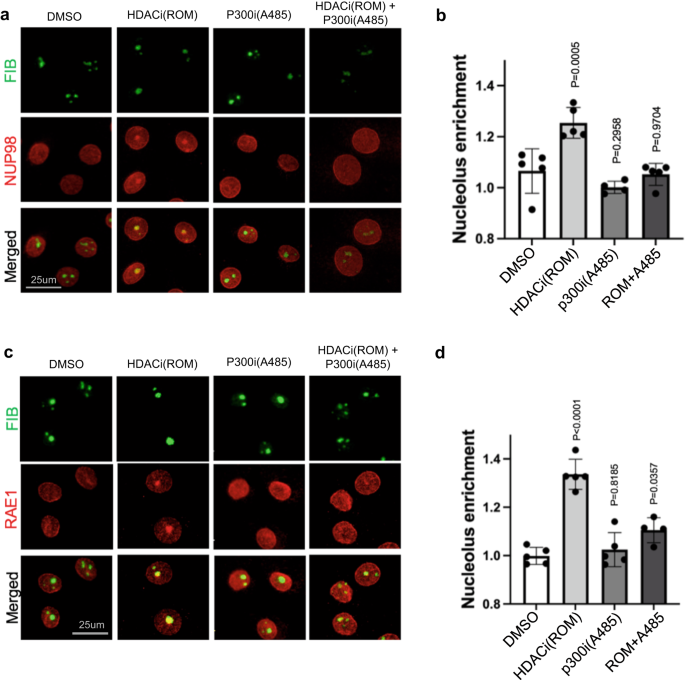
El enriquecimiento nucleolar de NUP98 y RAE1, inducido por la inhibición de HDAC, sugiere que la acetilación de proteínas podría estar involucrada en este proceso. Para probar esto, diseñamos un experimento para inhibir la acetilación de proteínas en combinación con la inhibición de HDAC, aprovechando los inhibidores A300 y C485 de p646/CBP HAT27,28. Como se muestra en la figura. 6 y Fig. Suplementaria. 7, la inhibición de HAT sola no alteró significativamente la localización subnuclear de NUP98 o RAE1; sin embargo, cuando los queratinocitos se trataron previamente con inhibidores de HAT durante 6 h, la inhibición de HDAC ya no pudo inducir la localización nucleolar de NUP98 o RAE1. Este antagonismo entre la inhibición de HAT y la inhibición de HDAC se observó consistentemente entre los dos inhibidores independientes de p300/CBP (A485 o C646). Estos datos respaldan el papel de la acetilación de proteínas en la mediación de la localización nucleolar de NUP98 o RAE1, como consecuencia de la inhibición de HDAC.
a Co-inmunotinción de NUP98 y marcador de nucléolo FIB en queratinocitos tratados con inhibidor de HDAC, ROM y/o inhibidor de HAT A485 (barra de escala = 25 μm). b cuantificación del enriquecimiento del nucléolo NUP98 en estas condiciones de tratamiento (ANOVA unidireccional con prueba post-hoc, N = 5, los datos se representan como media ± desviación estándar). c Coinmunotinción de RAE1 y el marcador de nucléolo FIB en queratinocitos tratados con inhibidor de HDAC, ROM y/o inhibidor de HAT A485 (barra de escala = 25 μm). d cuantificación del enriquecimiento del nucléolo RAE1 entre estas condiciones (ANOVA unidireccional con prueba post-hoc, N = 5, los datos se representan como media ± desviación estándar).
Interdependencia entre NUP98 y RAE1 en la localización nucleolar tras la inhibición de HDAC
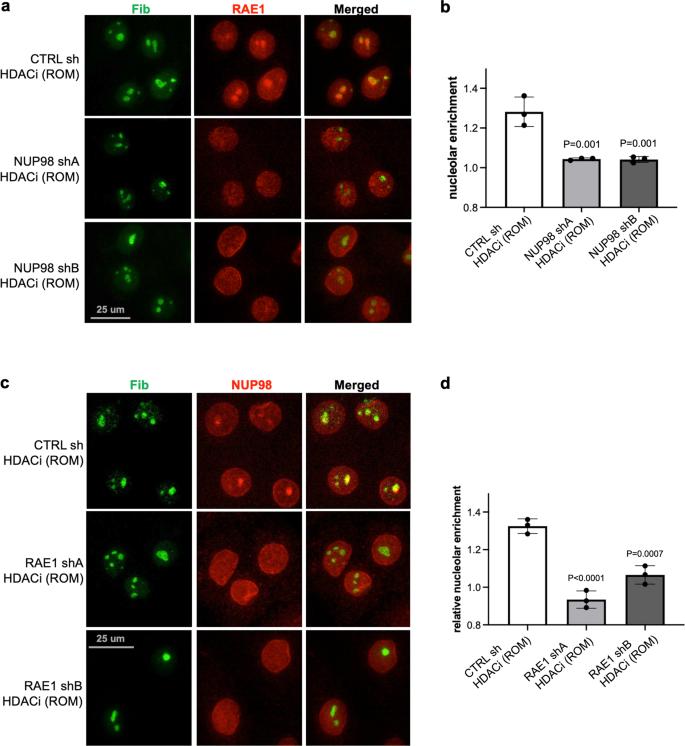
Dada la asociación entre NUP98 y RAE1, preguntamos además si la orientación nucleolar de NUP98 sobre la inhibición de HDAC depende de RAE1. Mientras que los queratinocitos que expresaban el shRNA de control no dirigido todavía mostraban agregación de NUP98 con inhibición de HDAC, esta agregación nucleolar se abolió en los queratinocitos que expresaban shRNA dirigidos a RAE1 (Fig. 7a, b). Por lo tanto, el enriquecimiento nucleolar de NUP98 requiere la función intacta de RAE1. Posteriormente, investigamos si la orientación nucleolar de RAE1 sobre la inhibición de HDAC también dependía de NUP98. Tratamos los queratinocitos que expresan NUP98 shA, NUP98 shB o control de shRNA no dirigido con la ROM del inhibidor de HDAC. El direccionamiento nucleolar de RAE1 se eliminó en los queratinocitos que expresaban shRNA NUP98, pero no en el control sin direccionamiento. Usando dos shRNA independientes dirigidos a NUP98, RAE1 tampoco se dirigió al nucléolo (Fig. 7c, d). La eliminación de RAE1 sola no indujo la localización nucleolar de NUP98, y la eliminación de NUP98 no indujo la localización nucleolar de RAE1, sin la inhibición de HDAC (Fig. 8). Estos hallazgos indican que NUP98 y RAE1 dependen el uno del otro para localizarse en el nucléolo tras la inhibición de HDAC.
a, b Imágenes representativas que muestran inmunotinción conjunta de RAE1 y el marcador de nucleolo FIB en queratinocitos tratados con eliminación de NUP98 versus eliminación de control (barra de escala = 25 μm) y cuantificaciones del enriquecimiento nucleolar de RAE1 entre estas condiciones (ANOVA unidireccional con prueba post-hoc, N = 3, los datos de cuantificación se representan como media ± desviación estándar). c, d Co-inmunotinción de NUP98 y el marcador de nucleolo FIB en queratinocitos tratados con reducción de RAE1 frente a reducción de control (barra de escala = 25 μm), y cuantificaciones del enriquecimiento nucleolar de NUP98 entre estas condiciones (ANOVA unidireccional con prueba post-hoc, N = 3, los datos de cuantificación se representan como media ± desviación estándar).
En conjunto, estos hallazgos sugieren un modelo en el que NUP98 y RAE1 promueven el mantenimiento de progenitores al unirse directamente y controlar la expresión de reguladores epigenéticos clave en dos condiciones: expresión elevada y actividad HDAC. La expresión elevada en el estado progenitor permite la presencia de un grupo intranuclear soluble de NUP98 y RAE1, lo que permite la unión de cromatina además de su asociación de poro nuclear; La actividad de HDAC también es esencial para antagonizar la orientación nucleolar de NUP98 y RAE1, promoviendo la orientación genómica de NUP98 a los reguladores epigenéticos clave para la autorrenovación (Fig. 8).
NUP98 y RAE1 respaldan el mantenimiento del progenitor epidérmico mediante la unión a la cromatina cerca de los sitios de inicio de la transcripción de genes diana clave, como el represor epigenético DNMT1. Los roles de NUP98 y RAE1 en el mantenimiento de progenitores dependen de dos factores: alta expresión y actividad HDAC. La alta expresión de NUP98 y RAE1 proporciona el grupo de unión a cromatina, además de su incorporación de poro nuclear; La actividad de HDAC respalda la unión de NUP98 a la cromatina cerca de los TSS de genes diana clave, lo que antagoniza la localización nucleolar de NUP98 y RAE1.
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- PlatoData.Network Vertical Generativo Ai. Empodérate. Accede Aquí.
- PlatoAiStream. Inteligencia Web3. Conocimiento amplificado. Accede Aquí.
- PlatoESG. Automoción / vehículos eléctricos, Carbón, tecnología limpia, Energía, Ambiente, Solar, Gestión de residuos. Accede Aquí.
- Desplazamientos de bloque. Modernización de la propiedad de compensaciones ambientales. Accede Aquí.
- Fuente: https://www.nature.com/articles/s42003-023-05043-2