Generación y caracterización de hiPSC y hiPSC-CM específicos del paciente.
Se establecieron líneas hiPSC a partir de dos pacientes TOF-DG, dos pacientes TOF-ND y dos controles sanos con marcadores de pluripotencia y marcadores de capa germinal verificados (Figs. 1 y 2). La secuenciación del genoma completo confirmó, respectivamente, la presencia y ausencia de deleción 22q11.2 en las líneas hiPSC de pacientes TOF-DG y TOF-ND (Fig. 3). No se encontraron mutaciones sin sentido o sin sentido de genes relacionados con la enfermedad coronaria previamente informados en ninguna de las líneas hiPSC. La diferenciación cardíaca se logró a través del APLNR+ protocolo de clasificación desarrollado por nuestro laboratorio32. Todas las líneas hiPSC mostraron una eficiencia de diferenciación satisfactoria con más del 70 % de células TNNT2+. El día 12 después de la diferenciación, las hiPSC-CM se fabricaron en hCAS y se dejaron madurar durante otros 10 días antes de las evaluaciones transcriptómicas y funcionales.
Los hiPSC-CM específicos del paciente TOF-DG presentan una especificación ventricular defectuosa
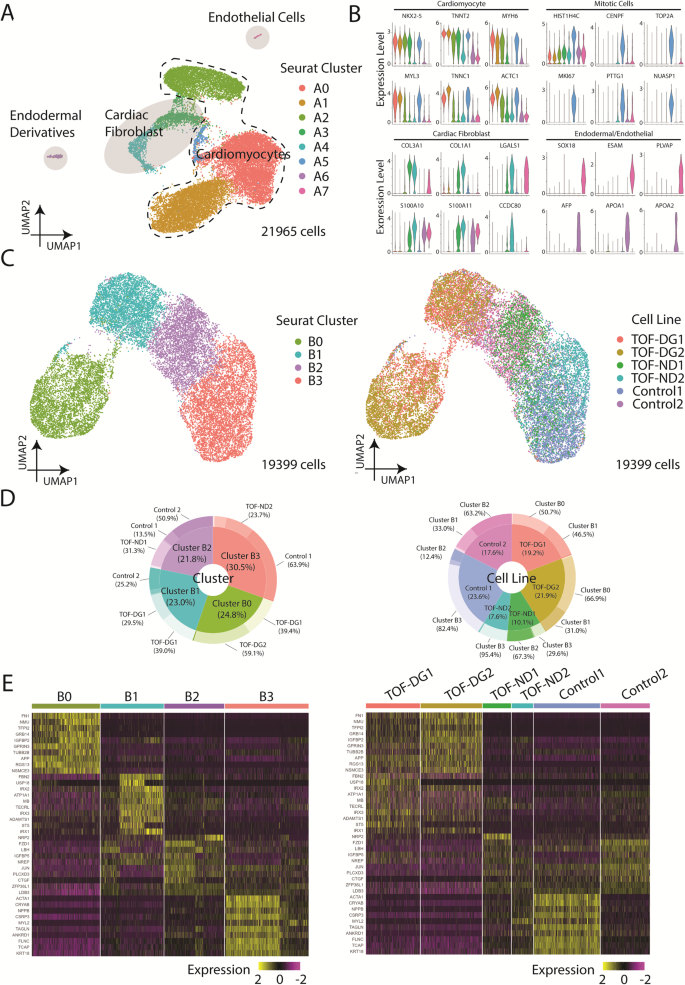
Primero determinamos el perfil transcriptómico de hiPSC-CM diferenciados de pacientes y controles mediante scRNA-seq utilizando la plataforma 10X Genomics. La agrupación de 21,965 células utilizando la aproximación y proyección de colector uniforme (UMAP) identificó 7 grupos de Seurat distintos (Fig. 1A). Los grupos de Seurat A0, A1, A2, A5 expresaron niveles relativamente más altos de genes cardíacos, incluidos NKX2-5, cadena ligera/pesada de miosina y troponina (Fig. 1B), que identificó estos grupos como cardiomiocitos. El grupo A5 también expresó niveles más altos de genes mitóticos, lo que sugiere la naturaleza proliferativa de los cardiomiocitos. Los grupos A3 y A4 expresaron niveles relativamente más bajos de genes cardíacos (NKX2-5, TNNT2) pero enriquecido en la expresión de genes relacionados con la remodelación de la matriz extracelular (MEC) (Fig. 1B), cuyo perfil es compatible con los fibroblastos cardíacos. Los grupos A6 y A7 se definieron por linaje endodérmico (AFP, APOA1, APOA2) y endotelial (FLT1, ESAM, PLVAP) marcadores, respectivamente (Fig. 1B). Así, entre las células secuenciadas, la mayoría eran cardiomiocitos (86%), algunas eran fibroblastos cardíacos (10%) y muy pocas eran endodérmicas (<1%) y derivados endoteliales (<1%).
A Presentación UMAP de todos los tipos celulares identificados en hCAS (TOF-DG, TOF-ND y control). Coloración: Racimos de Seurat; línea discontinua: cardiomiocitos; gris claro: no miocitos. B Gráficos de violín de expresiones genéticas de cardiomiocitos, células mitóticas y no miocitos: fibroblastos cardíacos, derivados endodérmicos y células endoteliales. Coloración: grupos de células de Seurat como en (A). C Presentaciones UMAP de todos los hiPSC-CM identificados en hCAS (TOF-DG, TOF-ND y control). Los datos fueron coloreados por grupos de Seurat (panel izquierdo) y línea celular (panel derecho), respectivamente. D Gráficos de anillos de las composiciones en cada grupo de Seurat (panel izquierdo) y línea celular (panel derecho), respectivamente. Los datos se calcularon únicamente a partir de hiPSC-CM. E Presentaciones de mapas de calor de los 10 principales genes regulados positivamente expresados en cada grupo hiPSC-CMs-Seurat. Los datos se agruparon por grupo de Seurat (panel izquierdo) y línea celular (panel derecho), respectivamente.
Centrándonos en examinar el transcriptoma cardíaco, aislamos hiPSC-CM (grupos A0, A1, A2, A5) y realizamos la agrupación utilizando UMAP (Fig. 1C). Se identificaron nuevos grupos de Seurat (B0, B1, B2, B3) (Fig. 1C). Los hiPSC-CM derivados de pacientes y controles se distribuyeron de manera desigual entre los grupos (Fig. 1D). El grupo B0 consistía en hiPSC-CM casi exclusivamente (98%) derivados de pacientes con TOF-DG, el grupo B1 consistía en células principalmente de un sujeto de control y las células restantes de pacientes con TOF-DG, mientras que los grupos B2 y B3 consistían en hiPSC-CM. derivados de pacientes y controles TOF-ND.
Se encontró que el grupo B0 tenía una expresión regulada positivamente de genes que no son miocitos, y 4 de cada 10 genes regulados positivamente se encuentran raramente (<1%) en otros grupos (Fig. 1E). Estos genes no miocíticos no estaban relacionados con el endodermo ni con el endotelio. La ausencia de enriquecimiento en genes relacionados con la remodelación de la matriz extracelular también distinguió las células del grupo B0 de los fibroblastos cardíacos. El grupo B1 se enriqueció en el IRX familia de genes (IRX1, IRX2, IRX3), mientras que el grupo B2 estuvo marcado por LWH expresión (fig. 1E). Los ortólogos de IRX genes y LWH Se ha informado que se expresan en cardiomiocitos murinos tempranos diferenciados in vivo.33,34. El grupo B2, en comparación con el grupo B1, tuvo una expresión aumentada de marcadores de cardiomiocitos ventriculares (MYH7, NPPB). El grupo B3 mostró la mayor expresión del marcador de cardiomiocitos ventriculares (MYL2) (Higo. 1E). La existencia de los grupos B1, B2 y B3 sugirió una especificación progresiva de hiPSC-CM hacia un perfil transcriptómico ventricular.
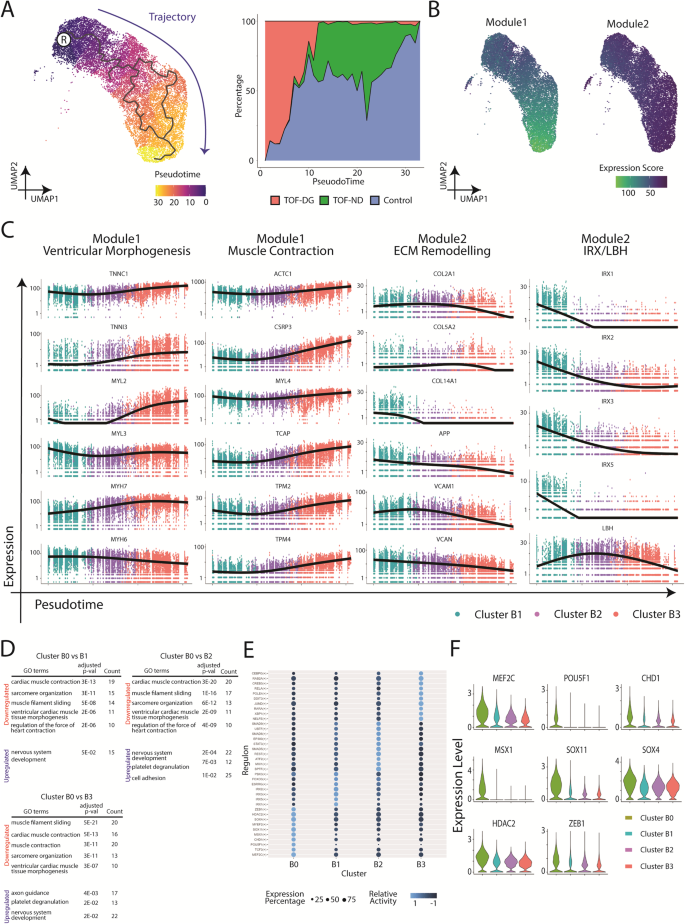
Para investigar más a fondo los cambios transcriptómicos durante la especificación ventricular, realizamos un análisis de pseudotiempo en los grupos B1, B2 y B3. Basado en el enriquecimiento en el IRX En la familia de genes, el grupo B1 se seleccionó manualmente como el nodo raíz para el análisis de pseudotiempo (Fig. 2A). A lo largo de la trayectoria, los TOF-DG-hiPSC-CM se distribuyeron principalmente al principio, mientras que los TOF-ND-hiPSC-CM se distribuyeron principalmente desde el medio hasta el final. Se pudieron encontrar Control-hiPSC-CM a lo largo de la trayectoria (Fig. 2A).
A Análisis de pseudotiempo de hCAS-hiPSC-CM. Coordinación UMAP y expresiones genéticas de los grupos B1, B2 y B3 (Fig. 1C) se extrapolaron para el cálculo de Monocle3. El nodo raíz se denota con ®. La trayectoria estaba coloreada en azul. B Los módulos genéticos sirven como función del análisis de pseudotiempo y la inferencia de trayectoria de hCAS-hiPSC-CM. C Dinámica de la expresión genética a lo largo de la trayectoria. La morfogénesis ventricular, la contracción muscular y la remodelación de la ECM fueron los términos GO enriquecidos en los módulos genéticos. X-eje: Pseudotiempo; Y-eje: Nivel de expresión. Matriz extracelular de la MEC. D Análisis de enriquecimiento GO del grupo B0 de hCAS-hiPSC-CM. Se mostraron los términos GO que se encuentran enriquecidos en los genes regulados negativamente/aumentados del grupo B0 (en comparación con el grupo B1/B2/B3). Los valores de p se ajustaron para comparaciones múltiples de Bonferroni. E Análisis de Regulon de hCAS-hiPSC-CM. Los principales regulones (como máximo 10) con mayor actividad relativa en cada grupo de Seurat se muestran en el gráfico del mapa de calor de puntos. F Expresión genética de los regulos superiores (identificados como DEG) del grupo B0. Las expresiones genéticas se mostraron en el gráfico del violín.
Los genes con patrones de expresión similares se agruparon en módulos (Fig. 2B) y sometido a análisis de enriquecimiento de ontología genética (GO). Se encontraron términos GO significativos tanto en el Módulo 1 como en el Módulo 2 (Fig. 2C). El módulo 1, que muestra altas puntuaciones de expresión a lo largo de la trayectoria, se enriqueció con genes relacionados con la morfogénesis ventricular (TNNI3, MYL2, MYH7) y contracción muscular (ACTC1, CSRP3, TCAP). El módulo 2, que muestra puntuaciones de expresión decrecientes a lo largo de la trayectoria, se enriqueció con genes relacionados con la remodelación de la matriz extracelular (COL2A1, COL5A2, COL14A1). La IRX familia de genes y LWH También fueron asignados al módulo 2, mostrando puntuaciones de expresión decrecientes a lo largo de la trayectoria.
En resumen, el análisis de pseudotiempo en hiPSC-CM derivados de la plataforma hCAS identificó una especificación ventricular progresiva con una mayor expresión de genes cardíacos. Los CM Control y TOF-ND-hiPSC eran comparables en especificaciones tan progresivas. Sin embargo, la mayoría de los TOF-DG-hiPSC-CM conservaron el perfil de expresión más primitivo.
Un subconjunto de hiPSC-CM específicos de pacientes de TOF-DG mostró una regulación negativa de la expresión de genes cardíacos pero una regulación positiva de la expresión de genes neuronales
Para analizar más a fondo la expresión de genes no miocitarios en el grupo B0, se identificaron genes expresados diferencialmente (DEG) comparando el grupo B0 con cada uno de los otros grupos. Se identificaron más de 200 DEG, regulados al alza o a la baja, en el grupo B0 (Datos complementarios 1). El análisis de enriquecimiento de GO identificó consistentemente términos GO significativos relacionados con la contracción del músculo cardíaco entre genes regulados negativamente y aquellos relacionados con el desarrollo neuronal entre genes regulados positivamente en el grupo B0 (Fig. 2D). Por lo tanto, el grupo B0 se diferenciaba del resto de los grupos por la expresión de genes cardíacos regulados negativamente, así como por la expresión de genes neurales regulados positivamente. Sin embargo, ninguno de los DEG anteriores, incluidos los genes cardíacos regulados negativamente y los genes neuronales regulados positivamente, pudo asignarse a la región haploinsuficiente en ambas líneas TOF-DG hiPSC.
Además, intentamos identificar los factores de transcripción (TF) correspondientes y las redes reguladoras de genes de cada uno de los grupos a través de la implementación en Python de la agrupación e inferencia de redes reguladoras unicelulares (pySCENIC). Entre los 10 TF principales con mayor actividad en el grupo B0, 4 estaban involucrados en el desarrollo neuronal (TCF3, SOX11, SOX4 y ZEB1) y 2 en el desarrollo cardíaco y neural (MEF2C y MIEF2) (Higo. 2E). Los niveles de expresión de cinco TF neuronales (SOX11, SOX4, ZEB1, MEF2C y MIEF2) estaban regulados positivamente en el grupo B0 en comparación con aquellos en otros grupos (Fig. 2F). El hallazgo de una expresión regulada positivamente de estos TF neuronales fue consistente con una mayor expresión de genes neuronales en el grupo B0. De manera similar a los DEG, los TF neuronales anteriores no se asignaron a la región haploinsuficiente en ambas líneas celulares TOF-DG.
Por lo tanto, una porción significativa de hiPSC-CM de TOF-DG (grupo B0) mostró una regulación negativa de los genes cardíacos y una regulación positiva de los genes neuronales ectópicos. La imposibilidad de asignar los DEG y TF en el grupo B0 a la región 22q11.2 sugirió que esta firma transcriptómica es probablemente una consecuencia indirecta de la haploinsuficiencia. A diferencia de los hiPSC-CM de los grupos control y TOF-ND, que mostraron una continuidad en la especificación ventricular, el grupo B0 formó un grupo distinto del resto de los hiPSC-CM de TOF-DG (grupo B1). El origen del grupo B0 se exploró más a fondo como se describe a continuación.
Diferenciación cardíaca bifurcada de progenitores cardíacos TOF-DG
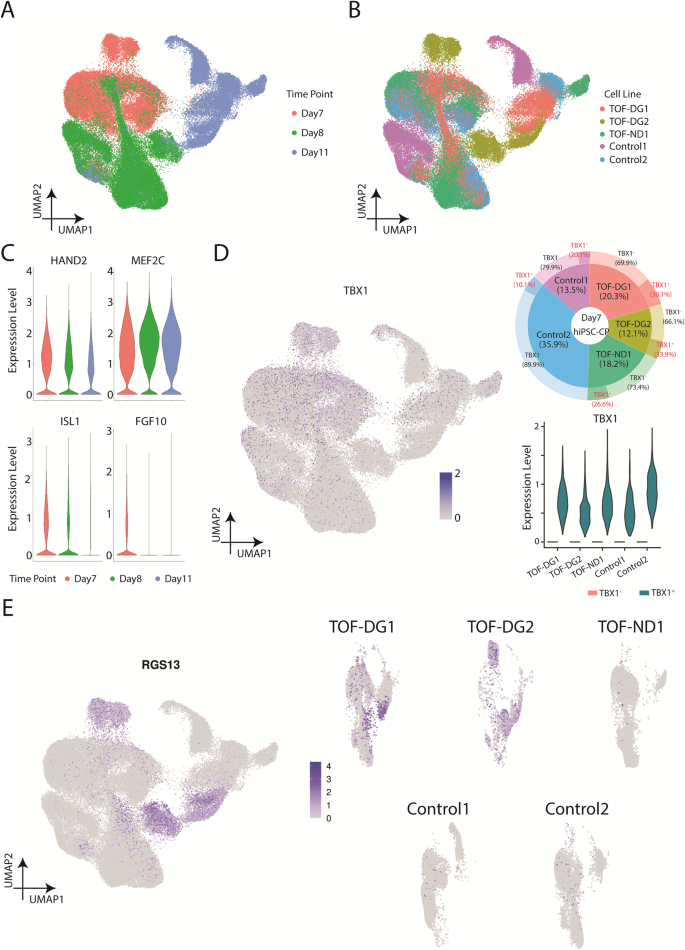
Para comprender la aparición del grupo B0 de TOF-DG, examinamos el perfil transcriptómico de progenitores cardíacos in vitro. Basado en nuestro estudio anterior32, la expresión del gen del segundo campo cardíaco (SHF) alcanzó su punto máximo en los días 7 y 8 posteriores a la diferenciación. Por lo tanto, realizamos scRNA-seq en progenitores cardíacos hiPSC (hiPSC-CP) en D7 y D8 y hiPSC-CM inmaduros en D11 de 2 controles (Control 1 y 2) y 2 pacientes TOF-DG (TOF-DG1 y 2) y 1 TOF-ND (TOF-ND1) (Fig. 3A,B). En total, se retuvieron 87834 células después de excluir menos del 1% de los derivados endodérmicos y las células endoteliales. El análisis de enriquecimiento de pseudotiempo y GO identificó una regulación positiva en la expresión de genes cardíacos y una regulación negativa en la transcripción y traducción y la actividad del ciclo celular a lo largo de la trayectoria (Figura complementaria. 4A-D). Similar a nuestro estudio anterior32, expresiones del gen SHF, incluyendo ISL1, MEF2C, HAND2 y FGF10, se encontraron en los puntos de tiempo secuenciados (Fig. 3C). TBX1, uno de los genes dentro de la región de microdeleción 22q11.2, también es un marcador de SHF. Entre los puntos de tiempo secuenciados, TBX1 expresión se encontró el día 7, con una proporción variable de TBX1+ hiPSC-CP (10-30%) encontrados en diferentes líneas (Fig. 3D) sin embargo, el TBX1 El nivel de expresión fue comparable entre los D7-TBX1+ hiPSC-CP de diferentes líneas (Fig. 3D).
A Presentaciones UMAP de todos los hiPSC-CP y hiPSC-CM (TOF-DG, TOF-ND y control). Los datos fueron coloreados por punto de tiempo secuenciado. B Presentaciones UMAP de todos los hiPSC-CP y hiPSC-CM (TOF-DG, TOF-ND y control). Los datos fueron coloreados por línea celular. C Gráficos de violín de la expresión del gen SHF, que incluyen ISL1, MEF2C, MANO2 y FGF10. D TBX1 expresión en hiPSC-CP. TBX1 La expresión el día 7/8/11 se mostró la diferenciación cardíaca in vitro en el gráfico UMAP. más alto TBX1 La expresión se encontró en D7-hiPSC-CP, y la proporción de los TBX1+ se mostró en un diagrama de anillos y se marcó con una etiqueta roja. TBX1 expresión en TBX1+ D7-hiPSC-CP se mostró en un diagrama de violín. E parcelas UMAP de RGS13 expresión el día 7/8/11 diferenciación cardíaca in vitro. RGS13 La expresión se presentó con todas las líneas celulares juntas (panel izquierdo) y se dividió en cada línea celular individual (panel derecho).
La comparación de tiempo coincidente con los controles solo identificó 20, 10 y 22 DEG de D7-, D8- y D11-TOF-DG-hiPSC-CP/CM sin encontrar términos GO significativos. Ninguno de estos DEG se asignó a las regiones de microdeleción 22q11.2 en las dos líneas TOF-DG-hiPSC. Luego se examinaron las expresiones de genes ectópicos del grupo B0 de TOF-DG en los hiPSC-CP y en los hiPSC-CM inmaduros. A diferencia de TOF-DG-cluster B0 (hiPSC-CM diferenciados), no se encontraron genes relacionados con los nervios en TOF-DG-hiPSC-CP (D7 y D8) y D11-hiPSC-CM inmaduros. Por otro lado, un subconjunto de células marcadas por RGS13 expresión, 1 de los 10 principales genes regulados positivamente en el grupo B0 de TOF-DG (Fig. 1E), se encontró consistentemente en células derivadas de TOF-DG en todos los puntos de tiempo secuenciados (D7, D8 y D11) (Fig. 3E), pero no en TOF-ND1 y los grupos de control.
Recientemente se ha informado de la expresión de genes ectópicos en los progenitores de tbx1 nulo condicional (tbx1-cKO) ratón35. Para comparar nuestros datos de hiPSC-CP y hiPSC-CM inmaduro con el tbx1-cKO mouse, hicimos una comparación entre especies con el paquete R SingleCellNet36. Se identificaron catorce grupos de tbx1-Conjunto de datos de ratón cKO (Fig. 5A) con expresión genética reportada en la publicación35 (Fig. complementaria. 5B), incluidos cuatro progenitores cardíacos diferentes (progenitor multilinaje, MLP; SHF anterior, aSHF; SHF posterior, pSHF y proepicardio; PEO) y cardiomiocitos. Nuestros D7-hiPSC-CP fueron en su mayoría similares a aSHF, mientras que los cardiomiocitos inmaduros D11-hiPSC fueron en su mayoría similares a los cardiomiocitos (Fig. 5C). En el tbx1-cKO conjunto de datos del ratón, pax8, el principal gen ectópico informado por Nomaru et al.35, se encontró exclusivamente en MLP y progenitores pulmonares, pero no en aSHF y cardiomiocitos (Fig. 5D). no nos identificamos PAX8 expresión en nuestro conjunto de datos D7-D11. También, RGS13, el gen ectópico encontrado en nuestro conjunto de datos D7-D11, no fue identificado en el tbx1-cKO ratón. En otras palabras, si bien se encontró expresión de genes ectópicos en ambos tbx1-cKO ratón y nuestros DG-hiPSC-CP/CM, la identidad y la población que expresa el gen ectópico son diferentes.
Parámetros electrofisiológicos alterados y aumento de la arritmogenicidad en TOF-DG-hiPSC-CM
Además del perfil transcriptómico, exploramos la electrofisiología y la arritmogenicidad de los hiPSC-CM diferenciados utilizando nuestra plataforma hCAS para proporcionar una evaluación más completa de los hiPSC-CM. Los parámetros electrofisiológicos, incluido el potencial de acción (AP), la manipulación del calcio y el período refractario efectivo (ERP), se midieron con estimulación eléctrica a 1 Hz.
En comparación con ambos controles, TOF-DG2-hCAS mostró un acortamiento significativo de la duración de la AP (50 y 90% hasta la repolarización, APD50 y APD90) (Fig. 4A). De manera similar, en comparación con ambos controles, TOF-DG2-hCAS mostró un acortamiento significativo del tiempo de ascenso de AP y del tiempo de caída (50 y 90 % desde los picos) (Fig. 4A). Por lo tanto, las características de AP difirieron significativamente entre TOF-DG2 y los controles.
A Gráficos de puntos de la característica del potencial de acción (AP). Duración del potencial de acción de la APD. B Gráficos de puntos de la característica transitoria de calcio (CaT). C Diagrama de puntos del período refractario efectivo. Los datos se presentaron en media ± DE para la Fig. 1A – C. Prueba estadística: ANOVA unidireccional ordinario seguido de la prueba de comparaciones múltiples de Tukey (APD50, AP90%Decay, CaT 50%Decay, CaT Upstroke y período refractario efectivo); Prueba de Kruskal-Wallis seguida de la prueba de comparación múltiple de Dunn (APD90, carrera ascendente AP, AP50%Decay y CaT 90%Decay). Los hallazgos significativos entre TOF(DG/ND) y el control se marcaron con asteriscos. * P <0.05; ** P <0.01; *** P < 0.001; **** P <0.0001. D Porcentaje de evento de reentrada arrítmica de TOF (DG/ND) y hCAS de control durante la estimulación en estado estacionario (SteadyState) y la estimulación eléctrica programada (PES). Prueba estadística: prueba exacta de Fisher. *** P <0.001. E Seguimiento representativo del potencial de acción (AP) y mapa isócrono del evento de reentrada durante PES desde TOF-DG2. El panel superior mostró el trazado AP representativo durante PES (S1S2) seguido de la incidencia del evento de reentrada arrítmica. El panel inferior mostró dos mapas isócronos que corresponden a una AP normal (izquierda) y una AP arrítmica (derecha). F Regulación negativa de genes cardíacos en el grupo B0-TOF-DG2. El análisis de enriquecimiento de GO se realizó en la comparación entre el grupo B0-TOF-DG2 y el grupo B0-TOF-DG1. Los términos GO enriquecidos en los genes regulados negativamente se muestran en la tabla (izquierda) y los genes correspondientes se muestran en el gráfico del violín (derecha).
El manejo del calcio se evaluó con el tiempo de ascenso transitorio de calcio (CaT) y el tiempo hasta la caída (50 y 90 % desde el pico). Se encontró un acortamiento significativo del tiempo de ascenso de CaT en TOF-DG2-hCAS, mientras que se encontró un acortamiento significativo del tiempo hasta la caída (50% desde el pico) tanto en TOF-DG2-hCAS como en TOF-ND1-hCAS en comparación con ambos controles ( Higo. 4B).
Además, evaluamos el ERP de los hiPSC-CM, cuyo acortamiento predispone al desarrollo de arritmias cardíacas. Se encontró un acortamiento significativo de EPR en TOF-DG2-hCAS, mientras que se encontró una prolongación de ERP en TOF-ND2-hCAS (Fig. 4C). Es importante destacar que TOF-DG2-hCAS mostró una incidencia significativamente mayor de arritmia de reentrada durante la estimulación eléctrica programada (PES) (Fig. 4D). En la figura se muestra un trazado AP representativo con el mapa isócrono de arritmia de reentrada de TOF-DG2-hCAS. 4E.
Mientras que TOF-DG2-hCAS mostró un acortamiento significativo de la duración de AP, el tiempo de ascenso de CaT y el tiempo de caída, y ERP, TOF-DG1-hCAS fue similar a los controles en términos de estos parámetros electrofisiológicos evaluados (Fig. 4A-D). Por lo tanto, exploramos más a fondo las diferencias transcriptómicas entre TOF-DG1 y TOF-DG2 en el grupo B0. En comparación con el grupo B0-TOF-DG1, el grupo B0-TOF-DG2 mostró una regulación negativa de los genes cardíacos relacionados con la contracción muscular (Fig. 4F). Si bien estos genes cardíacos estaban regulados negativamente tanto en el grupo B0-TOF-DG1 como en el grupo B0-TOF-DG2 en comparación con otros grupos (B1, B2 y B3) (Fig. 4F), su grado de regulación negativa fue más prominente en el grupo B0-TOF-DG2 (Fig. 4F). Además, PKP2, se ha informado que su mutación está asociada con displasia arritmogénica del VD37, se encontró entre los genes cardíacos regulados negativamente en el grupo B0-TOF-DG2 (Fig. 4F). Por lo tanto, el grado variable de regulación negativa de los genes cardíacos puede explicar la discrepancia funcional observada entre líneas celulares de diferentes pacientes en la plataforma hCAS.
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- PlatoData.Network Vertical Generativo Ai. Empodérate. Accede Aquí.
- PlatoAiStream. Inteligencia Web3. Conocimiento amplificado. Accede Aquí.
- PlatoESG. Carbón, tecnología limpia, Energía, Ambiente, Solar, Gestión de residuos. Accede Aquí.
- PlatoSalud. Inteligencia en Biotecnología y Ensayos Clínicos. Accede Aquí.
- Fuente: https://www.nature.com/articles/s42003-023-05344-6