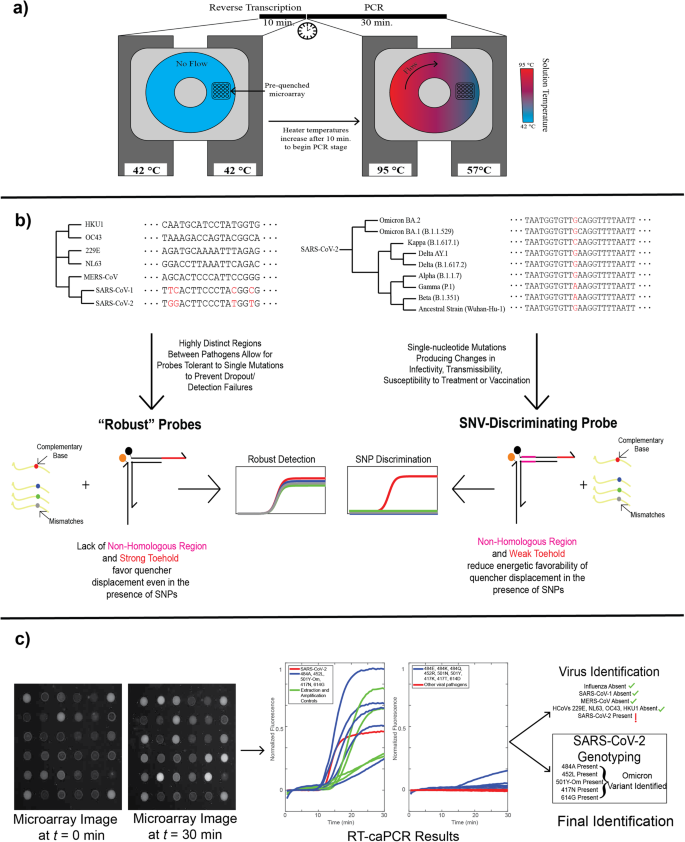
Integración de la transcripción inversa en caPCR
Primero introdujimos un paso de RT en el ensayo de caPCR estándar. La caPCR se realiza utilizando un conjunto de 4 calentadores que se sujetan a cada cara de un cartucho que contiene una cámara toroidal llena con la mezcla de reacción de PCR.4. Al configurar los calentadores en un lado de la cámara a la temperatura de hibridación de la PCR y el otro a la temperatura de desnaturalización, se establece un gradiente, produciendo así un flujo convectivo pasivo que impulsa la amplificación de la PCR. La matriz de sondas preapagadas se inmoviliza en la superficie del cartucho y responde a través de TMSD a medida que se sintetizan productos de PCR específicos. Para acomodar un paso inicial de transcripción inversa, programamos los calentadores para mantener una temperatura constante de 42 °C para la síntesis de ADNc durante 10 minutos, después de lo cual los calentadores se ajustan a 57 °C y 95 °C respectivamente para iniciar el flujo convectivo (Fig. 2a). Se utiliza un enfoque de RT-PCR en un solo recipiente para reducir el número de pasos de manipulación manual en el flujo de trabajo. La detección mediada por sonda en la plataforma caPCR funciona de manera óptima en condiciones de PCR asimétricas: un exceso del cebador que produce la hebra que desplaza al extintor genera una señal más fuerte en la matriz de sondas fluorescentes que una relación de cebador directo a inverso de 1:1. (Higo. S1). Nos aseguramos de que este mismo cebador también mediara la síntesis de ADNc a partir de objetivos de ARN para evitar la necesidad de introducir cebadores separados para las etapas de transcripción inversa y PCR.
a El flujo de trabajo de caPCR se puede ampliar para incluir un paso de transcripción inversa (RT-caPCR) mediante la adición de una enzima transcriptasa inversa y una etapa de RT dedicada con todos los calentadores del dispositivo configurados a la temperatura de RT (42 °C). Luego, las temperaturas del calentador se aumentan a diferentes temperaturas para la desnaturalización (95 °C) y el recocido (57 °C), induciendo el flujo convectivo y la amplificación por PCR de las plantillas de ADNc. b El esquema de diseño energético de sondas, basado en la termodinámica de unión al ADN, se puede utilizar para abordar dos necesidades en el diagnóstico de enfermedades infecciosas: I) La detección de patógenos virales debe ser robusta ante pequeños cambios en la secuencia de nucleótidos para evitar la abandono si surgen nuevas variantes. II) La discriminación de variantes requiere una resolución de un solo nucleótido. Se puede utilizar la variación de las energías de punto de apoyo, dominio y región no homóloga (NHR) para cumplir ambos requisitos. c Ejemplo de salida de datos de una ejecución de RT-caPCR de donut. Las imágenes de la matriz de sondas se adquieren cada 30 segundos durante 30 minutos de caPCR. Se extraen y analizan rastros de fluorescencia para identificar qué patógenos respiratorios están presentes y, si corresponde, qué variante del SARS-CoV-2 está presente.
Para desarrollar sondas sintonizables con selectividad de secuencia programable, que van desde sensibles a mutaciones hasta tolerantes a mutaciones, utilizamos 2 conjuntos de criterios energéticos para las sondas de punta (Fig. 2b). Las sondas robustas cuentan con una región de sujeción más fuerte y un dominio correspondientemente más débil para reducir la influencia general de una única mutación en el inicio de la reacción de unión y el posterior desplazamiento del brazo extintor. Para la discriminación SNV, se reduce la longitud del punto de apoyo y se introduce una región no homóloga (NHR) que carece de complementariedad con el objetivo. Esta combinación sirve para reducir la favorabilidad energética general para la unión de los pies y el desplazamiento del brazo extintor, proporcionando así peso adicional a los desajustes individuales en la secuencia objetivo.
Aplicamos RT-caPCR con sondas robustas y que discriminan SNV para la detección de virus respiratorios. Las imágenes de la matriz de sondas del sistema RT-caPCR se procesan utilizando un script MATLAB personalizado para producir rastros de fluorescencia que luego se interpretan para identificar tanto los objetivos virales presentes como, si corresponde, la variante del SARS-CoV-2. En la figura 2 se muestra un ejemplo de la variante Omicron del SARS-CoV-XNUMX. 2c. Por lo tanto, nuestro ensayo RT-caPCR en un solo recipiente demuestra su capacidad para realizar simultáneamente una detección sensible con sondas robustas y una resolución de un solo nucleótido con sondas discriminatorias de SNV para identificar con precisión objetivos de ARN de interés en una sola reacción.
Ajuste de la energía entre sondas robustas y que discriminan SNV
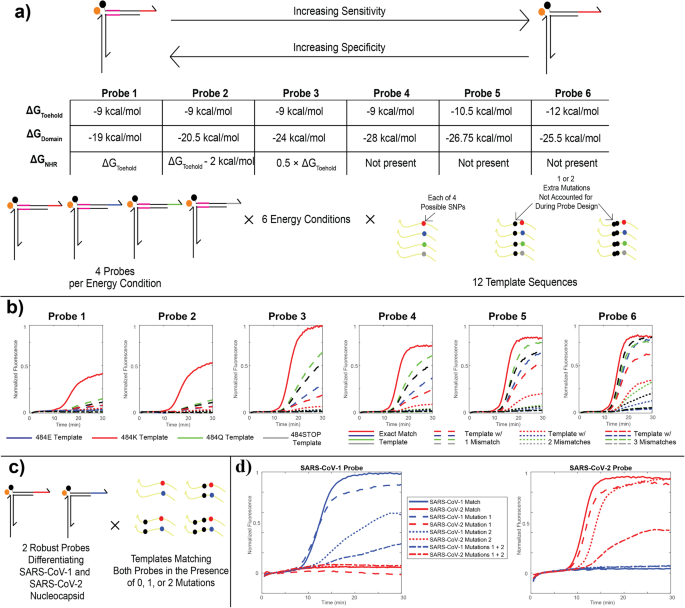
Para aumentar la diversidad de objetivos detectados en nuestro ensayo RT-caPCR, optimizamos la energía de la unión del pie y el desplazamiento de la cadena para modular el equilibrio entre sensibilidad y especificidad. Como estudio de caso, utilizamos el genotipo '2E' del SARS-CoV-484 como modelo para nuestra evaluación energética. Las mutaciones en este sitio están asociadas con una actividad neutralizante reducida de los anticuerpos del huésped.12, y 3 de 4 posibles bases de nucleótidos (adenina, guanina y citosina) se encuentran en la primera posición del codón en las variantes del SARS-CoV-2 que evolucionaron naturalmente9. La sustitución de uracilo en este sitio produciría un codón de parada y, por lo tanto, no se ha observado en la naturaleza, pero por razones de rigor incluimos una condición de timina en nuestra evaluación. Diseñamos una sonda para cada uno de los 4 posibles nucleótidos en este locus y establecimos 6 condiciones termodinámicas diferentes basadas en la energía de unión (ΔGo) de las 3 regiones de la sonda del pie (NHR, dominio y punto del pie; Fig. 3a, ver Materiales y Métodos). Estas condiciones energéticas van desde las más específicas (Sonda 1), en la que la energía del NHR y el punto de apoyo son aproximadamente iguales (alrededor de −9 kcal/mol), hasta las más sensibles (Sonda 6), en las que no hay NHR y el punto de apoyo es ligeramente más fuerte (−12 kcal/mol). En todos los casos, la energía de unión al dominio se ajusta para mantener constante la energía de unión de la sonda general en aproximadamente −40 kcal/mol.
a Parámetros de diseño para la caracterización energética de sondas. Se diseñaron seis esquemas energéticos diferentes variando las energías de unión de las regiones de punto de apoyo, dominio y NHR, para permitir un equilibrio entre la especificidad de un solo nucleótido y la sensibilidad en forma de robustez a las mutaciones. Se diseñaron cuatro sondas diferentes para cada uno de los seis diseños energéticos, cubriendo los cuatro codones posibles en el sitio de mutación 484 en la proteína de pico del SARS-CoV-2, tres de los cuales se han observado en variantes. Cada sonda se probó con 12 plantillas de plásmidos diferentes, incluidas las 4 mutaciones únicas de un solo nucleótido solas y en presencia de una o dos mutaciones más encontradas aguas arriba en la variante Omicron. b Resultados de caPCR de la serie de sondas 484 K. Las curvas sólidas de color rojo indican el desempeño en el objetivo sin mutaciones. Las líneas discontinuas, punteadas y de puntos indican plantillas con 1, 2 o 3 discrepancias en relación con la secuencia de la sonda, respectivamente. El color especifica el codón en el sitio 484 (E, K, Q o STOP). c Esquema para probar una discriminación sólida del SARS-CoV-1/2. Se diseñaron dos sondas según el esquema energético. “Sonda 5” en parte a para una región del gen de la nucleocápside en el SARS-CoV-1 y el SARS-CoV-2. Ambas sondas se probaron en presencia de la plantilla de coincidencia exacta y variantes que incluyen uno o ambos de dos desajustes aguas arriba, así como en presencia de la plantilla de coincidencia exacta y variantes no coincidentes de la otra sonda. d Resultados de las pruebas descritas en (c). Las líneas continuas, discontinuas, punteadas y de puntos indican plantillas de coincidencia exacta, plantillas con cualquiera de dos SNV o plantillas con ambos SNV en relación con el genoma del SARS-CoV-1 (azul) o del SARS-CoV-2 (rojo).
Probamos estas sondas con 12 plantillas de ADN diferentes, 4 de las cuales representan la plantilla de coincidencia exacta para cada una de las 4 bases posibles en el codón 484. Para demostrar la capacidad de ajuste de la energía de nuestra sonda, tomamos mutaciones que surgieron en linajes posteriores de la evolución del SARS-CoV-2 y las introdujimos en las plantillas: la mutación T478K encontrada en los linajes Delta AY y Omicron, y la mutación S477N encontrada en Omicron. linajes (Fig. 3a)10. Las 24 sondas se evaluaron en respuesta a la amplificación de cada una de las 12 plantillas. Cifra 3b muestra un subconjunto de estos resultados, específicamente para las sondas diseñadas para apuntar a la mutación 484 K que se encuentra en los COV Beta y Gamma.10. A medida que se reduce la fuerza del NHR y aumenta el punto de apoyo, la detección de la plantilla de coincidencia exacta (línea roja sólida) sigue siendo fuerte y la sonda se vuelve más sensible a las plantillas con 1 (líneas discontinuas) y 2 (líneas de puntos) no coincidentes. Esto demuestra nuestra capacidad para diseñar energías de sonda para obtener resultados de unión a objetivos muy específicos. Con base en estos resultados, seleccionamos el esquema energético 2 para sondas que discriminan SNV y el esquema 5 para sondas robustas.
Diseñamos dos sondas robustas dirigidas a la secuencia del gen de la nucleocápside del SARS-CoV-2 y el SARS-CoV-1. Para garantizar que estas secuencias de patógenos pudieran diferenciarse utilizando sondas robustas y al mismo tiempo seguir siendo tolerantes a las mutaciones, diseñamos 2 plantillas que no contenían desajustes, 8 de 1 desajustes o ambos desajustes juntos (Fig. 3c, ver Materiales y Métodos). La región seleccionada para la detección de SARS-CoV-1 y SARS-CoV-2 difiere entre las 2 secuencias objetivo en 8 mutaciones: como tal, se seleccionaron discrepancias en la plantilla en función de estas diferencias. Como tal, la plantilla del SARS-CoV-2 con 2 desajustes solo tiene 6 diferencias en relación con la secuencia del SARS-CoV-1, lo que resalta una aplicación en la vida real de la importancia de una capacidad de ajuste precisa de las sondas para la sensibilidad frente a la especificidad. Cifra 3d muestra los resultados de estas pruebas. En todos los casos, no observamos reactividad cruzada entre las sondas y plantillas del SARS-CoV-1 y el SARS-CoV-2: el ARN del SARS-CoV-1 solo fue detectado por la sonda del SARS-CoV-1, y el ARN del SARS-CoV-2 solo fue detectado por la sonda del SARS-CoV-2. El ARN sólo fue detectado por la sonda SARS-CoV-2. Las plantillas que contienen cualquiera de las 1 mutaciones también pueden detectarse sin ambigüedades mediante cada sonda apropiada, y una de las 2 mutaciones afecta más claramente el rendimiento que la otra. De manera similar, todavía se detectan plantillas con ambas mutaciones juntas, aunque el rendimiento se ve ligeramente comprometido debido a una menor preferencia energética en el desplazamiento del brazo extintor.
A través de nuestros estudios específicos sobre la energía de las sondas, demostramos nuestra capacidad para diseñar sondas con precisión para que tengan el equilibrio deseado entre sensibilidad y especificidad. Las sondas discriminatorias de SNV tienen energías que requieren una coincidencia exacta para el desplazamiento del brazo extintor, mientras que las sondas robustas son más promiscuas en su unión a objetivos con la capacidad de detectar un amplicón que contiene hasta dos mutaciones. El rango energético que exploramos se puede aplicar para ajustar las sondas según sea necesario desde el esquema de energía 1 al esquema de energía 6 para permitir la detección de objetivos con el número deseado de discrepancias de la secuencia de la sonda.
Rendimiento de sondas robustas y discriminatorias SNV
Para demostrar la prueba de concepto de nuestro diseño de sonda altamente sintonizable, creamos un panel completo de sondas robustas y discriminatorias SNV. Nuestro panel destaca la utilidad de sondas robustas mediante la detección de los 7 coronavirus que se sabe que infectan a los humanos (HCoV 229E, HKU1, NL63 y OC43; MERS-CoV, SARS-CoV-1 y SARS-CoV-2), así como la influenza. A y B8. Además, nuestro panel demuestra la funcionalidad de las sondas discriminatorias del SNV mediante el uso de 14 sondas y 3 amplicones que cubren 5 sitios comúnmente mutados en el gen de pico del SARS-CoV-2 para permitir la diferenciación entre las variantes preocupantes (417, 452, 484, 501 y 614; figura. S2)9,10,11.
Para evaluar el rendimiento de las sondas robustas en nuestro ensayo RT-caPCR para la detección de objetivos sensibles, realizamos 65 pruebas RT-caPCR utilizando muestras de medios de transporte viral (VTM) negativos para coronavirus que cubren una variedad de datos demográficos de pacientes (Tablas S1–S4) enriquecido con ARN sintético de cada uno de los 7 coronavirus humanos, ya sea adquirido en Twist Bioscience o sintetizado internamente mediante transcripción in vitro (IVT) a partir de fragmentos de genes (Tabla S5). El ARN se aisló y purificó a partir de estas muestras clínicas artificiales utilizando un método de extracción basado en perlas.13 debido al precedente del uso de dicha metodología para la extracción de ARN en los flujos de trabajo de diagnóstico del SARS-CoV-214 y fue utilizado como entrada para nuestro sistema. Este ensayo también incluyó un control de extracción (ARN del bacteriófago MS2 añadido a la muestra artificial antes de la purificación) y un control de amplificación (ARN del control positivo interno (IPC) añadido a la mezcla de reacción RT-caPCR). A menos que se especifique lo contrario, los experimentos aquí recibieron aproximadamente 106 copias de ARN MS2 y 107 copias de ARN viral. La reacción RT-caPCR se realizó en 40 minutos utilizando 5,000 moléculas de ARN de control de IPC, la muestra de ARN extraída y 11 pares de cebadores que cubrían todos los objetivos y controles.
Los datos de RT-caPCR se analizaron para identificar los objetivos virales presentes en cada muestra y, en el caso del SARS-CoV-2, qué variante específica estaba presente (Fig. 4). Figura 4a muestra una ejecución representativa con la variante Omicron del SARS-CoV-2 como objetivo de ARN. Todas las sondas robustas para la discriminación viral se muestran en rojo, mientras que las sondas que discriminan SNV se muestran en azul. Los controles de extracción y amplificación se muestran en verde. Los puntos de sonda positivos y negativos corresponden a sondas completamente desactivadas o completamente desactivadas que utilizan secuencias para las que no se incluyeron cebadores ni plantilla y se muestran en gris. Este tipo de datos se puede resumir en un gráfico comprimido como se muestra en la Fig. 4b: todas las curvas positivas esperadas se representan en el gráfico de la izquierda y todas las curvas negativas esperadas se muestran en el gráfico de la derecha. Si bien hay un pequeño nivel de actividad fuera del objetivo en algunas sondas específicas de SNV, al comparar las curvas de las sondas específicas de SNV en el objetivo (484 A, 452 L, 501Y-Om, 417 N y 614 G) con otras sondas SNV- Las sondas específicas confirman que las sondas en el objetivo responden con mayor fuerza en todos los casos, según lo evaluado visualmente y a través de nuestro algoritmo de discriminación SNV basado en derivados de fluorescencia (consulte Materiales y métodos y Métodos complementarios). Observamos que, debido a la alta carga mutacional de la variante Omicron en relación con otras variantes11, diseñamos la secuencia de la sonda 484 A para incluir también las mutaciones S477N y T478K encontradas en esta variante para mejorar la detección.10. De manera similar, una sonda 501Y-Om única incluye mutaciones Q493R, G496S y Q498R que se encuentran específicamente en la variante Omicron. Posteriormente se introdujo una sonda específica para la subvariante BA.2 del linaje Omicron (501Y-Om2). Esta sonda es idéntica a la secuencia 501Y-Om pero carece de la mutación G496S.10.
a Resultados de ejemplo de una ejecución de RT-caPCR que detecta la variante preocupante Omicron del SARS-CoV-2. Las sondas robustas de detección viral están en rojo, las sondas que discriminan SNV están en azul, los controles de extracción y amplificación están en verde y los controles positivos y negativos están en gris. Cada curva representa la intensidad del punto de la matriz de sondas correspondiente en cada punto temporal de las 60 imágenes adquiridas, normalizadas al punto más brillante de la matriz. b Resultados resumidos correspondientes al rodaje a. Todos los positivos esperados (controles de extracción y amplificación, identificación viral en el objetivo y todas las sondas de discriminación de SNV en el objetivo) se muestran en el gráfico de la izquierda, mientras que todas las sondas negativas esperadas (todas las demás sondas de discriminación de SNV y virales, como así como los controles negativos) se muestran en el gráfico de la derecha. c Resultados de detección viral para el coronavirus humano 229E, el coronavirus del síndrome respiratorio de Oriente Medio (MERS-CoV) y el SARS-CoV-1 en el formato descrito en (b). d. La discriminación SNV resulta de una plantilla de variante Delta del SARS-CoV-2 en el formato especificado anteriormente.
Figura 4c muestra resultados representativos comprimidos de 3 ejecuciones para 3 plantillas de patógenos virales diferentes: coronavirus humano 229E, MERS-CoV y SARS-CoV-1. Las 3 muestras muestran una detección clara del objetivo de ARN viral con sondas robustas de identificación de patógenos, sin actividad de sonda de identificación viral fuera del objetivo y la presencia de todos los controles.
Mesa 1 Resume los resultados de identificación viral de todos los ensayos válidos. La competencia por los recursos de la reacción de PCR (p. ej., polimerasa, dNTP) puede suprimir la amplificación de objetivos con un número de copias más bajo, por lo que una ejecución del ensayo se consideró válida si se detectó un patógeno viral y se detectó al menos 1 de los 2 controles (MS2 e IPC). , o no se detectó ningún patógeno viral pero se detectaron ambos controles. 11 de las muestras eran controles negativos, que incluían solo ARN de control de MS2 e IPC. En las 11 muestras, se detectaron tanto IPC como MS2 (100%). Se identificaron 36 de 36 muestras de SARS-CoV-2 (100 %) según lo evaluado mediante la detección de la sonda de nucleocápside del SARS-CoV-2. Los coronavirus humanos restantes (229E, NL63, OC43 y HKU1), así como los objetivos SARS-CoV-1 y MERS-CoV, también muestran una alta sensibilidad, con una detección del 100 % para todos los objetivos. Tanto el proceso de unión del cebador como el desplazamiento del extintor de las sondas mediado por amplicones contribuyen a la especificidad del panel, que fue del 100% en todos los objetivos. Aquí, la especificidad se calcula como la proporción de verdaderos negativos detectados correctamente. En resumen, ninguna plantilla indujo la detección en una sonda robusta distinta de aquella para la que fue diseñada específicamente.
Figura 4d muestra una ejecución representativa que destaca la capacidad de nuestro ensayo RT-caPCR para identificar el objetivo de ARN y proporcionar una resolución de un solo nucleótido al mostrar la detección de variantes virales simultáneamente con la identificación de patógenos para la variante Delta del SARS-CoV-2. Si bien la sonda 452 L muestra una actividad significativa fuera del objetivo, su detección es claramente menos eficiente que la sonda 452 R en el objetivo (la curva azul con mayor intensidad en el gráfico de la izquierda). Por lo tanto, las sondas que discriminan SNV permiten una distinción clara entre variantes del SARS-CoV-2 que difieren solo en unas pocas mutaciones.
Mesa 2 resume el rendimiento de las sondas específicas de SNV para las variantes del SARS-CoV-2 en 36 ensayos. El número de detecciones esperadas es igual al número de ejecuciones para las que se incluyó una plantilla que muestra esa mutación, mientras que el número de negativos esperados es el número de ejecuciones para las que no se incluyó la plantilla correspondiente. Los verdaderos positivos son series en las que la sonda de interés era la sonda específica de SNV dominante para ese sitio de mutación (según se evaluó algorítmicamente), y los verdaderos negativos son aquellas series en las que no lo era. En una ejecución, observamos que la sonda 614 G quedó oscurecida por una partícula extraña (polvo), por lo que el total de estos objetivos es 35, en lugar de 36. Aparte de la 484E, todas las sondas muestran al menos un 90 % de sensibilidad. El bajo rendimiento de la sonda 484E puede explicarse en parte por la presencia de un SNV (mutación T478K) en el amplicón de las variantes Delta. Todas las demás sondas muestran una especificidad del 100%, ya que un amplicón siempre desplazará preferentemente a su extintor correspondiente sobre uno con una mutación, suponiendo que las energías del objetivo sean iguales.
En conjunto, estos datos resaltan la capacidad de las sondas robustas y las sondas discriminatorias de SNV para trabajar en conjunto para identificar rápidamente el objetivo de ARN presente (con la sonda robusta) y proporcionar una resolución de un solo nucleótido (con la sonda discriminatoria de SNV).
Evaluación del límite de detección de RT-caPCR
A continuación, realizamos una serie de experimentos para determinar el límite de detección de nuestro ensayo RT-caPCR. En todas las pruebas anteriores en la Fig. 4, un aporte viral de 107 Las moléculas se introdujeron en el VTM antes de la extracción, similar a las estimaciones de carga viral máxima para los pacientes de Omicron y Delta.15,16. Aquí cuantificamos el LOD de nuestro ensayo variando el número de copias de entrada de ARN viral y manteniendo las mismas copias de control. Utilizando ARN purificado obtenido de Twist Bioscience o sintetizado internamente a partir de fragmentos de genes mediante IVT, logramos un LOD de 1,000 moléculas para los 7 coronavirus. Los resultados representativos se muestran en la Fig. S3. En ambas Fig. S3a y S3b, encontramos que podemos detectar exitosamente hasta 1,000 moléculas por reacción para un solo objetivo con ambos controles (HCoV 229E; Fig. S3a) y para una variante del SARS-CoV-2 con ambos controles (Omicron; Fig. S3b), una cantidad 3 órdenes de magnitud inferior a la utilizada para las pruebas VTM cuando se utiliza ARN purificado. Cuando sólo se varía la presencia de un único objetivo, como en la Fig. S3a, encontramos un retraso claro en el tiempo umbral para cada orden de magnitud solo para este objetivo, mientras que los 2 controles son consistentes en todas las ejecuciones. Esta tendencia es menos clara para el SARS-CoV-2, lo que era de esperar dado que este objetivo requiere la amplificación simultánea de 4 amplicones y 2 controles. La competencia por los recursos enzimáticos y los dNTP parece alterar la clara tendencia observada para los objetivos de un solo amplicón. En la Fig. S3c, se realizó una serie de diluciones variando la cantidad de ARN de entrada a la tubería de extracción para la variante Omicron. Aquí también vemos una detección hasta el valor más bajo probado (entrada de 10,000 moléculas en el paso de extracción de ARN con aproximadamente 1,000 moléculas en el paso de RT debido a pérdidas durante la extracción y sin utilizar el volumen completo de elución de 25 µL en la reacción). y un retraso esperado en la amplificación en función de la concentración de entrada.
Detección de múltiples objetivos en una sola muestra.
Habiendo demostrado el éxito de nuestro ensayo RT-caPCR con sonda sintonizable en la identificación de objetivos de ARN con sondas robustas (cada virus) con especificidad de un solo nucleótido con sondas que discriminan SNV (variantes de SARS-CoV-2), a continuación exploramos cómo se desempeñó nuestro ensayo cuando evaluar entradas más complejas mediante la simulación de muestras de coinfección que contienen múltiples objetivos de ARN. Aquí, una cantidad de entrada de 105 Las moléculas de cada plantilla se agregaron directamente a la reacción RT-caPCR sin pasos de extracción VTM. También ampliamos nuestro ensayo RT-caPCR para detectar más objetivos de ARN al incluir 3 conjuntos de cebadores adicionales y 4 sondas para detectar influenza A y B (2 dirigidas al gen de la proteína de matriz M1 de la influenza A17 y 2 dirigidos a la proteína no estructural 1 (nsp1) en los linajes Yamagata y Victoria de influenza B18). Este ensayo de 14 complejos se probó para los 4 nuevos objetivos para confirmar su eficacia (Fig. S4).
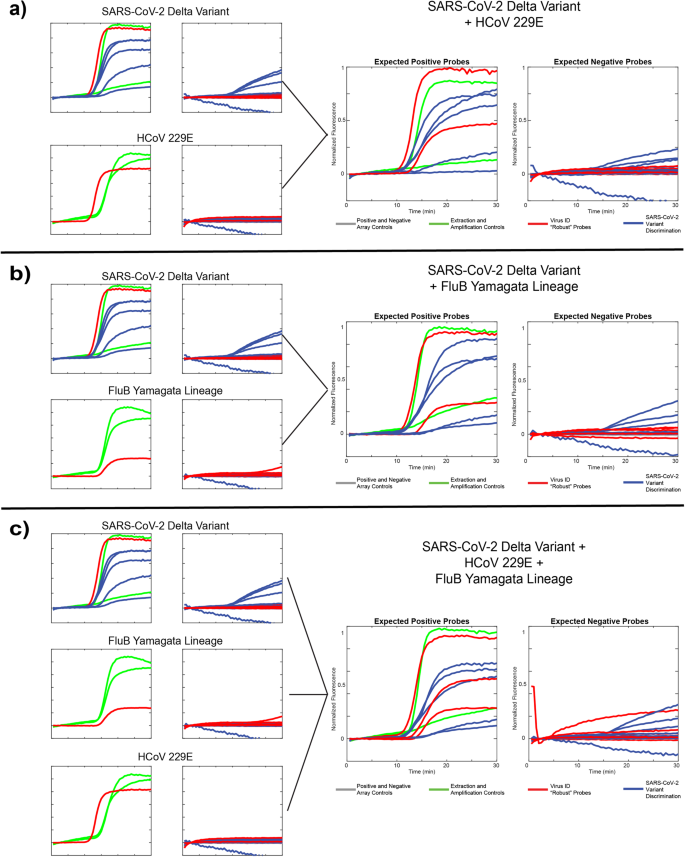
Evaluamos cada una de 3 plantillas virales diferentes solas y en 3 combinaciones diferentes: variante Delta del SARS-CoV-2 + HCoV229E (Fig. 5a), variante Delta del SARS-CoV-2 + linaje FluB Yamagata (Fig. 5b), y el linaje SARS-CoV-2 + HCoV229E + FluB Yamagata (Fig. 5c). Los datos se presentan como imágenes de trazas fluorescentes comprimidas como se describe anteriormente con las sondas positivas esperadas en el panel izquierdo y las sondas negativas esperadas en el panel derecho. En todas las ejecuciones, observamos el comportamiento esperado: las sondas robustas (identificación de virus) informan la presencia de todas las plantillas de patógenos incluidas, y las sondas que discriminan SNV (identificación de variantes) identifican correctamente la variante Delta con niveles bajos de actividad de sonda fuera del objetivo en todas las pruebas. 5 sitios SNV. La sonda 484E tiene el desempeño más débil, probablemente debido a la falta de coincidencia antes mencionada en la región del dominio de esta sonda. Además, en algunos experimentos, el control de extracción de MS2 tuvo un desempeño deficiente debido a la competencia por los recursos entre 8 amplicones diferentes. También observamos que estas ejecuciones solo incluyen 1 réplica de cada control MS2 e IPC, en lugar de 2 como en la Fig. 4, ya que se trataba de un panel actualizado que incluía la sonda 501Y-Om2. Estos datos demuestran la capacidad de nuestra tecnología para evaluar con precisión muestras más complejas que contienen múltiples objetivos de ARN de interés, lo que demuestra la versatilidad del ensayo.
a Resultados de una sola plantilla de la variante Delta del SARS-CoV-2 y del coronavirus humano 229E junto con una muestra de “coinfección” que incluye ambas plantillas. b Resultados de una sola plantilla de la variante Delta del SARS-CoV-2 y el linaje Yamagata de influenza B, junto con una muestra de coinfección que incluye ambas plantillas. c. Resultados de un solo objetivo de los mismos objetivos que en a y b, junto con una muestra de coinfección que incluye las tres plantillas.
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- PlatoData.Network Vertical Generativo Ai. Empodérate. Accede Aquí.
- PlatoAiStream. Inteligencia Web3. Conocimiento amplificado. Accede Aquí.
- PlatoESG. Carbón, tecnología limpia, Energía, Ambiente, Solar, Gestión de residuos. Accede Aquí.
- PlatoSalud. Inteligencia en Biotecnología y Ensayos Clínicos. Accede Aquí.
- Fuente: https://www.nature.com/articles/s42003-023-05346-4